下载最新版GO,并且解析好
首先要明白,需要下载什么?
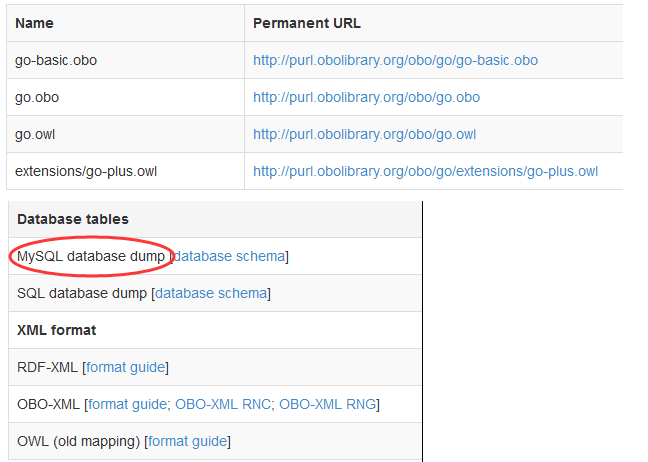
要下载四万多条GO记录的详细信息(http://purl.obolibrary.org/obo/go/go-basic.obo)
要下载GO与GO之间的关系(http://archive.geneontology.org/latest-termdb/go_daily-termdb-tables.tar.gz)
要下载GO与基因之间的对应关系!(物种)(ftp://ftp.ncbi.nlm.nih.gov/gene/DATA)
去官网!
http://geneontology.org/page/download-ontology
grep '\[Term\]' go-basic.obo |wc
43992 43992 307944
版本的区别!刚才我们下载的GO共有43992条,而以前的版本才38804条
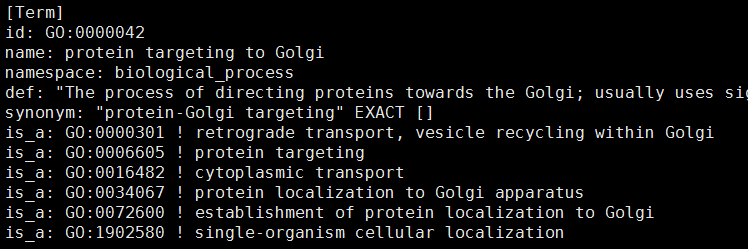
GO与GO之间的关系
对应关系也在更新
> as.list(GOBPPARENTS['GO:0000042'])
$`GO:0000042`
is_a is_a is_a is_a
"GO:0000301" "GO:0006605" "GO:0016482" "GO:0072600"
library(org.Hs.eg.db)
library(GO.db)
> tmp=toTable(org.Hs.egGO) ##这个只包括基因与最直接的go的对应关系
> dim(tmp)
[1] 213101 4
> tmp2=toTable(org.Hs.egGO2ALLEGS) #这个是所有的基因与go的对应关系
> dim(tmp2)
[1] 2218968 4
基因与GO的对应关系也在更新
grep '^9606' gene2go |wc -l ### ##这个只包括基因与最直接的go的对应关系
269063
ftp://ftp.informatics.jax.org/pub/reports/index.html#go
ftp://ftp.ncbi.nlm.nih.gov/gene/DATA
ftp://ftp.informatics.jax.org/pub/reports/index.html#go
关于limma包差异分析结果的logFC解释
首先,我们要明白,limma接受的输入参数就是一个表达矩阵,而且是log后的表达矩阵(以2为底)。
那么最后计算得到的logFC这一列的值,其实就是输入的表达矩阵中case一组的平均表达量减去control一组的平均表达量的值,那么就会有正负之分,代表了case相当于control组来说,该基因是上调还是下调。
我之前总是有疑问,明明是case一组的平均表达量和control一组的平均表达量差值呀,跟log foldchange没有什么关系呀。
后来,我终于想通了,因为我们输入的是log后的表达矩阵,那么case一组的平均表达量和control一组的平均表达量都是log了的,那么它们的差值其实就是log的foldchange
首先,我们要理解foldchange的意义,如果case是平均表达量是8,control是2,那么foldchange就是4,logFC就是2咯
那么在limma包里面,输入的时候case的平均表达量被log后是3,control是1,那么差值是2,就是说logFC就是2。
这不是巧合,只是一个很简单的数学公式log(x/y)=log(x)-log(y)
用excel表格做差异分析
其实主要要讲的不是用excel来做差异分析,只是想讲清楚差异分析的原理,用excel可视化的操作可能会更方便理解,而且想告诉大家,其实生物信息学分析,本来就很简单的,那么多软件,只有你理解了原理,你自己就能写出来的!
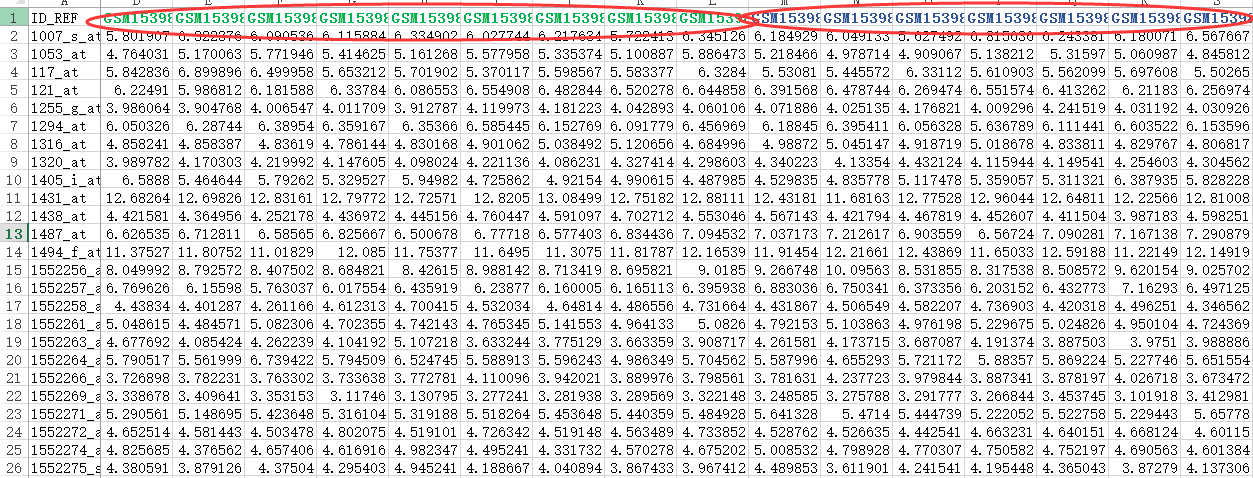
首先,还是得到表达矩阵,下面绿色的样本是NASH组,蓝色的样本是normal组
我们进行差异分析,很简单,就是看两组的表达值,是否差异,而检验的方法就是T检验。
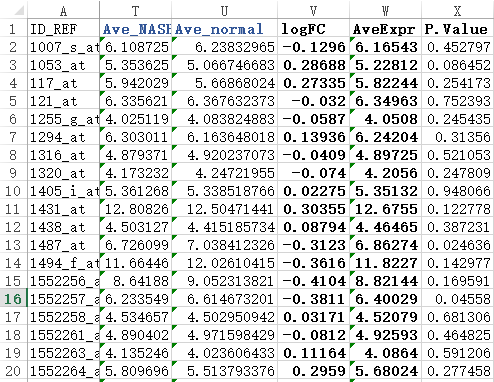
=AVERAGE(D2:L2) ##求NASH组的平均表达量
=AVERAGE(M2:S2) ###求normal的平均表达量
=T2-U2 ##计算得到logFOLDchange值
=AVERAGE(D2:S2) ###得到所有样本的平均表达量
=T.TEST(D2:L2,M2:T2,2,3) ###用T检验得到两个组的表达量的差异显著程度。
简单检查几个值就可以看到跟limma包得到的结果差不多。
用limma包对芯片数据做差异分析
下载该R语言包,然后看说明书,需要自己做好三个数据(表达矩阵,分组矩阵,差异比较矩阵),总共三个步骤(lmFit,eBayes,topTable)就可以啦
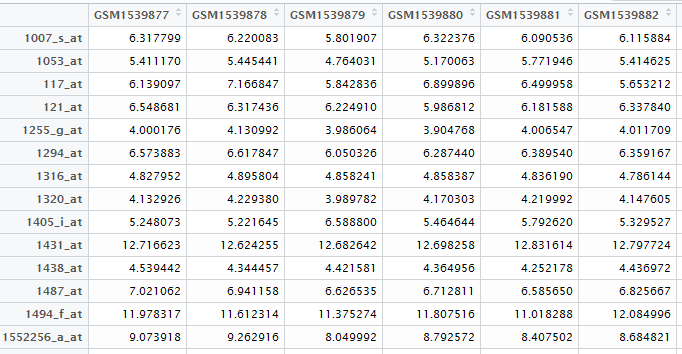
首先做第一个数据,基因表达矩阵!
自己在NCBI里面可以查到下载地址,然后用R语言读取即可
exprSet=read.table("GSE63067_series_matrix.txt.gz",comment.char = "!",stringsAsFactors=F,header=T)
rownames(exprSet)=exprSet[,1]
exprSet=exprSet[,-1]
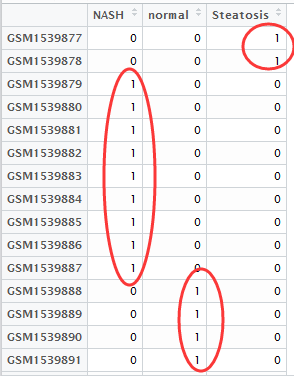
然后做好分组矩阵,如下
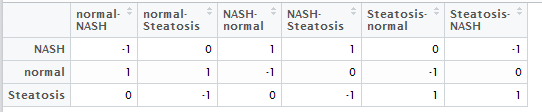
然后做好,差异比较矩阵,就是说明你想把那些组拿起来做差异分析,如下
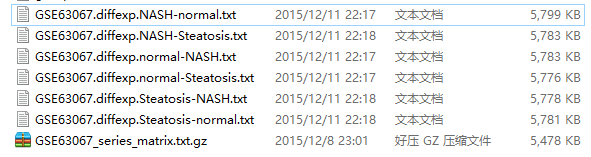
最后输出结果:
我进行了6次比较,所以会输出6次比较结果
最后打开差异结果,解读,说明书如下!
在我的github有完整代码
下载最新版的KEGG信息,并且解析好
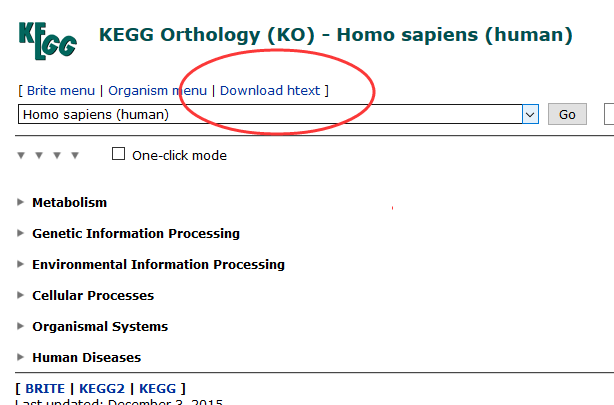
打开官网:http://www.genome.jp/kegg-bin/get_htext?hsa00001+3101
http://www.genome.jp/kegg-bin/get_htext#A1 (这个好像打不开)
可以在里面找到下载链接
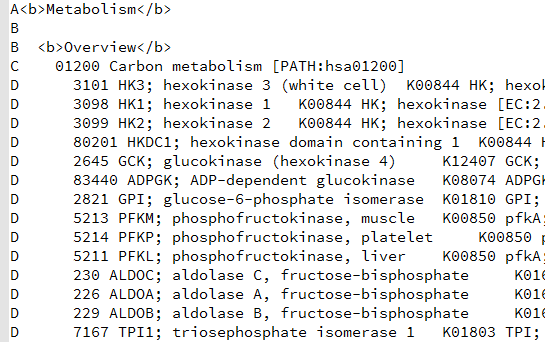
下载得到文本文件,可以看到里面的结构层次非常清楚,
C开头的就是kegg的pathway的ID所在行,D开头的就是属于它的kegg的所有的基因
A,B是kegg的分类,总共是6个大类,42个小类
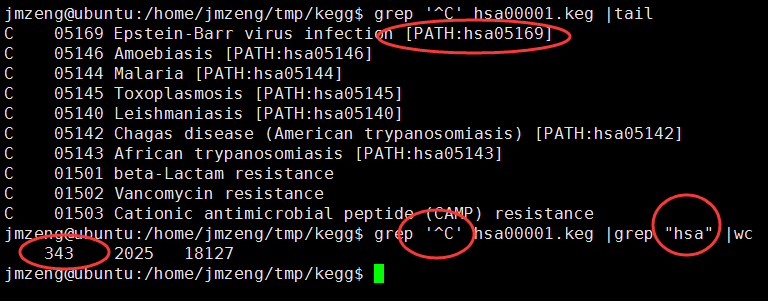
grep ^A hsa00001.keg
A<b>Metabolism</b>
A<b>Genetic Information Processing</b>
A<b>Environmental Information Processing</b>
A<b>Cellular Processes</b>
A<b>Organismal Systems</b>
A<b>Human Diseases</b>
也可以看到,到目前为止(2015年12月8日20:26:57),共有343个kegg的pathway信息啦
接下来我们就把这个信息解析一下:
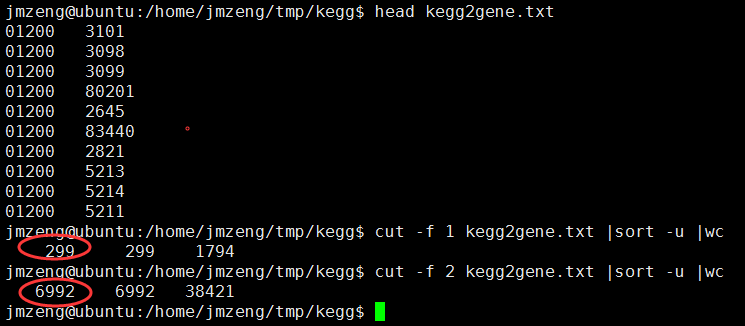
perl -alne '{if(/^C/){/PATH:hsa(\d+)/;$kegg=$1}else{print "$kegg\t$F[1]" if /^D/ and $kegg;}}' hsa00001.keg >kegg2gene.txt
这样就得到了
但是我发现了一个问题,有些通路竟然是没有基因的,我不是很明白为什么?
C 04030 G protein-coupled receptors [BR:hsa04030]
C 01020 Enzyme-linked receptors [BR:hsa01020]
C 04050 Cytokine receptors [BR:hsa04050]
C 03310 Nuclear receptors [BR:hsa03310]
C 04040 Ion channels [BR:hsa04040]
C 04031 GTP-binding proteins [BR:hsa04031]
那我们来看看kegg数据库更新的情况吧。
首先我们看org.Hs.eg.db这个R包里面自带的数据
Date for KEGG data: 2011-Mar15
org.Hs.egPATH has 5869 entrez genes and 229 pathways
2015年八月我用的时候是 6901 entrez genes and 295 pathways
现在是299个通路,6992个基因
所以这个更新其实很缓慢的,所以大家还在用DAVID这种网络工具做kegg的富集分析结果也差不大!
我的生物信息学视频上线啦
虽然优酷比较坑,但是他的受众多,大家可以去http://i.youku.com/trainee 上面找到我的全部视频,免费观看!
视频列表是http://www.bio-info-trainee.com/tmp/tutorial/video_list.html ,能顺利点击的链接代表视频录制完毕。
这次视频课程的大纲是http://www.bio-info-trainee.com/tmp/tutorial/syllabus.htm,但是很有可能会修改!
声明
大家好,欢迎观看由生信菜鸟团举办的生物信息学公益视频课程,我是主讲人Jimmy。
本课程仅面向那些生物出身却想转生物信息学数据分析的同学,其它人均不在考虑范围。
本课程在中国大陆录制,会尽量遵循中国大陆法律,如有法律问题,请联系我的律师,谢谢!
我会尽量保证视频中知识点的准确无误,如有任何讲解不当之处,欢迎批评指正!
发邮件联系我(jmzeng1314@outlook.com)即可,没什么必要不要QQ或者微信找我。
还有,本人不差钱,所以视频均免费的,录制完毕后我会放出百度云共享,现在还在测试阶段。
请不要无缘无故的批评我,我不会服务任何人,所以不可能有人是我的顾客,我不需要对你好,爱看不看!
hapmap计划资源收集
官网是:http://hapmap.ncbi.nlm.nih.gov/index.html.en
Jul 07 2009 00:00 Directory affy100k Mar 05 2010 00:00 Directory affy500k Jun 02 2010 00:00 Directory hapmap3_affy6.0
当然,数据也在更新
Jul 07 2009 00:00 Directory 2005-03_phaseI Dec 03 2009 00:00 Directory 2005-11_phaseII Jul 07 2009 00:00 Directory 2007-03 Jul 07 2009 00:00 Directory 2008-03 Jul 07 2009 00:00 Directory 2008-07_phaseIII Jul 07 2009 00:00 Directory 2008-10_phaseII Jul 07 2009 00:00 Directory 2009-01_phaseIII Jul 07 2009 00:00 Directory 2009-02_phaseII+III Aug 18 2010 00:00 Directory 2010-05_phaseIII Sep 19 2010 00:00 Directory 2010-08_phaseII+III
数据都被整合好了:
- Bulk data
- Genotypes: Individual genotype data submitted to the DCC to date. Phase 3 data is available in PLINK format and HapMap format.
- Frequencies: Allele & genotype frequencies compiled from genotyping data submitted to the DCC to date. These have also been submitted to dbSNP and should be available in the next dbSNP build.
- LD Data: Linkage disequilibrium properties D', LOD , R2 compiled from the genotype data to date
- Phasing Data: Phasing data generated using the PHASE software, compiled from the genotype data to date.
- Allocated SNPs: dbSNP reference SNP clusters that have been picked and prioritized for genotyping according to several criteria (see info on how SNPs were selected). The file 00README contains per-chromosome SNP counts and further details.
- CNV Genotypes: CNV data from HapMap3 samples.
- Recombination rates and Hotspots: Recombination rates and hotspots compiled from the genotyping data.
- SNP assays: Details about assays submitted to the DCC to date. PCR primers, extension probes etc., specific to each genotyping platform.
- Perlegen amplicons: Details for mapping Perlegen amplicons to HapMap assayLSID. For primer sequences, see Perlegen's Long Range PCR Amplicon data.
- Raw data: Raw signal intensity data from HapMap genotypes. Currently includes data from Affymetrix GeneChip 100k and 500k Mapping Arrays.
- Inferred genotypes: Genotypes inferred using the method of Burdick et al. Nat Genet 38:1002-4.
- Mitochondrial and chrY haplogroups: Classification of phase I HapMap samples into mtDNA and chrY haplogroups. The distribution shown in Table 4 of the HapMap phase I paper (Nat Genet 38:1002-4) corresponds to unrelated parents in each one of the populations analyzed.
同时也发了很多篇文章:
- The International HapMap Consortium. Integrating common and rare genetic variation in diverse human populations.
Nature 467, 52-58. 2010. [Abstract] [PDF] [Supplementary information] - The International HapMap Consortium. A second generation human haplotype map of over 3.1 million SNPs.
Nature 449, 851-861. 2007. [Abstract] [PDF] [Supplementary information] - The International HapMap Consortium. A Haplotype Map of the Human Genome.
Nature 437, 1299-1320. 2005. [Abstract] [PDF] [Supplementary information] - The International HapMap Consortium. The International HapMap Project.
Nature 426, 789-796. 2003. [Abstract] [PDF] [Supplementary information] - The International HapMap Consortium. Integrating Ethics and Science in the International HapMap Project.
Nature Reviews Genetics 5, 467 -475. 2004. [Abstract] [PDF] - Thorisson, G.A., Smith, A.V., Krishnan, L., and Stein, L.D. The International HapMap Project Web site.
Genome Research,15:1591-1593. 2005. [Abstract] [PDF]
点突变详解
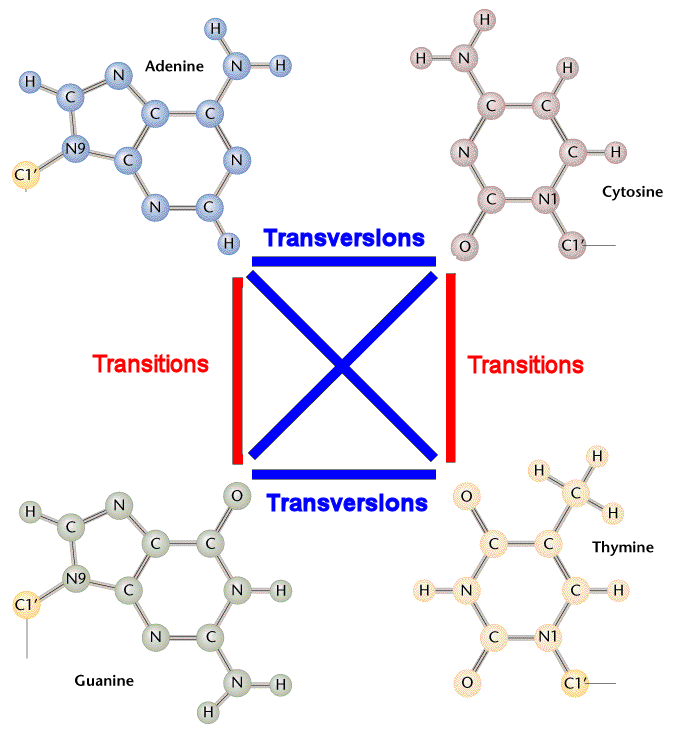
DNA分子中某一个碱基为另一种碱基置换,导致DNA碱基序列异常,是基因突变的一种类型。可分为转换和颠换两类。转换(transitions)是同类碱基的置换(AT→GC及GC→AT),颠换(transversions) 是不同类碱基的置换(AT→TA或CG,GC→CG或TA。
DNA substitution mutations are of two types. Transitions are interchanges of two-ring purines (A G) or of one-ring pyrimidines (C T): they therefore involve bases of similar shape. Transversions are interchanges of purine for pyrimidine bases, which therefore involve exchange of one-ring and two-ring structures.
我们在分析driver mutation的时候会区分各种点突变:
- 1. CpG transitions
- 2. CpG transversions
- 3. C:G transitions
- 4. C:G transversions
- 5. A:T transitions
- 6. A:T transversions
那么,我们有64种密码子,每种密码子都会有9种突变可能,我们如何得到一个所有的突变可能的分类并且打分表格呢?
类似于下面这样的表格:共576行!!!
head category.acgt
AAA>AAT 2 A T 6
AAA>AAC 2 A C 6
AAA>AAG 2 A G 5
AAA>ATA 1 A T 6
AAA>ACA 1 A C 6
AAA>AGA 1 A G 5
AAA>TAA 0 A T 6
AAA>CAA 0 A C 6
AAA>GAA 0 A G 5
AAT>AAA 2 T A 6
tail category.acgt
GGC>GGG 2 C G 2
GGG>AGG 0 G A 3
GGG>TGG 0 G T 4
GGG>CGG 0 G C 4
GGG>GAG 1 G A 3
GGG>GTG 1 G T 4
GGG>GCG 1 G C 4
GGG>GGA 2 G A 3
GGG>GGT 2 G T 4
GGG>GGC 2 G C 4
我本来以为这是一件很简单的事情,写起来,才发现好麻烦
里面用到的一个函数如下:就是判断突变属于上述六种的哪一种!
参考:https://www.mun.ca/biology/scarr/Transitions_vs_Transversions.html
https://en.wikipedia.org/wiki/Transversion
http://www.uvm.edu/~cgep/Education/Mutations.html
点突变,也称作单碱基替换(single base substitution),指由单个碱基改变发生的突变。
可以分为转换(transitions)和颠换(transversions)两类。
转换:嘌呤和嘌呤之间的替换,或嘧啶和嘧啶之间的替换。
颠换:嘌呤和嘧啶之间的替换。
方便理解下面再附上一张示意图,如下:
使用mutsig软件来找驱动基因
从数以万计的突变里面找到driver mutation这个课题很大,里面的软件我接触的就有十几个了,但是我尝试了其中几个,总是无法运行成功,不知道为什么,终于今天成功了一个,就是mutsig软件! 其实关于突变数据找driver mutation ,台湾一个大学做了一个数据库DriverDB http://ngs.ym.edu.tw/driverdb/: 还因此发了一篇文章:http://nar.oxfordjournals.org/content/early/2013/11/07/nar.gkt1025.full.pdf,挺不错的!
关于driver mutation的理论最近也进化了很多,算是比较完善了吧,但是我一直没时间静下心来好好补充理论知识,很多软件,都只是用过,很多数据,也只是处理了一下,不知道为什么要去做,╮(╯▽╰)╭扯远了,开始谈这个软件吧!
mutsig软件是broadinstitute出品的,所以可靠性非常好咯,来源于一篇nature文章:http://www.nature.com/nature/journal/v505/n7484/full/nature12912.html,而该软件的地址是:http://www.broadinstitute.org/cancer/cga/mutsig_run 需要简单注册才能下载的。
该nature文章是这样描述这个软件的优点的:We used the most recent version of the MutSig suite of tools, which looks for three independent signals: highmutational burden relative to background expectation, accounting for heterogeneity; clustering of mutations within the gene; and enrichment of mutations in evolutionarily conserved sites. Wecombined the significance levels (P values) fromeach test to obtain a single significance level per gene (Methods).
这个软件需要安装matlab环境才能使用,所以我前面就写了教程,如何安装!http://www.bio-info-trainee.com/?p=1166
如果已经安装好了matlab环境,那么直接下载这个软件就可以使用了,软件解压就OK拉,而且人家还提供了测试文件!
软件下载后,解压可以看到里面的一个脚本,软件说明书写的非常简单,当然,使用这个软件也的确非常简单:
run_MutSigCV.sh <path_to_MCR> mutations.maf coverage.txt covariates.txt output.txt 即可,其中所有的数据都是可以下载的,
运行完了测试数据, 就证明你的软件安装没有问题啦!如果你只有突变数据的maf格式,maf格式可以参考:https://www.biostars.org/p/69222/ ,也可以使用该软件:如下
run_MutSigCV.sh <path_to_MCR> my_mutations.maf exome_full192.coverage.txt gene.covariates.txt my_results mutation_type_dictionary_file.txt chr_files_hg19
上面三个zip文件,都是可以在mutsig软件官网找到下载链接的,是必须下载的!使用很简单,就一个命令即可,但是把你的vcf突变数据做成该软件需要的maf格式,是一个难题!
在linux系统里面安装matlab运行环境mcr
matlab毕竟是收费软件,而且是有界面的。所以搞生物信息的都用R和linux替代了,但是很多高大上的单位,比如大名鼎鼎的broadinstitute,是用matlab的,所以他们开发的程序也会以matlab代码的形式发布。但是考虑到大多研究者用不起matlab,或者不会用,所以就用linux系统里面安装matlab运行环境来解决这个问题,我们仍然可以把人家写的matlab程序,在linux命令行下面,当做一个脚本来运行!
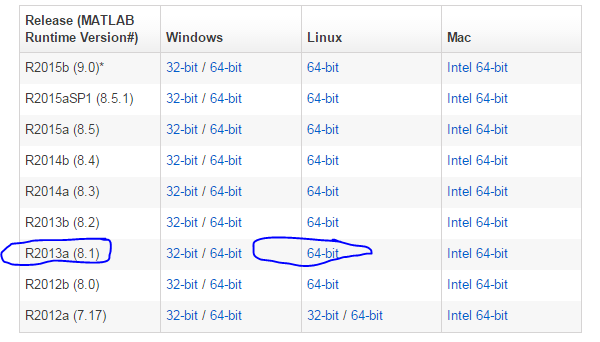
比如,这次我就需要用broadinstitute的一个软件:Mutsig,找cancer driver gene的,http://www.broadinstitute.org/cancer/cga/mutsig_run,但是我看了说明才发现,它是用matlab写的,所以我要想在我的服务器用,就必须按照安装matlab运行环境,在官网可以下载:http://www.mathworks.com/products/compiler/mcr/
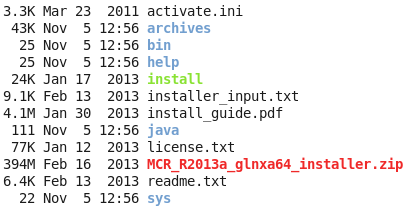
我这里选择的是R2013a (8.1),下载之后解压是这样的,压缩包约四百多M
然后直接在解压后的目录里面运行那个install即可,然后如果你的linux可以传送图像,那么就会想安装windows软件一样方便!如果你的linux是纯粹的命令行,那么,就需要一步步的命令行交互,选择安装地址,等等来安装了。
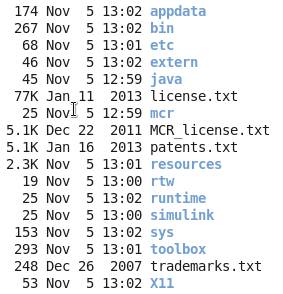
记住你安装之后,会显示一些环境变量给你,请千万要记住,然后自己去修改自己的环境变量,如果你忘记了,就需要搜索来解决环境变量的问题啦!安装之后是这样的:
请记住你的安装目录,以后你运行其它matlab相关的程序,都需要把这个安装目录,当做一个参数传给你的其它程序的!!!
如果你没有设置环境变量,就会出各种各样的错误,用下面这个脚本可以设置
其中MCRROOT一般是$path/biosoft/matlab_running/v81/ 这样的东西,请务必注意,LD_LIBRARY_PATH非常重要,非常重要,非常重要!!!!
MCRROOT=$1
echo ---
LD_LIBRARY_PATH=.:${MCRROOT}/runtime/glnxa64 ;
LD_LIBRARY_PATH=${LD_LIBRARY_PATH}:${MCRROOT}/bin/glnxa64 ;
LD_LIBRARY_PATH=${LD_LIBRARY_PATH}:${MCRROOT}/sys/os/glnxa64;
MCRJRE=${MCRROOT}/sys/java/jre/glnxa64/jre/lib/amd64 ;
LD_LIBRARY_PATH=${LD_LIBRARY_PATH}:${MCRJRE}/native_threads ;
LD_LIBRARY_PATH=${LD_LIBRARY_PATH}:${MCRJRE}/server ;
LD_LIBRARY_PATH=${LD_LIBRARY_PATH}:${MCRJRE}/client ;
LD_LIBRARY_PATH=${LD_LIBRARY_PATH}:${MCRJRE} ;
XAPPLRESDIR=${MCRROOT}/X11/app-defaults ;
export LD_LIBRARY_PATH;
export XAPPLRESDIR;
echo LD_LIBRARY_PATH is ${LD_LIBRARY_PATH};
如果你没有设置正确,那么会报一下的错误!
error while loading shared libraries: libmwmclmcrrt.so.8.1: cannot o pen shared object file: No such file or directory
error while loading shared libraries: libmwlaunchermain.so: cannot o pen shared object file: No such file or directory
等等!!!
WES(七)看de novo变异情况
de novo变异寻找这个也属于snp-calling的一部分,但是有点不同的就是该软件考虑了一家三口的测序文件,找de novo突变
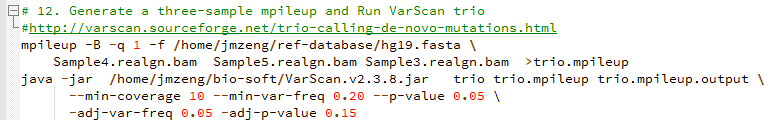
软件有介绍这个功能:http://varscan.sourceforge.net/trio-calling-de-novo-mutations.html
而且还专门有一篇文章讲ASD和autism与de novo变异的关系,但是文章不清不楚的,没什么意思
Trio Calling for de novo Mutations
Min coverage: 10
Min reads2: 4
Min var freq: 0.2
Min avg qual: 15
P-value thresh: 0.05
Adj. min reads2: 2
Adj. var freq: 0.05
Adj. p-value: 0.15
Reading input from trio.filter.mpileup
1371416525 bases in pileup file (137M的序列)
83123183 met the coverage requirement of 10 (其中有83M的测序深度大于10X)
145104 variant positions (132268 SNP, 12836 indel) (共发现15.5万的变异位点)
4403 were failed by the strand-filter
139153 variant positions reported (126762 SNP, 12391 indel)
502 de novo mutations reported (376 SNP, 126 indel) (真正属于 de novo mutations只有502个)
1734 initially DeNovo were re-called Germline
12 initially DeNovo were re-called MIE
3 initially DeNovo were re-called MultAlleles
522 initially MIE were re-called Germline
1 initially MIE were re-called MultAlleles
3851 initially Untransmitted were re-called Germline
然后我看了看输出的文件trio.mpileup.output.snp.vcf
软件是这样解释的:The output of the trio subcommand is a single VCF in which all variants are classified as germline (transmitted or untransmitted), de novo, or MIE.
- FILTER - mendelError if MIE, otherwise PASS
- STATUS - 1=untransmitted, 2=transmitted, 3=denovo, 4=MIE
- DENOVO - if present, indicates a high-confidence de novo mutation call
里面的信息量好还是不清楚
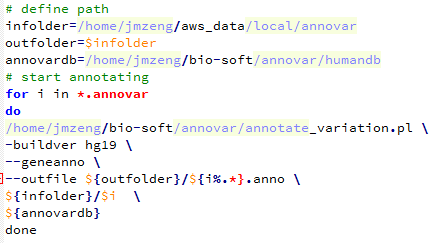
我用annovar注释了一下
/home/jmzeng/bio-soft/annovar/convert2annovar.pl -format vcf4 trio.mpileup.output.snp.vcf > trio.snp.annovar
/home/jmzeng/bio-soft/annovar/annotate_variation.pl -buildver hg19 --geneanno --outfile trio.snp.anno trio.snp.annovar /home/jmzeng/bio-soft/annovar/humandb
结果是:
A total of 132268 locus in VCF file passed QC threshold, representing 132809 SNPs (86633 transitions and 46176 transversions) and 3 indels/substitutions
可以看到最后被注释到外显子上面的突变有两万多个
23794 284345 3123333 trio.snp.anno.exonic_variant_function
这个应该是非常有意义的,但是我还没学会后面的分析。只能先做到这里了
WES(六)用annovar注释
使用annovar软件参考自:http://www.bio-info-trainee.com/?p=641
/home/jmzeng/bio-soft/annovar/convert2annovar.pl -format vcf4 Sample3.varscan.snp.vcf > Sample3.annovar
/home/jmzeng/bio-soft/annovar/convert2annovar.pl -format vcf4 Sample4.varscan.snp.vcf > Sample4.annovar
/home/jmzeng/bio-soft/annovar/convert2annovar.pl -format vcf4 Sample5.varscan.snp.vcf > Sample5.annovar
然后用下面这个脚本批量注释
Reading gene annotation from /home/jmzeng/bio-soft/annovar/humandb/hg19_refGene.txt ... Done with 50914 transcripts (including 11516 without coding sequence annotation) for 26271 unique genes
最后查看结果可知,真正在外显子上面的突变并不多
23515 Sample3.anno.exonic_variant_function
23913 Sample4.anno.exonic_variant_function
24009 Sample5.anno.exonic_variant_function
annovar软件就是把我们得到的十万多个snp分类了,看看这些snp分别是基因的哪些位置,是否引起蛋白突变
downstream
exonic
exonic;splicing
intergenic
intronic
ncRNA_exonic
ncRNA_intronic
ncRNA_splicing
ncRNA_UTR3
ncRNA_UTR5
splicing
upstream
upstream;downstream
UTR3
UTR5
UTR5;UTR3
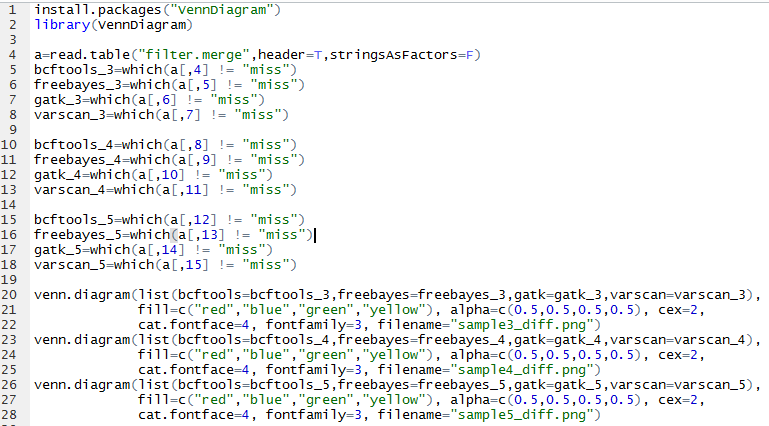
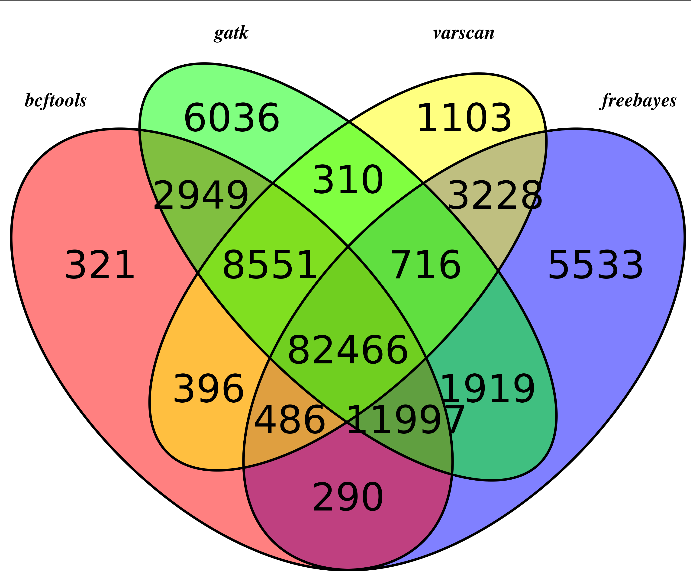
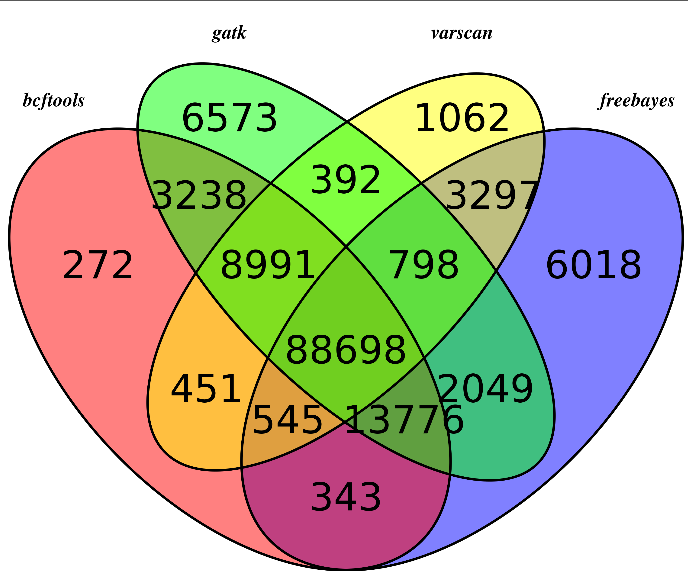
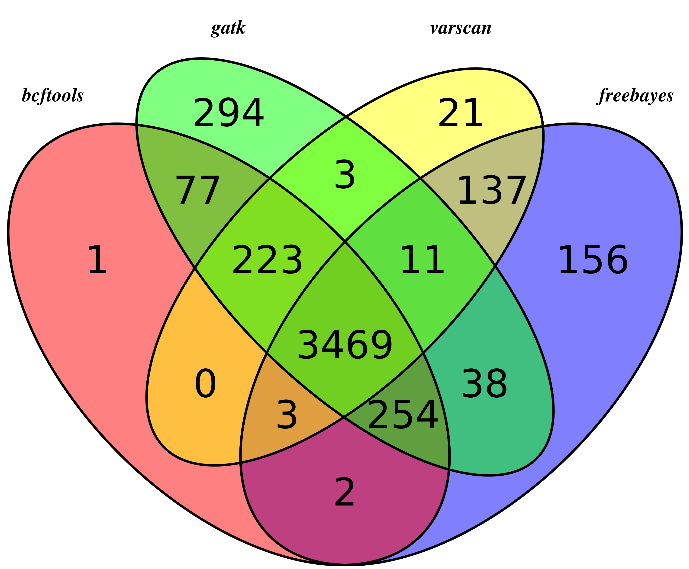
WES(五)不同软件比较
主要是画韦恩图看看,参考:http://www.bio-info-trainee.com/?p=893
对合并而且过滤的高质量snp信息来看看四种不同的snp calling软件的差异
我们用R语言来画韦恩图
可以看出不同软件的差异还是蛮大的,所以我只选四个软件的公共snp来进行分析
首先是sample3
然后是sample4
然后是sample5
可以看出,不同的软件差异还是蛮大的,所以我重新比较了一下,这次只比较,它们不同的软件在exon位点上面的snp的差异,毕竟,我们这次是外显子测序,重点应该是外显子snp
然后我们用同样的程序,画韦恩图,这次能明显看出来了,大部分的snp位点都至少有两到三个软件支持
所以,只有测序深度达到一定级别,用什么软件来做snp-calling其实影响并不大。
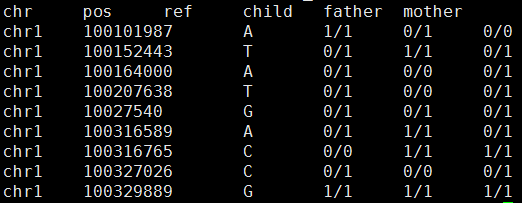
WES(四)不同个体的比较
3-4-5分别就是孩子、父亲、母亲
我对每个个体取他们的四种软件的公共snp来进行分析,并且只分析基因型,看看是否符合孟德尔遗传定律
结果如下:
粗略看起来好像很少不符合孟德尔遗传定律耶
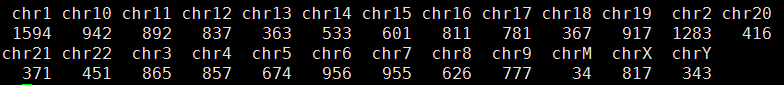
然后我写了程序计算
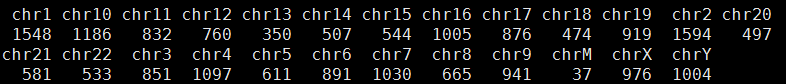
总共127138个可以计算的位点,共有18063个位点不符合孟德尔遗传定律,而且它们在染色体的分布情况如下
我检查了一下,不符合的原因,发现我把
chr1 100617887 C T:DP4=0,0,36,3 T:1/1:40 T:1/1:0,40:40 miss T:DP4=0,0,49,9 T:1/1:59 T:1/1:0,58:59 miss T:DP4=0,0,43,8 T:1/1:53 T:1/1:0,53:53 T:1/1:50
计算成了chr1 100617887 C 0/0 0/0 1/1 所以认为不符合,因为我认为只有四个软件都认为是snp的我才当作是snp的基因型,否则都是0/0
那么我就改写了程序,全部用gatk结果来计算。这次可以计算的snp有个176036,不符合的有20309,而且我看了不符合的snp的染色体分布,Y染色体有点异常
但是很失败,没什么发现!
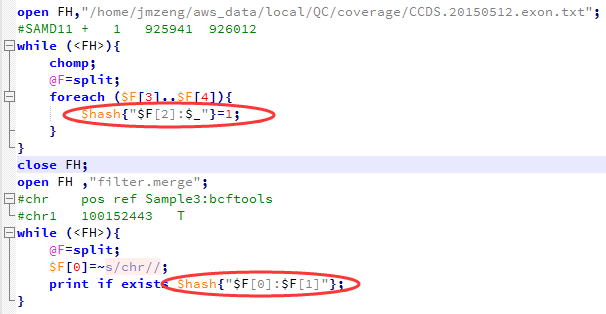
WES(三)snp-filter
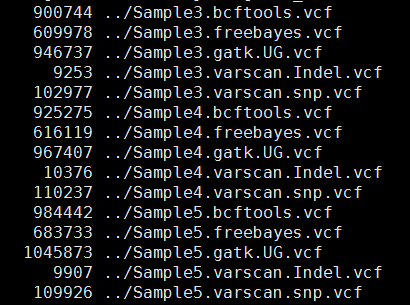
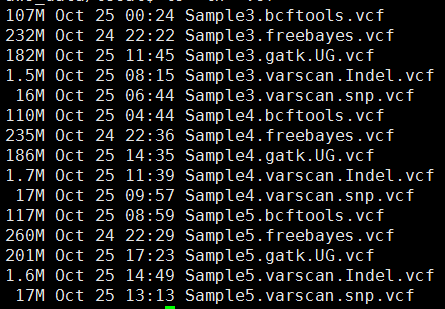
其中freebayes,bcftools,gatk都是把所有的snp细节都call出来了,可以看到下面这些软件的结果有的高达一百多万个snp,而一般文献都说外显子组测序可鉴定约8万个变异!
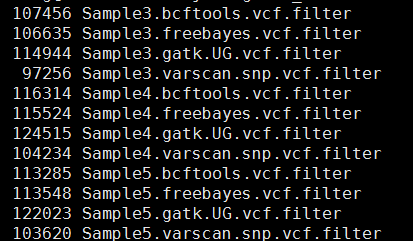
这样得到突变太多了,所以需要过滤。这里过滤的统一标准都是qual大于20,测序深度大于10。过滤之后的snp数量如下
perl -alne '{next if $F[5]<20;/DP=(\d+)/;next if $1<10;next if /INDEL/;/(DP4=.*?);/;print "$F[0]\t$F[1]\t$F[3]\t$F[4]:$1"}' Sample3.bcftools.vcf >Sample3.bcftools.vcf.filter
perl -alne '{next if $F[5]<20;/DP=(\d+)/;next if $1<10;next if /INDEL/;/(DP4=.*?);/;print "$F[0]\t$F[1]\t$F[3]\t$F[4]:$1"}' Sample4.bcftools.vcf >Sample4.bcftools.vcf.filter
perl -alne '{next if $F[5]<20;/DP=(\d+)/;next if $1<10;next if /INDEL/;/(DP4=.*?);/;print "$F[0]\t$F[1]\t$F[3]\t$F[4]:$1"}' Sample5.bcftools.vcf >Sample5.bcftools.vcf.filter
perl -alne '{next if $F[5]<20;/DP=(\d+)/;next if $1<10;next unless /TYPE=snp/;@tmp=split/:/,$F[9];print "$F[0]\t$F[1]\t$F[3]\t$F[4]:$tmp[0]:$tmp[1]"}' Sample3.freebayes.vcf > Sample3.freebayes.vcf.filter
perl -alne '{next if $F[5]<20;/DP=(\d+)/;next if $1<10;next unless /TYPE=snp/;@tmp=split/:/,$F[9];print "$F[0]\t$F[1]\t$F[3]\t$F[4]:$tmp[0]:$tmp[1]"}' Sample4.freebayes.vcf > Sample4.freebayes.vcf.filter
perl -alne '{next if $F[5]<20;/DP=(\d+)/;next if $1<10;next unless /TYPE=snp/;@tmp=split/:/,$F[9];print "$F[0]\t$F[1]\t$F[3]\t$F[4]:$tmp[0]:$tmp[1]"}' Sample5.freebayes.vcf > Sample5.freebayes.vcf.filter
perl -alne '{next if $F[5]<20;/DP=(\d+)/;next if $1<10;next if length($F[3]) >1;next if length($F[4]) >1;@tmp=split/:/,$F[9];print "$F[0]\t$F[1]\t$F[3]\t$F[4]:$tmp[0]:$tmp[1]:$tmp[2]"}' Sample3.gatk.UG.vcf >Sample3.gatk.UG.vcf.filter
perl -alne '{next if $F[5]<20;/DP=(\d+)/;next if $1<10;next if length($F[3]) >1;next if length($F[4]) >1;@tmp=split/:/,$F[9];print "$F[0]\t$F[1]\t$F[3]\t$F[4]:$tmp[0]:$tmp[1]:$tmp[2]"}' Sample4.gatk.UG.vcf >Sample4.gatk.UG.vcf.filter
perl -alne '{next if $F[5]<20;/DP=(\d+)/;next if $1<10;next if length($F[3]) >1;next if length($F[4]) >1;@tmp=split/:/,$F[9];print "$F[0]\t$F[1]\t$F[3]\t$F[4]:$tmp[0]:$tmp[1]:$tmp[2]"}' Sample5.gatk.UG.vcf >Sample5.gatk.UG.vcf.filter
perl -alne '{@tmp=split/:/,$F[9];next if $tmp[3]<10;print "$F[0]\t$F[1]\t$F[3]\t$F[4]:$tmp[0]:$tmp[3]"}' Sample3.varscan.snp.vcf >Sample3.varscan.snp.vcf.filter
perl -alne '{@tmp=split/:/,$F[9];next if $tmp[3]<10;print "$F[0]\t$F[1]\t$F[3]\t$F[4]:$tmp[0]:$tmp[3]"}' Sample4.varscan.snp.vcf >Sample4.varscan.snp.vcf.filter
perl -alne '{@tmp=split/:/,$F[9];next if $tmp[3]<10;print "$F[0]\t$F[1]\t$F[3]\t$F[4]:$tmp[0]:$tmp[3]"}' Sample5.varscan.snp.vcf >Sample5.varscan.snp.vcf.filter
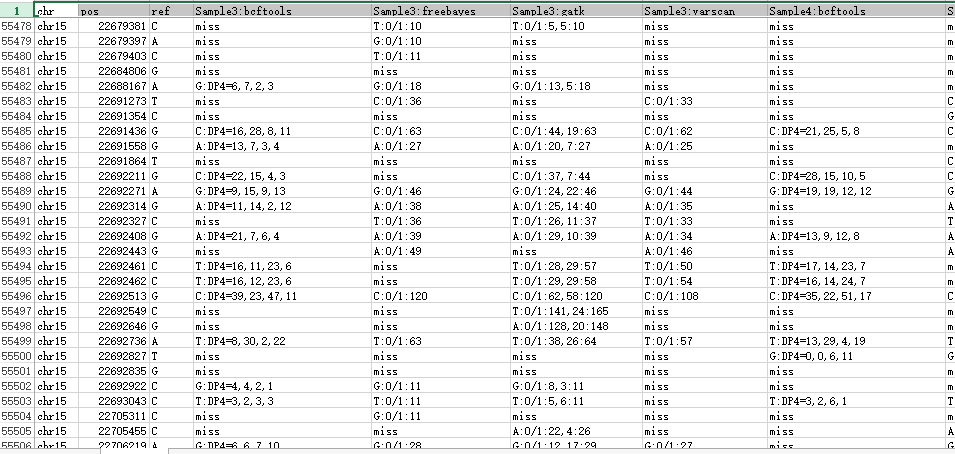
这样不同工具产生的snp记录数就比较整齐了,我们先比较四种不同工具的call snp的情况,然后再比较三个人的区别。
然后写了一个程序把所有的snp合并起来比较
WES(二)snp-calling
准备文件:下载必备的软件和参考基因组数据
1、软件
ps:还有samtools,freebayes和varscan软件,我以前下载过,这次就没有再弄了,但是下面会用到
2、参考基因组
3、参考 突变数据
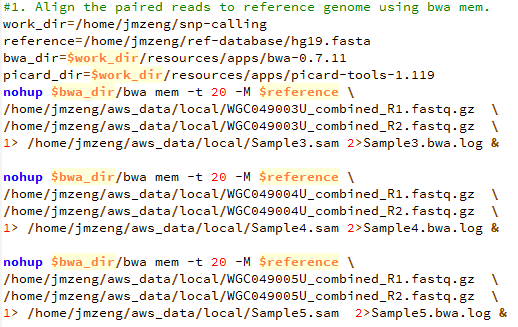
第一步,下载数据
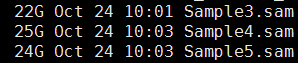
第二步,bwa比对
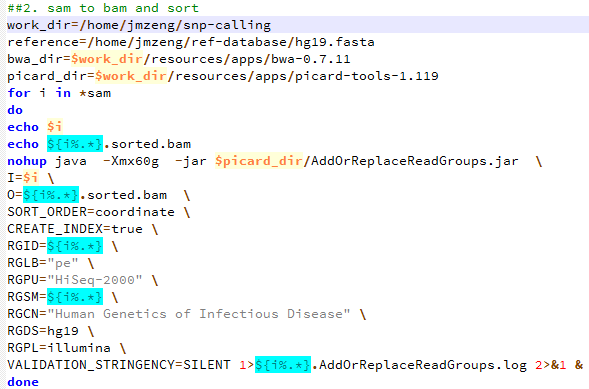
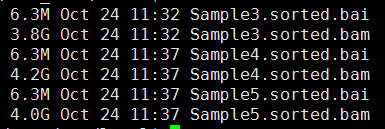
第三步,sam转为bam,并sort好
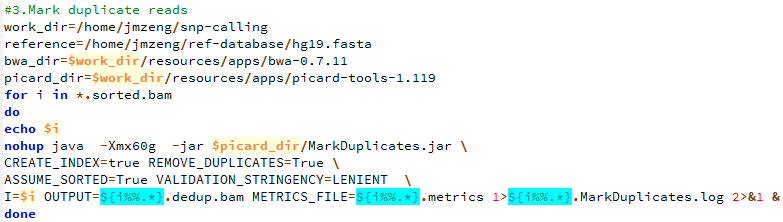
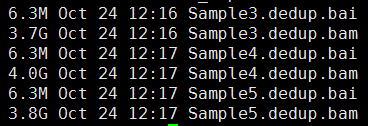
第四步,标记PCR重复,并去除
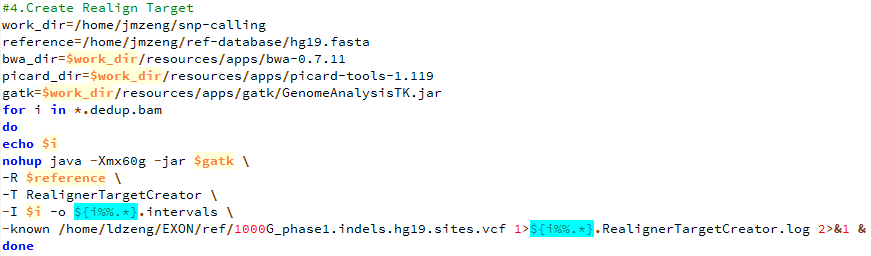
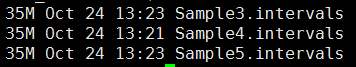
第五步,产生需要重排的坐标记录
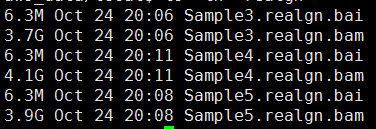
第六步,根据重排记录文件把比对结果重新比对
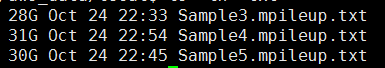
第七步,把最终的bam文件转为mpileup文件
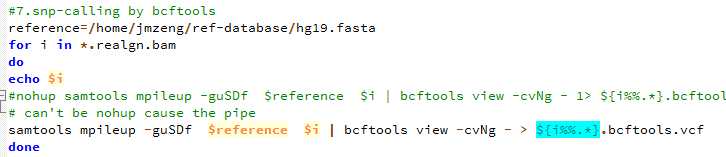
第八步,用bcftools 来call snp
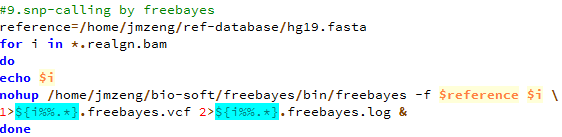
第九步,用freebayes来call snp
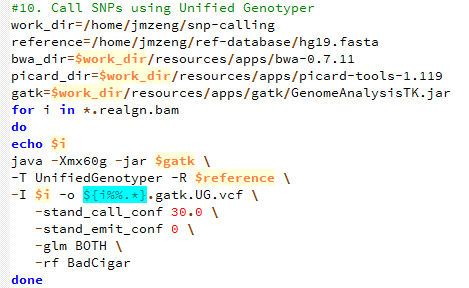
第十步,用gatk 来call snp
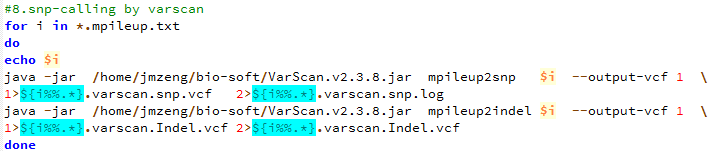
第十一步,用varscan来call snp
下面的图片是按照顺序来的,我就不整理了
WES(一)测序质量控制
这一步主要看看这些外显子测序数据的测序质量如何:
基因及外显子到底占基因组多少比例
很多文献都提到外显子组的序列仅占全基因组序列的1%左右,但大多数与疾病相关的变异位于外显子区。通过外显子组测序可鉴定约8万个变异,全基因组测序可鉴定300万个变异,因此与全基因组测序相比,外显子组测序不仅费用较低,数据阐释也更为简单。外显子组测序技术以其经济有效的优势广泛应用于孟德尔遗传病、罕见综合征及复杂疾病的研究,并于2010年被Science杂志评为十大突破之一。所以我一直想验证一下外显子到底占基因组什么样的百分比,是哪些位点。
首先我们拿到外显子记录和基因信息,下面是通过R来从bioconductor中心提取数据
library("TxDb.Hsapiens.UCSC.hg19.knownGene")
txdb <- TxDb.Hsapiens.UCSC.hg19.knownGene
txdb
exon_txdb=exons(txdb)
genes_txdb=genes(txdb)
tmp=as.data.frame(exon_txdb)
write.table(tmp,'exon.pos',row.names=F)
tmp=as.data.frame(genes_txdb)
write.table(tmp,'gene.pos',row.names=F)
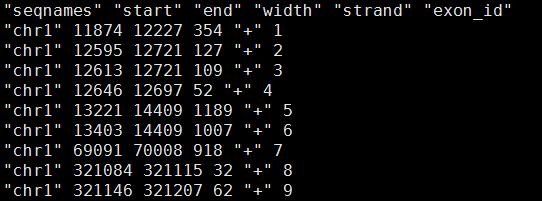
首先我们看看我刚才从TxDb.Hsapiens.UCSC.hg19.knownGene包里面提取的外显子记录文件exon.pos
我简单用脚本统计了一下:perl -alne '{$tmp+=$F[3]} END{print $tmp}' exon.pos
共289970个外显子记录,长度共98759283bp,也就是98.5Mb的区域都是外显子,感觉有点不大对,毕竟真正的外显子也就三十多M的,所以必须有些外显子是并列关系,也就是有的外显子包含着另外一些外显子,然后我写脚本检查了一下,的确太多了,这样的重复!
也可以去NCBI里面拿到 consensus coding sequence (CCDS)记录:ftp://ftp.ncbi.nlm.nih.gov/pub/CCDS/current_human/
那么我再看看NCBI里面拿到 consensus coding sequence (CCDS)记录CCDS.20150512.txt文件。
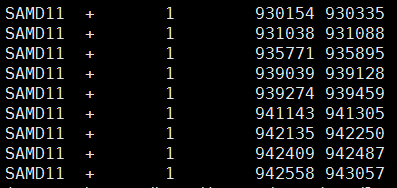
共33140行,一个基因一行,所以需要格式化成外显子文件,简单看了一下文件的列,很容易格式化。每一行的信息如下
1 NC_000001.11 SAMD11 148398 CCDS2.2 Public + 925941 944152 [925941-926012, 930154-930335, 931038-931088, 935771-935895, 939039-939128, 939274-939459, 941143-941305, 942135-942250, 942409-942487, 942558-943057, 943252-943376, 943697-943807, 943907-944152] Identical
用下面这个脚本格式化
格式化后的文件如下
外显子记录增加到了346493个,我再用单行命令计算长度,是56321786bp,这次只有56M了,还是有点大,所以应该是还有重复!
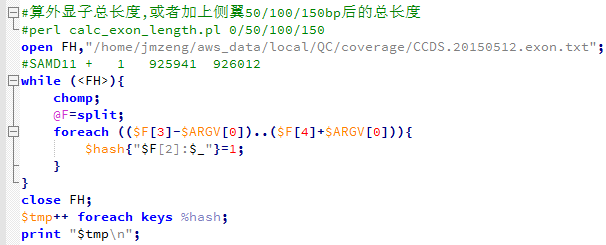
所以,我写了一个脚本来精确计算外显子的总长度,不能那样马马虎虎的用单行命令了。
这次才是34729283bp,也就是约35M,根很多文献里面说的都一样!
同理,我统计了一下外显子的侧翼长度
54692160 外显子加上前后50bp
73066288 外显子加上前后100bp
90362533 外显子加上前后150bp
而hg19版本基因组里面有着entrez gene ID号的基因是23056个基因,所以我接下来探究一下这些基因的信息!
我们首先看看基因与基因之间的交叉情况
其中有12454266bp的位点,是多个外显子共有的,可能是一个基因的多个外显子,或者是不同基因的外显子
我们主要看看不同基因的交叉情况
不同基因交叉情况共516种,共涉及498种基因!
ZNF341 NFS1
ZNF397 ZSCAN30
ZNF428 SRRM5
ZNF436-AS1 RPL11
ZNF578 ZNF137P
ZNF619 ZNF621
ZNF816 ZNF816-ZNF321P
ZSCAN26 ZSCAN31
ZSCAN5A ZNF542P
ZZEF1 ANKFY1
我随便在数据库里面搜索一下验证,发现这些基因的确是交叉重叠的
R语言也可以用hash啦
本来想着是如何习惯R里面的list对象,来实现hash的功能,无意中发现了居然还有这个包
https://cran.r-project.org/web/packages/hash/hash.pdf
我简单看了看文档,的确跟perl里面的hash功能类似,非常好用。
创建一个hash很简单
h <- hash( keys=letters, values=1:26 )
h <- hash( letters, 1:26 )
类似于下面的列表
L=setNames(as.list(LETTERS),1:26)
如果是列表要提取keys需要用names函数,如果需要提取values,需要用unlist函数,而用了hash之后,这两个函数就可以独自运行啦
还有很多其它函数,其实在list中也可以实现,主要是看你对哪种语法更熟悉,感觉自己现在编程能力差不多算是小有所成了,看什么都一样了,要是以前学R的时候看到了这个hash包,我肯定会很兴奋的!
clear signature(x = "hash"): Remove all key-value pairs from hash
del signature(x = "ANY", hash = "hash"): Remove specified key-value pairs from hash
has.key signature(key = "ANY", hash = "hash"): Test for existence of key
is.empty signature(x = "hash"): Test if no key-values are assigned
length signature(x = "hash"): Return number of key-value pairs from the hash
keys signature(hash = "hash"): Retrieve keys from hash
values signature(x = "hash"): Retrieve values from hash
copy signature(x = "hash"): Make a copy of a hash using a new environment.
format signature(x = "hash"): Internal function for displaying hash