最近打理自己的生信菜鸟团博客发现阿里云又开始搞活动了,这次虽然不是2年免费,不过也差不多,三年才五百多块钱! Continue reading
Category Archives: tutorial
神技能-自动化批量从PDF里面提取表格
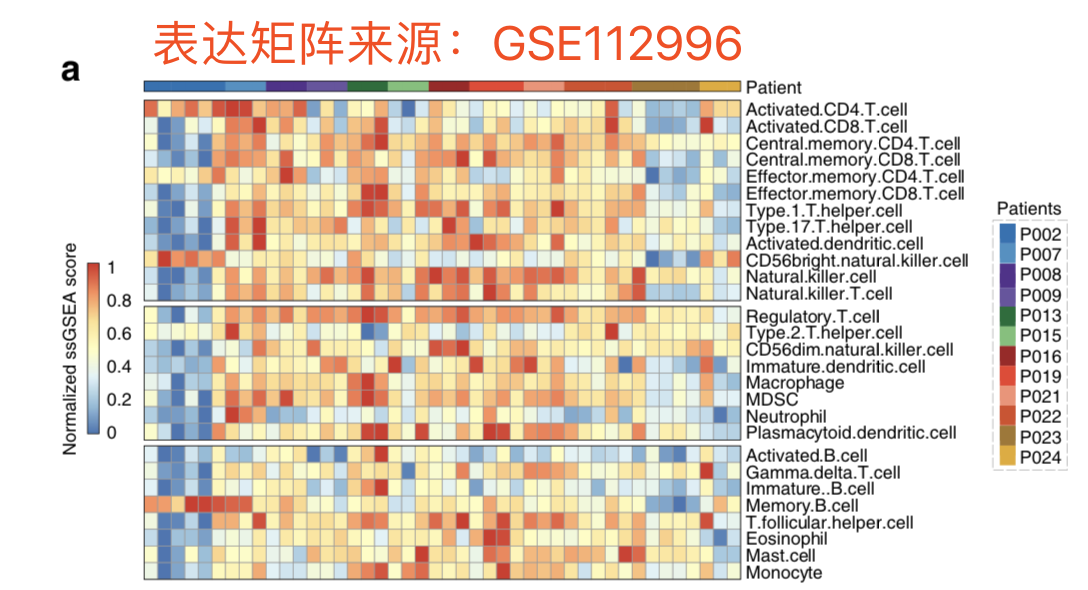
最近给学徒布置了一个作业,是一篇文章的数据图表重现,如下:
想做单细胞数据分析,完成一个R考核题
打开你的Rstudio,运行下面的代码: Continue reading
hg38按照200k分区间统计测序深度及GC含量
使用DSS包多种方式检验差异甲基化信号区域
一起学一个包吧! Continue reading
conda测试2019镜像问题
最近很多人反映conda镜像挂掉的问题,所以我有必要给粉丝测试一下: Continue reading
为什么要学编程-excel基因名错误的优雅解决方案
为什么要学编程
帮同学处理一下他从公司拿到的差异分析结果,当然,给我的是Excel表格,老规矩,导出csv然后读入R,然后准备顺手画个火山图,做个GO/KEGG富集分析。下意识的看了看数据结构,然后顺手按照基因名排序了一下,哈哈哈~ Continue reading
找变异的流程
找变异简单点说,就是把高通量测序得到的成千上万条序列片段比对到合适的参考基因组,找到那些成
功比对的片段与参考基因组的微小差异情况。 那么就涉及到存储测序数据的fastq数据格式,比对的工具,比对后的sam格式,找微小差异的工具,差异结果的vcf文件,每个步骤的软件选择,参数 调整。当然,最重要的是走通整个流程,明白自己在做什么。
自学无参RNAseq数据分析第一讲之参考文献解读
这是我为新创办的 生信技能树 论坛写的帖子,也适合本博客,所以转载过来: http://www.biotrainee.com/thread-243-1-1.html
以前做的都是有参转录组分析,只需要找到参考基因组和注释文件,然后走QC-->alignment-->counts->DEG-->annotation的流程图即可。
现在开始学习新的东西了,就是无参转录组分析,这里记录一下自己的学习笔记,首先还是资料收集,这次,我就针对性的看5个 全流程化的转录组 de novo 分析 文章,如下:
http://bmcgenomics.biomedcentral.com/articles/10.1186/1471-2164-15-554 2014年栀子花的花瓣衰老的标准de novo 转录组分析,数据如下:用Trinity做组装,用NCBI non-redundant (Nr) database库做注释,做了差异分析(栀子花花期分成4个阶段),GO/KEGG注释,然后做了RT-qPCR的实验验证。
多做了一个 Clusters of Orthologus Groups (COG)的数据库注释
|
Raw Reads
|
Clean Reads
|
Contigs
|
Unigenes
|
Annotated
|
|
|
Transcriptome
|
55,092,396
|
50,335,672
|
102,263
|
57,503
|
39,459
|
http://bmcgenomics.biomedcentral.com/articles/10.1186/1471-2164-15-236 2014 巴西橡胶树的研究,是一个综合多组织样本的RNA库,ployT建库,454测序,用的是est2Assembly 和gsassembler 软件做组装,用 NCBI RefSeq, Plant Protein Database 做注释,因为没有分组,所以不必做差异分析,只需要找SNV和SSR标记即可,最后也是做GO/KEGG注释
https://bmcgenomics.biomedcentral.com/articles/10.1186/s12864-016-2633-2 2015 萝卜,用illumina进行转录组测序,用Trinity组装,用RPKM值算unigene的表达量,也是用 BLASTx来对Trinity结果进行注释,注释到NR,NT,Swiss-Prot,GO,COG,kegg数据库,其中GO注释用的是Blast2GO,最后也做了RT-qPCR 实验验证,某些基因在leaf里面的表达量显著高于其它tissue,有原始数据:http://www.ncbi.nlm.nih.gov/sra/?term=SRX1671013
转录组分析结果结果:A total of 54.64 million clean reads and 111,167 contigs representing 53,642 unigenes were obtained from the radish leaf transcriptome.
http://www.nature.com/articles/srep08259 2015 芹菜 叶片发育中木质素的探究,测序的reads是A total of 32,477,416 quality reads were recorded for the leaves at Stage 1, 53,675,555 at Stage 2, and 27,158,566 at Stage 3, respectively.,也是用Trinity组装,kmer值设为25,组装结果:33,213 unigenes with an average length of 1,478 bp, a maximum length of 17,075 bp, and an N50 of 2,060 bp,然后用eggNOG/GO/KEGG数据库来注释。文章正文给了所用到的软件和数据库的详细链接
最后还用了 real-time PCR assays 来看 roots, stems, petioles, and leaf blade 这些组织的基因表达差异情况
http://journals.plos.org/plosone/article?id=10.1371/journal.pone.0128659 对 三疣梭子蟹 的卵巢和睾丸的转录组研究,,也是标准的转录组de novo 分析流程,非常值得借鉴
NCBI有上传原始数据:SRR1920180 和SRR1920180
总结好这5篇文献的数据分析流程,就差不多明白如何做无参的转录组de novo分析了
RNAseq数据完整生物信息分析流程第一讲之文献数据下载
我这里拿的是bioconductor里面最常用的airway数据,因为差异表达分析在bioconductor里面是重点,它们这些包在介绍自己的算法以及做示范的时候都用的这个数据。可以在GEO数据库里面看到信息描述:http://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE52778 可以看到是Illumina HiSeq 2000 (Homo sapiens) ,75bp paired-end 这个信息很重要,决定了下载sra数据之后如何解压以及如何比对。也可以看到作者把所有的测序原始数据都上传到了SRA中心:http://www.ncbi.nlm.nih.gov/sra?term=SRP033351 ,这里可以在linux服务器上面写一个简单的脚本批量下载所有的测序数据,然后根据GEO里面描述的metadata把原始数据改名。
for ((i=508;i<=523;i++)) ;do wget ftp://ftp-trace.ncbi.nlm.nih.gov/sra/sra-instant/reads/ByStudy/sra/SRP/SRP033/SRP033351/SRR1039$i/SRR1039$i.sra;done
ls *sra |while read id; do ~/biosoft/sratoolkit/sratoolkit.2.6.3-centos_linux64/bin/fastq-dump --split-3 $id;done
需要自己看SRA里面的数据记录,上面的脚本不难写出,然后因为是Illumina的双端测序,所以我们用fastq-dump --split-3命令来把sra格式数据转换为fastq,但是因为这里有16个测序数据,所以最好是同步改名,我这里用脚本批量生成改名脚本如下:
为了节省空间,我用了--gzip压缩,该文件名,用-A参数。
nohup ~/biosoft/sratoolkit/sratoolkit.2.6.3-centos_linux64/bin/fastq-dump --split-3 --gzip -A N61311_untreated SRR1039508.sra &
nohup ~/biosoft/sratoolkit/sratoolkit.2.6.3-centos_linux64/bin/fastq-dump --split-3 --gzip -A N61311_Dex SRR1039509.sra &
nohup ~/biosoft/sratoolkit/sratoolkit.2.6.3-centos_linux64/bin/fastq-dump --split-3 --gzip -A N61311_Alb SRR1039510.sra &
nohup ~/biosoft/sratoolkit/sratoolkit.2.6.3-centos_linux64/bin/fastq-dump --split-3 --gzip -A N61311_Alb_Dex SRR1039511.sra &
nohup ~/biosoft/sratoolkit/sratoolkit.2.6.3-centos_linux64/bin/fastq-dump --split-3 --gzip -A N052611_untreated SRR1039512.sra &
nohup ~/biosoft/sratoolkit/sratoolkit.2.6.3-centos_linux64/bin/fastq-dump --split-3 --gzip -A N052611_Dex SRR1039513.sra &
nohup ~/biosoft/sratoolkit/sratoolkit.2.6.3-centos_linux64/bin/fastq-dump --split-3 --gzip -A N052611_Alb SRR1039514.sra &
nohup ~/biosoft/sratoolkit/sratoolkit.2.6.3-centos_linux64/bin/fastq-dump --split-3 --gzip -A N052611_Alb_Dex SRR1039515.sra &
nohup ~/biosoft/sratoolkit/sratoolkit.2.6.3-centos_linux64/bin/fastq-dump --split-3 --gzip -A N080611_untreated SRR1039516.sra &
nohup ~/biosoft/sratoolkit/sratoolkit.2.6.3-centos_linux64/bin/fastq-dump --split-3 --gzip -A N080611_Dex SRR1039517.sra &
nohup ~/biosoft/sratoolkit/sratoolkit.2.6.3-centos_linux64/bin/fastq-dump --split-3 --gzip -A N080611_Alb SRR1039518.sra &
nohup ~/biosoft/sratoolkit/sratoolkit.2.6.3-centos_linux64/bin/fastq-dump --split-3 --gzip -A N080611_Alb_Dex SRR1039519.sra &
nohup ~/biosoft/sratoolkit/sratoolkit.2.6.3-centos_linux64/bin/fastq-dump --split-3 --gzip -A N061011_untreated SRR1039520.sra &
nohup ~/biosoft/sratoolkit/sratoolkit.2.6.3-centos_linux64/bin/fastq-dump --split-3 --gzip -A N061011_Dex SRR1039521.sra &
nohup ~/biosoft/sratoolkit/sratoolkit.2.6.3-centos_linux64/bin/fastq-dump --split-3 --gzip -A N061011_Alb SRR1039522.sra &
nohup ~/biosoft/sratoolkit/sratoolkit.2.6.3-centos_linux64/bin/fastq-dump --split-3 --gzip -A N061011_Alb_Dex SRR1039523.sra &
可以看到这里的16个样本来源于同样的4个人,是HASM细胞系,处理详情如下:
1) no treatment;2) treatment with a β2-agonist (i.e. Albuterol, 1μM for 18h);3) treatment with a glucocorticosteroid (i.e. Dexamethasone (Dex), 1μM for 18h);4) simultaneous treatment with a β2-agonist and glucocorticoid
下载的sra大小如下:
-rw-rw-r-- 1 jmzeng jmzeng 1.6G Aug 9 04:21 SRR1039508.sra
-rw-rw-r-- 1 jmzeng jmzeng 1.5G Aug 9 05:20 SRR1039509.sra
-rw-rw-r-- 1 jmzeng jmzeng 1.6G Aug 9 06:14 SRR1039510.sra
-rw-rw-r-- 1 jmzeng jmzeng 1.5G Aug 9 07:05 SRR1039511.sra
-rw-rw-r-- 1 jmzeng jmzeng 2.1G Aug 9 08:07 SRR1039512.sra
-rw-rw-r-- 1 jmzeng jmzeng 2.3G Aug 9 09:17 SRR1039513.sra
-rw-rw-r-- 1 jmzeng jmzeng 3.1G Aug 9 10:56 SRR1039514.sra
-rw-rw-r-- 1 jmzeng jmzeng 1.9G Aug 9 11:56 SRR1039515.sra
-rw-rw-r-- 1 jmzeng jmzeng 2.1G Aug 9 13:02 SRR1039516.sra
-rw-rw-r-- 1 jmzeng jmzeng 2.6G Aug 9 14:16 SRR1039517.sra
-rw-rw-r-- 1 jmzeng jmzeng 2.3G Aug 9 15:17 SRR1039518.sra
-rw-rw-r-- 1 jmzeng jmzeng 2.0G Aug 9 16:05 SRR1039519.sra
-rw-rw-r-- 1 jmzeng jmzeng 2.1G Aug 9 16:56 SRR1039520.sra
-rw-rw-r-- 1 jmzeng jmzeng 2.4G Aug 9 17:57 SRR1039521.sra
-rw-rw-r-- 1 jmzeng jmzeng 2.0G Aug 9 18:46 SRR1039522.sra
-rw-rw-r-- 1 jmzeng jmzeng 1.4G Aug 9 19:28 SRR1039523.sra
解压后成双端测序的fastq数据如下:
-rw-rw-r-- 1 jmzeng jmzeng 2.5G Aug 9 20:12 N052611_Alb_1.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 2.5G Aug 9 20:12 N052611_Alb_2.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 1.3G Aug 9 20:44 N052611_Alb_Dex_1.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 1.3G Aug 9 20:44 N052611_Alb_Dex_2.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 289M Aug 9 20:44 N052611_Alb_Dex.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 951M Aug 9 20:59 N052611_Dex_1.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 954M Aug 9 20:59 N052611_Dex_2.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 1.7G Aug 9 20:53 N052611_untreated_1.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 1.7G Aug 9 20:53 N052611_untreated_2.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 1.5G Aug 9 20:45 N061011_Alb_1.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 1.5G Aug 9 20:45 N061011_Alb_2.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 1.9G Aug 9 20:59 N061011_Alb_Dex_1.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 1.9G Aug 9 20:59 N061011_Alb_Dex_2.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 16M Aug 9 20:45 N061011_Alb.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 1.4G Aug 9 20:48 N061011_Dex_1.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 1.4G Aug 9 20:48 N061011_Dex_2.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 1.2G Aug 9 20:00 N061011_untreated_1.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 1.2G Aug 9 20:00 N061011_untreated_2.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 759M Aug 9 20:00 N061011_untreated.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 1.9G Aug 9 20:03 N080611_Alb_1.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 1.9G Aug 9 20:03 N080611_Alb_2.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 1.3G Aug 9 19:59 N080611_Alb_Dex_1.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 1.3G Aug 9 19:59 N080611_Alb_Dex_2.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 535M Aug 9 19:59 N080611_Alb_Dex.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 2.1G Aug 9 20:06 N080611_Dex_1.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 2.1G Aug 9 20:06 N080611_Dex_2.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 1.6G Aug 9 20:01 N080611_untreated_1.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 1.6G Aug 9 20:01 N080611_untreated_2.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 1.3G Aug 9 08:09 N61311_Alb_1.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 1.3G Aug 9 08:09 N61311_Alb_2.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 1.3G Aug 9 08:08 N61311_Alb_Dex_1.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 1.3G Aug 9 08:08 N61311_Alb_Dex_2.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 1.2G Aug 9 08:07 N61311_Dex_1.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 1.2G Aug 9 08:07 N61311_Dex_2.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 1.3G Aug 9 08:09 N61311_untreated_1.fastq.gz
-rw-rw-r-- 1 jmzeng jmzeng 1.3G Aug 9 08:09 N61311_untreated_2.fastq.gz
接下来所有的分析就基于此数据啦
生信人必学ftp站点之1000genomes
千人基因组计划的重要性我也不想多说了,由于时间跨度比较长,最终的数据不只是一千人,最新版共有NA编号开头的1182个人,HG开头的1768个人!它的官方网站是:有一个ppt讲得很清楚如何通过官网做的data portal来下载数据:https://www.genome.gov/pages/research/der/ichg-1000genomestutorial/how_to_access_the_data.pdf 我不喜欢可视化的界面,我比较喜欢直接进入ftp自己翻需要的数据,千人基因组计划不仅仅有自己的ftp站点,而且在NCBI,EBI和sanger研究所里面也有数据源可以下载, 是非常丰富的生信入门资源!
生信人必学ftp站点之NCBI-GEO
NCBI的重要性我就不多说了,Gene Expression Omnibus database (GEO)是由NCBI负责维护的一个数据库,设计初衷是为了收集整理各种表达芯片数据,但是后来也加入了甲基化芯片,lncRNA,miRNA,CNV芯片等各种芯片,甚至高通量测序数据!所有的数据均可以在ftp站点下载:ftp://ftp-trace.ncbi.nih.gov/geo/ Continue reading
ChIP-Seq文献数据重新分析解读第二例
ChIP-Seq文献数据重新分析解读第一例
自学lncRNA-seq数据分析第一讲~学习大纲
自学CHIP-seq分析第九讲~CHIP-seq可视化大全
讲到这里,我们的自学CHIP-seq分析系列教程就告一段落了,当然,我会随时查漏补缺,根据读者的反馈来更新着系列教程。其实可视化这已经是一个比较复杂的方向了,不仅仅是针对于CHIP-seq数据。可视化本身是发文章的先决条件,而让人一目了然图片也说明了数据分析人员对数据本身的理解。我这里就列出一些目录和一些工具,和ppt。这个主要靠大家自学了,而且我博客空间有限,就不上传一大堆图片了,大家随便找一些经典的paper里面都会有很多可视化分析。
首先强烈推荐两个网页版工具,针对找到的peaks可视化:
然后再推荐一个哈佛刘小乐实验室出品的软件,也是专门为了作图http://liulab.dfci.harvard.edu/CEAS/usermanual.html
自学CHIP-seq分析第八讲~寻找motif
自学CHIP-seq分析第七讲~peaks注释
经过前面的CHIP-seq测序数据处理的常规分析,我们已经成功的把测序仪下机数据变成了BED格式的peaks记录文件,我选取的这篇文章里面做了4次CHIP-seq实验,分别是两个重复的野生型MCF7细胞系的 BAF155 immunoprecipitates和两个重复的突变型MCF7细胞系的 BAF155 immunoprecipitates,这样通过比较野生型和突变型MCF7细胞系的 BAF155 immunoprecipitates的结果的不同就知道该细胞系的BAF155 突变,对它在全基因组的结合功能的影响啦。
#我这里直接从GEO里面下载了peaks结果,它们详情如下:wc -l *bed
6768 GSM1278641_Xu_MUT_rep1_BAF155_MUT.peaks.bed
3660 GSM1278643_Xu_MUT_rep2_BAF155_MUT.peaks.bed
11022 GSM1278645_Xu_WT_rep1_BAF155.peaks.bed
5260 GSM1278647_Xu_WT_rep2_BAF155.peaks.bed
49458 GSM601398_Ini1HeLa-peaks.bed
24477 GSM601398_Ini1HeLa-peaks-stringent.bed
12725 GSM601399_Brg1HeLa-peaks.bed
12316 GSM601399_Brg1HeLa-peaks-stringent.bed
46412 GSM601400_BAF155HeLa-peaks.bed
37920 GSM601400_BAF155HeLa-peaks-stringent.bed
30136 GSM601401_BAF170HeLa-peaks.bed
25432 GSM601401_BAF170HeLa-peaks-stringent.bed
每个BED的peaks记录,本质是就3列是需要我们注意的,就是染色体,以及在该染色体上面的起始和终止坐标,如下:
#PeakID chr start end strand Normalized Tag Count region size findPeaks Score Clonal Fold Change
chr20 52221388 52856380 chr20-8088 41141 +
chr20 45796362 46384917 chr20-5152 31612 +
chr17 59287502 59741943 chr17-2332 29994 +
chr17 59755459 59989069 chr17-667 19943 +
chr20 52993293 53369574 chr20-7059 12642 +
chr1 121482722 121485861 chr1-995 9070 +
chr20 55675229 55855175 chr20-6524 7592 +
chr3 64531319 64762040 chr3-4022 7213 +
chr20 49286444 49384563 chr20-4482 6165 +
我们所谓的peaks注释,就是想看看该peaks在基因组的哪一个区段,看看它们在各种基因组区域(基因上下游,5,3端UTR,启动子,内含子,外显子,基因间区域,microRNA区域)分布情况,但是一般的peaks都有近万个,所以需要批量注释,如果脚本学的好,自己下载参考基因组的GFF注释文件,完全可以自己写一个,我这里会介绍一个R的bioconductor包ChIPpeakAnno来做CHIP-seq的peaks注释,下面的包自带的示例:
library(ChIPpeakAnno)
bed <- system.file("extdata", "MACS_output.bed", package="ChIPpeakAnno")
gr1 <- toGRanges(bed, format="BED", header=FALSE)
## one can also try import from rtracklayer
library(rtracklayer)
gr1.import <- import(bed, format="BED")
identical(start(gr1), start(gr1.import))
gr1[1:2]
gr1.import[1:2] #note the name slot is different from gr1
gff <- system.file("extdata", "GFF_peaks.gff", package="ChIPpeakAnno")
gr2 <- toGRanges(gff, format="GFF", header=FALSE, skip=3)
ol <- findOverlapsOfPeaks(gr1, gr2)
makeVennDiagram(ol)##还可以用binOverFeature来根据特定的GRanges对象(通常是TSS)来画分布图
## Distribution of aggregated peak scores or peak numbers around transcript start sites.
可以看到这个包使用起来非常简单,只需要把我们做好的peaks文件(GSM1278641_Xu_MUT_rep1_BAF155_MUT.peaks.bed等等)用toGRanges或者import读进去,成一个GRanges对象即可,上面的代码是比较两个peaks文件的overlap。然后还可以根据R很多包都自带的数据来注释基因组特征:
data(TSS.human.GRCh37) ## 主要是借助于这个GRanges对象来做注释,也可以用getAnnotation来获取其它GRanges对象来做注释
## featureType : TSS, miRNA, Exon, 5'UTR, 3'UTR, transcript or Exon plus UTR
peaks=MUT_rep1_peaks
macs.anno <- annotatePeakInBatch(peaks, AnnotationData=TSS.human.GRCh37,
output="overlapping", maxgap=5000L)## 得到的macs.anno对象就是已经注释好了的,每个peaks是否在基因上,或者距离基因多远,都是写的清清楚楚
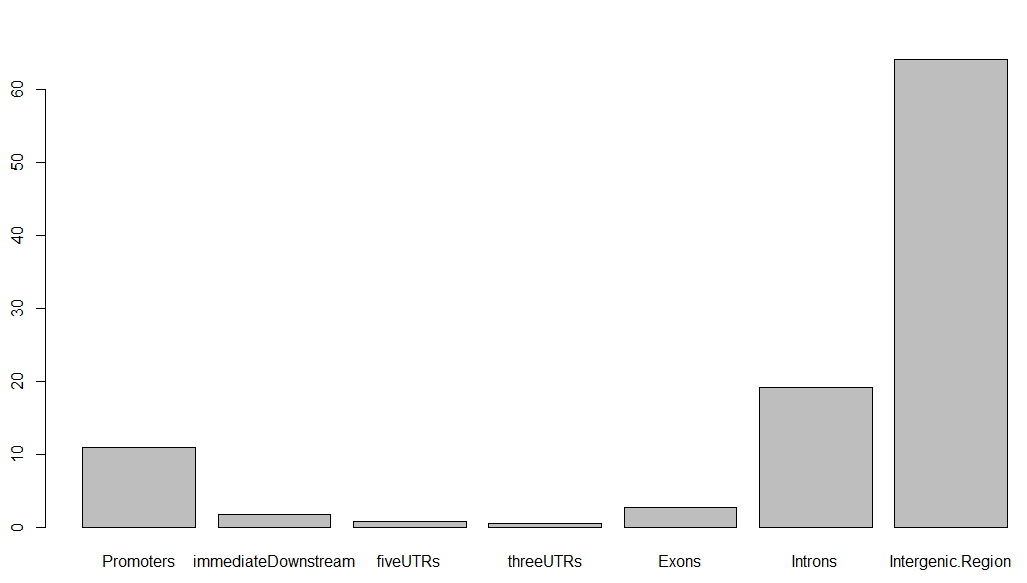
if(require(TxDb.Hsapiens.UCSC.hg19.knownGene)){
aCR<-assignChromosomeRegion(peaks, nucleotideLevel=FALSE,
precedence=c("Promoters", "immediateDownstream",
"fiveUTRs", "threeUTRs",
"Exons", "Introns"),
TxDb=TxDb.Hsapiens.UCSC.hg19.knownGene)
barplot(aCR$percentage)
}
得到的条形图如下,虽然很丑,但这就是peaks注释的精髓,搞清楚每个peaks在基因组的位置特征:
同理,对每个peaks文件,都可以做类似的分析!
但是对多个peaks文件,比如本文中的,想比较野生型和突变型MCF7细胞系的 BAF155 immunoprecipitates的结果的不同,就需要做peaks之间的差异分析,已经后续的差异基因注释啦
当然,值得一提的是peaks注释我更喜欢网页版工具,反正peaks文件非常小,直接上传到别人做好的web tools,就可立即出一大堆可视化图表分析结果啦,大家可以去试试看:
自学CHIP-seq分析第六讲~寻找peaks
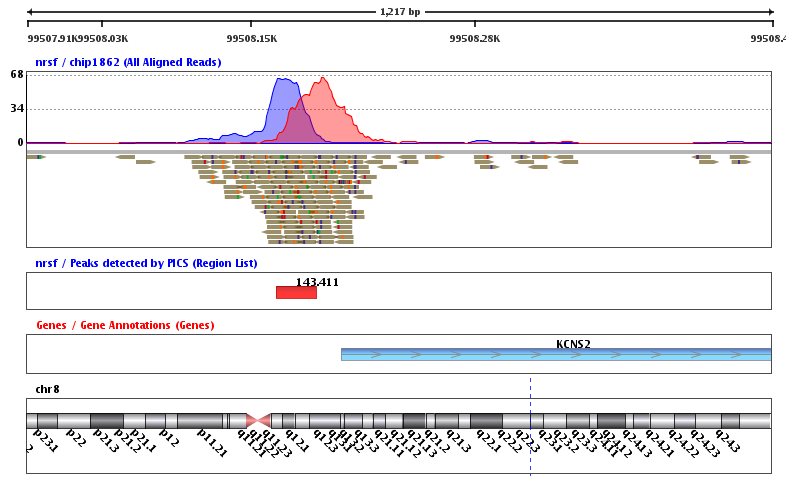
CHIP-seq测序的本质还是目标片段捕获测序,跟WES不同的是,它不是通过固定的芯片探针来固定的捕获基因组上面特定序列,而是根据你选择的IP不同,你细胞或者机体状态不同,捕获到的序列差异很大!而我们研究的重点,就是捕获到的差异。而我们对CHIP-seq测序数据寻找peaks的本质就是得到所有测序数据比对在全基因组之后在正个基因组上面的测序深度里面寻找比较突出的。比如对WES数据来说,各个外显子,或者外显子的5端到3端,理论上测序深度应该是一致的,都是50X~200X,画一个测序深度曲线,应该是近似于一条直线。对我们的CHIP-seq测序数据来说,在所捕获的区域上面,理论上测序深度是绝对不一样的,应该是近似于一个山峰。而那些覆盖度高的地方,山顶,就是我们的IP所结合的热点,也就是我们想要找的peaks,在IGV里面看到大致是下面这样:
可以看到测序的reads分布是绝对的不均匀的!我们通常说的CHIP-seq测序的IP,可以是各个组蛋白的各个修饰位点对应的抗体,或者是各种转录因子的抗体,等等
如何定义热点呢?通俗地讲,热点是这样一些位置,这些位置多次被测得的read所覆盖(我们测的是一个细胞群体,read出现次数多,说明该位置被TF结合的几率大)。那么,read数达到多少才叫多?这就要用到统计检验喽。假设TF在基因组上的分布是没有任何规律的,那么,测序得到的read在基因组上的分布也必然是随机的,某个碱基上覆盖的read的数目应该服从二项分布。
具体统计学原理直接看原创吧:http://www.plob.org/2014/05/08/7227.html
自学CHIP-seq分析第五讲~测序数据比对
比对本质是是很简单的了,各种mapping工具层出不穷,我们一般常用的就是BWA和bowtie了,我这里就挑选bowtie2吧,反正别人已经做好了各种工具效果差异的比较,我们直接用就好了,代码如下:
## step5 : alignment to hg19/ using bowtie2 to do alignment
## ~/biosoft/bowtie/bowtie2-2.2.9/bowtie2-build ~/biosoft/bowtie/hg19_index /hg19.fa ~/biosoft/bowtie/hg19_index/hg19
## cat >run_bowtie2.sh
ls *.fastq | while read id ;
do
echo $id
#~/biosoft/bowtie/bowtie2-2.2.9/bowtie2 -p 8 -x ~/biosoft/bowtie/hg19_index/hg19 -U $id -S ${id%%.*}.sam 2>${id%%.*}.align.log;
#samtools view -bhS -q 30 ${id%%.*}.sam > ${id%%.*}.bam ## -F 1548 https://broadinstitute.github.io/picard/explain-flags.html
# -F 0x4 remove the reads that didn't match
samtools sort ${id%%.*}.bam ${id%%.*}.sort ## prefix for the output
# samtools view -bhS a.sam | samtools sort -o - ./ > a.bam
samtools index ${id%%.*}.sorted.bam
done
这个索引~/biosoft/bowtie/hg19_index/hg19需要自己提取建立好,见前文
初步比对的sam文件到底该如何过滤,我查了很多文章都没有给出个子丑寅卯,各执一词,我也没办法给大家一个标准,反正我测试了好几种,看起来call peaks的差异不大,就是得不到文章给出的那些结果!!
一般来说,初步比对的sam文件只能选取unique mapping的结果,所以我用了#samtools view -bhS -q 30,但是结果并没什么改变,有人说是peak caller这些工具本身就会做这件事,所以取决于你下游分析所选择的工具。
给大家看比对的日志吧:
SRR1042593.fastq
16902907 reads; of these:
16902907 (100.00%) were unpaired; of these:
667998 (3.95%) aligned 0 times
12467095 (73.76%) aligned exactly 1 time
3767814 (22.29%) aligned >1 times
96.05% overall alignment rate
[samopen] SAM header is present: 93 sequences.
SRR1042594.fastq
60609833 reads; of these:
60609833 (100.00%) were unpaired; of these:
9165487 (15.12%) aligned 0 times
39360173 (64.94%) aligned exactly 1 time
12084173 (19.94%) aligned >1 times
84.88% overall alignment rate
[samopen] SAM header is present: 93 sequences.
SRR1042595.fastq
14603295 reads; of these:
14603295 (100.00%) were unpaired; of these:
918028 (6.29%) aligned 0 times
10403045 (71.24%) aligned exactly 1 time
3282222 (22.48%) aligned >1 times
93.71% overall alignment rate
[samopen] SAM header is present: 93 sequences.
SRR1042596.fastq
65911151 reads; of these:
65911151 (100.00%) were unpaired; of these:
10561790 (16.02%) aligned 0 times
42271498 (64.13%) aligned exactly 1 time
13077863 (19.84%) aligned >1 times
83.98% overall alignment rate
[samopen] SAM header is present: 93 sequences.
SRR1042597.fastq
22210858 reads; of these:
22210858 (100.00%) were unpaired; of these:
1779568 (8.01%) aligned 0 times
15815218 (71.20%) aligned exactly 1 time
4616072 (20.78%) aligned >1 times
91.99% overall alignment rate
[samopen] SAM header is present: 93 sequences.
SRR1042598.fastq
58068816 reads; of these:
58068816 (100.00%) were unpaired; of these:
8433671 (14.52%) aligned 0 times
37527468 (64.63%) aligned exactly 1 time
12107677 (20.85%) aligned >1 times
85.48% overall alignment rate
[samopen] SAM header is present: 93 sequences.
SRR1042599.fastq
24019489 reads; of these:
24019489 (100.00%) were unpaired; of these:
1411095 (5.87%) aligned 0 times
17528479 (72.98%) aligned exactly 1 time
5079915 (21.15%) aligned >1 times
94.13% overall alignment rate
[samopen] SAM header is present: 93 sequences.
SRR1042600.fastq
76361026 reads; of these:
76361026 (100.00%) were unpaired; of these:
8442054 (11.06%) aligned 0 times
50918615 (66.68%) aligned exactly 1 time
17000357 (22.26%) aligned >1 times
88.94% overall alignment rate
[samopen] SAM header is present: 93 sequences.
可以看到比对非常成功!!!我这里就不用表格的形式来展现了,毕竟我又不是给客户写报告,大家就将就着看吧。