而且要求八百字!~
好气啊
感觉像是小时候语文考试的作文,不到最后半小时,绝对是写不出来的。
今晚12点是deadline,只能靠它了!!!! Continue reading
LASIK手术,全称是“准分子激光原位角膜磨镶术”(laser in situ keratomileusis, LASIK),手术对象是人的角膜,就是人们常说的近视眼手术。 Continue reading
在这个博客建立之处,我就公布了自己建站的全流程:菜鸟建站教程三部曲
刚才发现了一个更好的方案:用 R blogdown 搭建个人博客或科研网站
他已经介绍的非常详细了,我就不多说了,大家直接去他博客看哈!
背景:
Diffuse intrinsic pontine glioma (DIPG) is a universally fatal pediatric cancer.
A histone-3 K27M mutation affects ∼80% of DIPGs and drives aberrant transcription.
早在2015年的83个药物对14个DIPG细胞系的筛选实验中,就发现哪怕是效果最好的multi-HDAC inhibitor panobinostat也会被某些DIPG细胞系产生耐药性。(http://www.biotrainee.com/thread-1599-1-1.html) Continue reading
我看到一篇nature medicine文章里面提到了这个对肿瘤细胞系进行药物筛查文章链接:https://www.nature.com/nm/journal/v21/n6/full/nm.3855.html
首先,肿瘤细胞系是:
We assembled a panel of 14 patient-derived DIPG cell cultures, created using neurosphere and adherent models and obtained from both biopsy and autopsy samples (Fig. 1a), representing the breadth of DIPG cell cultures available worldwide at the initiation of the study. Continue reading
这篇文章是Received 29 March 2013, Revised 9 August 2013, Accepted 4 October 2013, Available online 31 October 2013
同时还有17 MAY 2013一篇science文章提到了K27M mutant影响了PRC2活性。
we performed IHC for H3K27me3 in a large cohort of pHGGs with known H3F3A mutation status (n = 104).
Strikingly, all K27M mutant pHGGs (n = 21) showed a strong reduction of overall H3K27me3 levels.
这篇文章就这一个目的,抢这个热点。
Continue reading
这篇文章最重要的观点是甲基化用来给GBM分组,分成了6组,至于突变什么的,随便讲了讲,反正数据也不给下载。
We identified six epigenetic and biological GBM subgroups displaying distinct global DNA methylation patterns, which harbor unique hotspot mutations, DNA copy-number alterations, and transcriptomic patterns.
分组如下: Continue reading
To decipher the molecular pathogenesis of paediatric GBM, we undertook a comprehensive mutation analysis in protein-coding genes by performing whole-exome sequencing (WES) on 48 well-characterized paediatric GBMs, including 6 patients for whom we had matched non-tumour (germline) DNA.
只有6个患者有NT配对样本,用来找somatic的mutation,结果发现其中4个患者就有H3F3A的突变,但是H3F3A本身是非常保守的,所以这个现象值得研究。
to our knowledge no human disorders have specifically been associated with mutations in histones, including H3.3
所以才扩大了WES测序样本数量。 Continue reading
最近在写一篇很有趣的文章,一张图说清楚wgs,wes,rna-seq,chip-seq的异同点!
需要用到一些测试数据,我准备拿17号染色体的40437407-40486397这约48Kb碱基区域来举例子,就需要把这个区域的bam提取出来。
我分别找了以前处理的wgs,wes,rna-seq,chip-seq公共数据,原始bam非常大,尤其是WGS的,45G的bam文件,所以只能抽取17号染色体的40437407-40486397这约48Kb碱基区域,以前我做mpileup或者其它都是用的-r 参数,所以我想当然的使用下面的代码: Continue reading
首先声明,我不会WGCNA分析,只是大概知道它会对大量样本(>8或者15)的表达矩阵进行统计学分析,然后把表达矩阵的基因找到一下基因集合,有一些基因集合大概是非常有意义的!
因为有朋友一直好奇,我是如何学习新的知识的,所以就趁这个机会,录制了3个视频,只是我的一个学习过程而已。感兴趣可以去链接:http://pan.baidu.com/s/1jIgBTzw 密码:yh42下载,但是最后一个视频录制过程中被打断了,所以我只好重新写了个文字版的,来补充解释一下。(如果你看视频,请先看那个必看!)
学习一个新的概念,新的分析方法,我首先是谷歌了一下这个关键词,找到两个非常赞的链接!
就是拿到表达矩阵,根据MAD来挑选top5000个基因的表达矩阵,然后用WGCNA的包构建共表达网络,检测每一个module是什么,有什么特性。接着把这些module跟个体结合起来。
我作为老一辈的生信工程师,所以喜欢perl一点,排斥python,其实呢,我也稍微看过一些python的语法,个人认为R和python几乎是一模一样的。R的特点就是内置了大量的函数,基本上你认识的英文单词都可以是一个函数,即使不是,你也可以自定义为函数。搞清楚了函数和变量,就可以看懂大部分的R代码了。
下面是生信菜鸟团QQ群管理员赵云对这3种编程语言的心得体会!

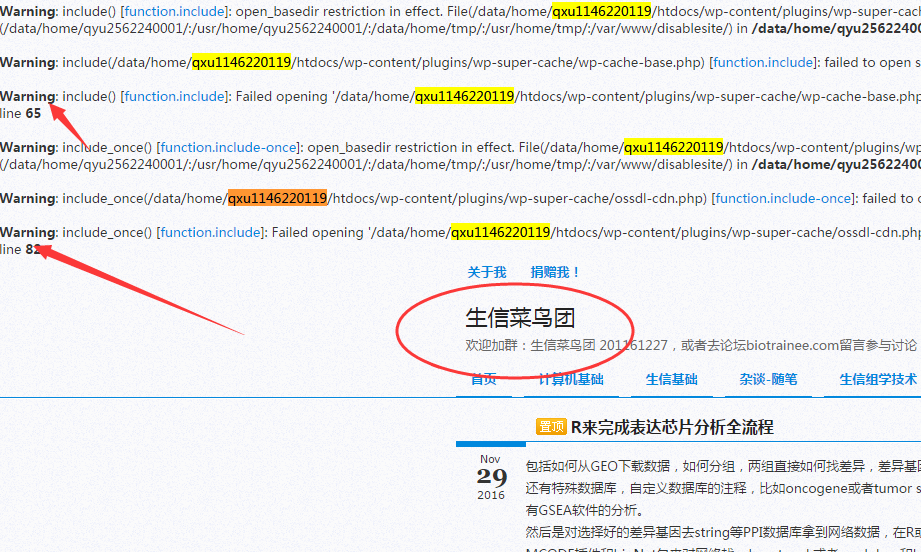
已经有非常多的小伙伴跟我反映了直接拷贝我的代码无法运行的问题,其实报错的原因很简单,就是中英文标点的bug而已。所以我给大家的理由是不用那么懒,拷贝我的代码,我就是希望你们能手动敲打每一个命令,来熟练记忆使用。
其实,我没那么好心,我就是懒而已。因为这个博客是host在阿里云的免费服务器上面的,各种IP密码我懒得去记忆,就差不多忘记了。当初弄好了我就懒得管了,正好现在博客免费时期快到了,也就想把这个问题解决掉。
<?php remove_filter('the_content','wptexturize'); ?> 正好看看这个标点符号被改变了吗?
好像还不错,以后大家就可以直接拷贝我的代码去运行啦!
下面是我登陆了ftp,发现以前用rmarkdown写的几个教程,感兴趣的小伙伴可以随便看看!
很多时候,我们都要选取unique mapped的reads,尤其是在RNA-seq和CHIP-seq的时候,但是如何保留,各种教程都不一致,我稍微总结了一下,是因为使用的比对工具不一样导致的!但是主要都反应在sam文件的一系列tag里面~
首先对bwa来说,如果它遇到一个reads可以比对到参考基因在的多个序列,只会随机的选取一个位置来输出到sam文件,但是会加上一个tag是XS:I:<N>来告诉我们第二好的比对情况的比对得分是多少,bowtie也是一样。但是它们都有参数来决定是否只对每个reads输出一条信息,还是输出全部的信息,在bwa是-a的参数,在bowtie里面是-m参数。
但是bowtie2里面取消了这个参数,它们都必须用XS:I:<N>这个tag来挑选unique mapped的reads
但是如果是用hisat来比对的话,决定是否是唯一比对的却是NH这个tag信息。默认情况下一条reads可以输出多条比对结果。
我想起了再补充吧,其实应该找几个例子用IGV看看,就明白了,可是我暂时没有时间了,只是觉得这个很重要,就提一下。
我对H3F3A这个基因做了两个突变的cellline,分别是G34V和K27M,现在知道这个基因在hg38上面的坐标是:
Genomic Location for H3F3A Gene
Chromosome: 1
Start:226,061,851 bp from pter End:226,072,002 bp from pter
Size:10,152 bases Orientation:Plus strand
然后我用samtools结合bcftools把该基因区域的snp位点call出来:
samtools mpileup -r chr1:226061851-226072001 -t "DP4" -ugf ~/reference/genome/hg38/hg38.fa *sorted.bam | bcftools call -vmO z -o H3F3A.vcf.gz