很多文献都提到外显子组的序列仅占全基因组序列的1%左右,但大多数与疾病相关的变异位于外显子区。通过外显子组测序可鉴定约8万个变异,全基因组测序可鉴定300万个变异,因此与全基因组测序相比,外显子组测序不仅费用较低,数据阐释也更为简单。外显子组测序技术以其经济有效的优势广泛应用于孟德尔遗传病、罕见综合征及复杂疾病的研究,并于2010年被Science杂志评为十大突破之一。所以我一直想验证一下外显子到底占基因组什么样的百分比,是哪些位点。
首先我们拿到外显子记录和基因信息,下面是通过R来从bioconductor中心提取数据
library("TxDb.Hsapiens.UCSC.hg19.knownGene")
txdb <- TxDb.Hsapiens.UCSC.hg19.knownGene
txdb
exon_txdb=exons(txdb)
genes_txdb=genes(txdb)
tmp=as.data.frame(exon_txdb)
write.table(tmp,'exon.pos',row.names=F)
tmp=as.data.frame(genes_txdb)
write.table(tmp,'gene.pos',row.names=F)
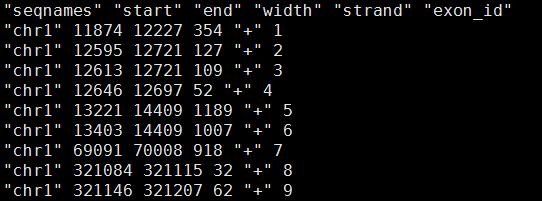
首先我们看看我刚才从TxDb.Hsapiens.UCSC.hg19.knownGene包里面提取的外显子记录文件exon.pos
我简单用脚本统计了一下:perl -alne '{$tmp+=$F[3]} END{print $tmp}' exon.pos
共289970个外显子记录,长度共98759283bp,也就是98.5Mb的区域都是外显子,感觉有点不大对,毕竟真正的外显子也就三十多M的,所以必须有些外显子是并列关系,也就是有的外显子包含着另外一些外显子,然后我写脚本检查了一下,的确太多了,这样的重复!
也可以去NCBI里面拿到 consensus coding sequence (CCDS)记录:ftp://ftp.ncbi.nlm.nih.gov/pub/CCDS/current_human/
那么我再看看NCBI里面拿到 consensus coding sequence (CCDS)记录CCDS.20150512.txt文件。
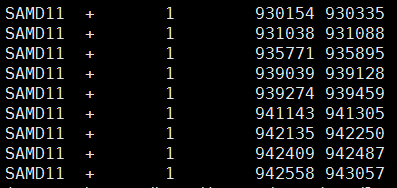
共33140行,一个基因一行,所以需要格式化成外显子文件,简单看了一下文件的列,很容易格式化。每一行的信息如下
1 NC_000001.11 SAMD11 148398 CCDS2.2 Public + 925941 944152 [925941-926012, 930154-930335, 931038-931088, 935771-935895, 939039-939128, 939274-939459, 941143-941305, 942135-942250, 942409-942487, 942558-943057, 943252-943376, 943697-943807, 943907-944152] Identical
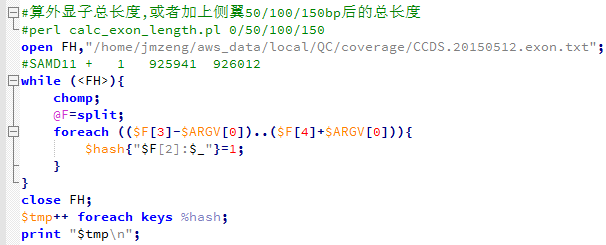
用下面这个脚本格式化
格式化后的文件如下
外显子记录增加到了346493个,我再用单行命令计算长度,是56321786bp,这次只有56M了,还是有点大,所以应该是还有重复!
所以,我写了一个脚本来精确计算外显子的总长度,不能那样马马虎虎的用单行命令了。
这次才是34729283bp,也就是约35M,根很多文献里面说的都一样!
同理,我统计了一下外显子的侧翼长度
54692160 外显子加上前后50bp
73066288 外显子加上前后100bp
90362533 外显子加上前后150bp
而hg19版本基因组里面有着entrez gene ID号的基因是23056个基因,所以我接下来探究一下这些基因的信息!
我们首先看看基因与基因之间的交叉情况
其中有12454266bp的位点,是多个外显子共有的,可能是一个基因的多个外显子,或者是不同基因的外显子
我们主要看看不同基因的交叉情况
不同基因交叉情况共516种,共涉及498种基因!
ZNF341 NFS1
ZNF397 ZSCAN30
ZNF428 SRRM5
ZNF436-AS1 RPL11
ZNF578 ZNF137P
ZNF619 ZNF621
ZNF816 ZNF816-ZNF321P
ZSCAN26 ZSCAN31
ZSCAN5A ZNF542P
ZZEF1 ANKFY1
我随便在数据库里面搜索一下验证,发现这些基因的确是交叉重叠的