Tag Archives: bioconductor
用R的bioconductor里面的stringDB包来做PPI分析
PPI本质上是根据一系列感兴趣的蛋白质或者基因(可以是几百个甚至上千个)来去PPI数据库里面找到跟这系列蛋白质或者基因的相互作用关系!
R一大利器之对象的操作函数查询
对于生物出身的部分生物信息学工程师来说,很多计算机概念让人很头疼,尤其是计算机语言里面的高级对象。我以前学编程的时候,给我一个变量,一个数据,一个hash,我就心满意足了,可以解决大部分我数据处理问题,可事情远比想象之中复杂。因为很多高手喜欢用封装,代码复用,喜欢用高级对象。在R的bioconductor里面尤其是如此,经常会遇到各种包装好的S3,S4对象,看过说明书,倒是知道一些对象里面有什么,可以去如何处理那些对象,提取我们想要的信息,比如我就写过一系列的帖子:
用lumi包来处理illumina的bead系列表达芯片
表达芯片大家最熟悉的当然是affymetrix系列芯片啦,而且分析套路很简单,直接用R的affy包,就可以把cel文件经过RMA或者MAS5方法得到表达矩阵。illumina出厂的芯片略微有点不一样,它的原始数据有3个层级,一般拿到的是Processed data (示例), 当仍然需要一系列的统计学方法才能提取到表达矩阵。我比较喜欢用bioconductor,所以下面讲一讲如何用lumi包来处理这个芯片数据!
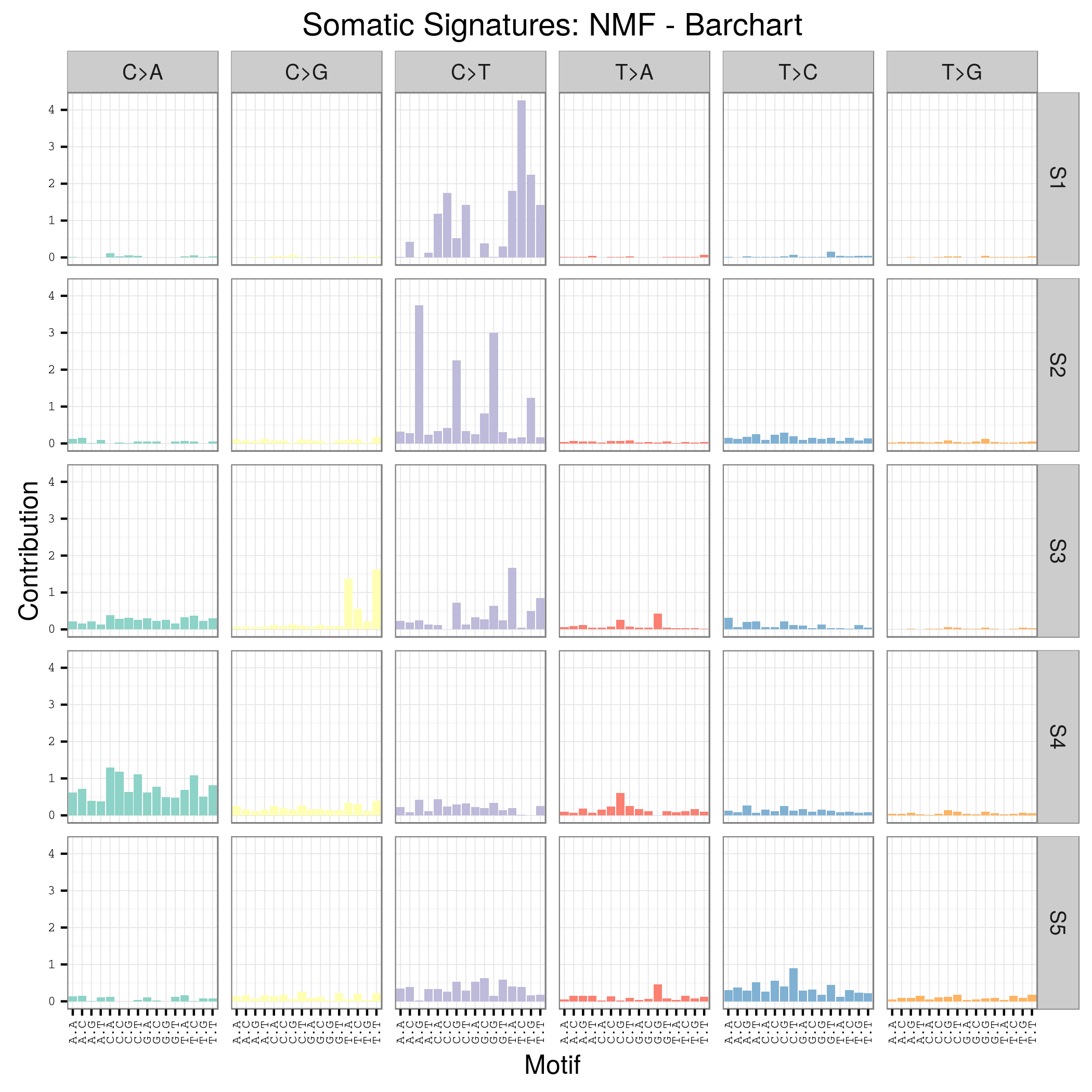
用SomaticSignatures包来解析maf突变数据获得mutation signature
mutation signature这个概念提出来还不久,我看了看文献,最早见于2013年的一篇nature文章,主要是用来描述癌症患者的somatic mutation情况的。
首先要自己分析癌症样本数据,拿到somatic mutation,TCGA计划发展到现在已经有非常多的somatic mutation结果啦,大家可以自行选择感兴趣的癌症数据拿来研究,解析一下mutation signature 。
我这里给大家推荐一个工具,是R语言的Bioconductor系列包中的一个,SomaticSignatures
其实它的说明书写的非常详细了已经,如果你理解了mutation signature的概念,很容易用那个包,其实你自己写一个脚本也是非常任意的,就是根据mutation的位置在基因组中找到它的前后一个碱基,然后组成三碱基突变模式,最后统计一下那96种突变模式的分布状况!
我这里简单讲一讲这个包如何用吧!
首先下载并加载几个必须的包:
然后根据突变数据做好一个GRanges对象,这个可以看我以前的博客
用samr包对芯片数据做差异分析
本来搞差异分析的工具和包就一大堆了,而且limma那个包已经非常完善了,我是不准备再讲这个的,正好有个同学问了一下这个包,我就随手测试了一下,顺便看看它跟limma有什么差异没有!手痒了就记录了测试流程!
学习一个包其实非常简单,就是找到包的官网看看说明书即可!说明书链接
用oligo包来读取affymetix的基因表达芯片数据-CEL格式数据
前面讲到affy处理的芯片平台是有限的,一般是hgu 95系列和133系列,[HuGene-1_1-st] Affymetrix Human Gene 1.1 ST Array这个平台虽然也是affymetrix公司的,但是affy包就无法处理 了,这时候就需要oligo包了!
oligo包是R语言的bioconductor系列包的一个,就一个功能,读取affymetix的基因表达芯片数据-CEL格式数据,处理成表达矩阵!!!
用affy包读取affymetix的基因表达芯片数据-CEL格式数据
Affymetrix的探针(proble)一般是长为25碱基的寡聚核苷酸;探针总是以perfect match 和mismatch成对出现,其信号值称为PM和MM,成对的perfect match 和mismatch有一个共同的affyID。
CEL文件:信号值和定位信息。
CDF文件:探针对在芯片上的定位信息
affy包是R语言的bioconductor系列包的一个,就一个功能,读取affymetix的基因表达芯片数据-CEL格式数据,处理成表达矩阵!!!
R包精讲第四篇:4种R包安装方式
请先看:R包精讲第一篇:如何查看你已经安装了和可以安装哪些R包?
第一种方式,当然是R自带的函数直接安装包了,这个是最简单的,而且不需要考虑各种包之间的依赖关系。
对普通的R包,直接install.packages()即可,一般下载不了都是包的名字打错了,或者是R的版本不够,如果下载了安装不了,一般是依赖包没弄好,或者你的电脑缺少一些库文件,如果实在是找不到或者下载慢,一般就用repos=来切换一些镜像。
> install.packages("ape") ##直接输入包名字即可 Installing package into ‘C:/Users/jmzeng/Documents/R/win-library/3.1’ (as ‘lib’ is unspecified) ##一般不指定lib,除非你明确知道你的lib是在哪里 trying URL 'http://mirror.bjtu.edu.cn/cran/bin/windows/contrib/3.1/ape_3.4.zip' Content type 'application/zip' length 1418322 bytes (1.4 Mb) opened URL ## 根据你选择的镜像,程序会自动拼接好下载链接url downloaded 1.4 Mb package ‘ape’ successfully unpacked and MD5 sums checked ##表明你已经安装好包啦 The downloaded binary packages are in ##程序自动下载的原始文件一般放在临时目录,会自动删除 C:\Users\jmzeng\AppData\Local\Temp\Rtmpy0OivY\downloaded_packages |
|
|
对于bioconductor的包,我们一般是
source("http://bioconductor.org/biocLite.R") ##安装BiocInstaller
#options(BioC_mirror=”http://mirrors.ustc.edu.cn/bioc/“) 如果需要切换镜像
biocLite("ggbio")或者直接BiocInstaller::biocLite('ggbio') ## 前提是你已经安装好了BiocInstaller
某些时候你还需要卸载remove.packages("BiocInstaller") 然后安装新的
第二种方式,是直接找到包的下载地址,需要进入包的主页
packageurl <- "http://cran.r-project.org/src/contrib/Archive/ggplot2/ggplot2_0.9.1.tar.gz"
packageurl <- "http://cran.r-project.org/src/contrib/Archive/gridExtra/gridExtra_0.9.1.tar.gz"
install.packages(packageurl, repos=NULL, type="source")
#packageurl <- "http://www.bioconductor.org/packages/2.11/bioc/src/contrib/ggbio_1.6.6.tar.gz"
#packageurl <- "http://cran.r-project.org/src/contrib/Archive/ggplot2/ggplot2_1.0.1.tar.gz"
install.packages(packageurl, repos=NULL, type="source")
这样安装的就不需要选择镜像了,也跨越了安装器的版本!
第三种是,先把包下载到本地,然后安装:
download.file("http://bioconductor.org/packages/release/bioc/src/contrib/BiocInstaller_1.20.1.tar.gz","BiocInstaller_1.20.1.tar.gz")
##也可以选择用浏览器下载这个包
install.packages("BiocInstaller_1.20.1.tar.gz", repos = NULL)
## 如果你用的RStudio这样的IDE,那么直接用鼠标就可以操作了
或者用choose.files()来手动交互的选择你把下载的源码BiocInstaller_1.20.1.tar.gz放到了哪里。
这种形式大部分安装都无法成功,因为R包之间的依赖性很强!
第四种是:命令行版本安装
如果是linux版本,命令行从网上自动下载包如下:
sudo su - -c \
"R -e \"install.packages('shiny', repos='https://cran.rstudio.com/')\""
如果是linux,命令行安装本地包,在shell的终端
sudo R CMD INSTALL package.tar.gz
window或者mac平台一般不推荐命令行格式,可视化那么舒心,何必自讨苦吃
R包精讲第三篇:如何切换镜像?
这个技巧很重要,一般来说,R语言自带的install.packages函数来安装一个包时,都是用的默认的镜像!
如果你是用的Rstudio这个IDE,你的默认镜像就是: https://cran.rstudio.com/
如果你直接用的R语言,那么就是:"http://cran.us.r-project.org" 但是一般你安装的时候会提醒你选择。
R包精讲第二篇:如何安装旧版本的包?
既然你点进来看,肯定是有需求咯! 一般来说,R语言自带的install.packages函数来安装一个包时,都是默认安装最新版的。 但是有些R包的开发者他会引用其它的一些R包,但是它用的是人家旧版本的功能,但他自己来不及更新或者疏忽了。 而我们又不得不用他的包,这时候就不得不卸载最新版包,转而安装旧版本包。
R包精讲第一篇:如何查看你已经安装了和可以安装哪些R包?
最近经常出现一个错误,类似于package ‘airway’ is not available (for R version 3.1.0)
就是某些包在R的仓库里面找不到,这个错误非常普遍,stackoverflow上面非常详细的解答:
在阅读这个答案的时候,我发现了一个非常有用的函数!available.packages()可以查看自己的机器可以安装哪些包!
用R语言的DESeq2包来对RNA-seq数据做差异分析
我以前写过DESeq,以及过时了:http://www.bio-info-trainee.com/867.html
正好准备筹集bioconductor中文社区,我写简单讲一下DESeq2这个包如何用!
ExpressionSet 对象简单讲解
这是我们bioconductor中文社区的一个简单测试
好像放在博客里面markdown的语法除了问题,欢迎直接去github查看
这个对象其实是对表达矩阵加上样本分组信息的一个封装,由biobase这个包引入。它是eSet这个对象的继承。
一个现成例子
下面是一个具体的例子,来源于CLL这个包,是用hgu95av2芯片测了22个样本
> library(CLL)
> data(sCLLex)
> sCLLex
ExpressionSet (storageMode: lockedEnvironment)
assayData: 12625 features, 22 samples ##表达矩阵
element names: exprs
protocolData: none
phenoData
sampleNames: CLL11.CEL CLL12.CEL ... CLL9.CEL (22 total)
varLabels: SampleID Disease ## 样本分组信息
varMetadata: labelDescription
featureData: none
experimentData: use 'experimentData(object)'
Annotation: hgu95av2
> exprMatrix=exprs(sCLLex)
> dim(exprMatrix)
[1] 12625 22
> meta=pData(sCLLex)
> table(meta$Disease)
progres. stable
14 8
>
根据上面的信息可以看出该芯片共12625个探针,这22个样本根据疾病状态分成两组,14vs8
这个数据对象就可以打包做很多包的分析输入数据。
对这个包的分析,重点就是 `exprs` 函数提取表达矩阵,`pData` 函数看看该对象的样本分组信息。
limma等包使用该对象作为输入数据
下面这个例子充分说明了
ExpressionSet对象的重要性
> library(limma)
> design=model.matrix(~factor(sCLLex$Disease))
> fit=lmFit(sCLLex,design)
> fit=eBayes(fit)
> options(digits = 4)
> topTable(fit,coef=2,adjust='BH')
logFC AveExpr t P.Value adj.P.Val B
39400_at 1.0285 5.621 5.836 8.341e-06 0.03344 3.234
36131_at -0.9888 9.954 -5.772 9.668e-06 0.03344 3.117
33791_at -1.8302 6.951 -5.736 1.049e-05 0.03344 3.052
1303_at 1.3836 4.463 5.732 1.060e-05 0.03344 3.044
36122_at -0.7801 7.260 -5.141 4.206e-05 0.10619 1.935
36939_at -2.5472 6.915 -5.038 5.362e-05 0.11283 1.737
41398_at 0.5187 7.602 4.879 7.824e-05 0.11520 1.428
32599_at 0.8544 5.746 4.859 8.207e-05 0.11520 1.389
36129_at 0.9161 8.209 4.859 8.212e-05 0.11520 1.389
37636_at -1.6868 5.697 -4.804 9.355e-05 0.11811 1.282
>
还有非常多的其它包会使用 ExpressionSet 对象,我就不一一介绍了。
自己构造 ExpressionSet 对象
根据上面的讲解,我们知道了在这个对象其实很简单,就是对表达矩阵加上样本分组信息的一个封装。 所以我们就用上面得到的exprMatrix和meta来构建一个ExpressionSet对象,biobase包里面提供了详细的说明,建议大家仔细看官方手册
metadata <- data.frame(labelDescription=c('SampleID', 'Disease'),
row.names=c('SampleID', 'Disease'))
phenoData <- new("AnnotatedDataFrame",data=meta,varMetadata=metadata)
myExpressionSet <- ExpressionSet(assayData=exprMatrix,
phenoData=phenoData,
annotation="hgu95av2")
> myExpressionSet
ExpressionSet (storageMode: lockedEnvironment)
assayData: 12625 features, 22 samples
element names: exprs
protocolData: none
phenoData
sampleNames: CLL11.CEL CLL12.CEL ... CLL9.CEL (22 total)
varLabels: SampleID Disease
varMetadata: labelDescription
featureData: none
experimentData: use 'experimentData(object)'
Annotation: hgu95av2
>
从上面的构造过程可以看出,重点就是表达矩阵加上样本分组信息
其它例子
ALL包的数据自带 ExpressionSet 对象
library(ALL)
data(ALL)
ALL
ExpressionSet (storageMode: lockedEnvironment)
assayData: 12625 features, 128 samples
element names: exprs
protocolData: none
phenoData
sampleNames: 01005 01010 … LAL4 (128 total)
varLabels: cod diagnosis … date last seen (21 total)
varMetadata: labelDescription
featureData: none
experimentData: use ‘experimentData(object)’
pubMedIds: 14684422 16243790
Annotation: hgu95av2
这个数据非常出名,很多其它算法包都会拿这个数据来举例子,只有真正理解了ExpressionSet对象才能学会bioconductor系列包
用GEOquery包来下载得到 ExpressionSet 对象
gse1009=GEOquery::getGEO("GSE1009")
gse1009[[1]] ## 这就是ExpressionSet对象
我发现糗世界讲的要比我好:http://blog.qiubio.com:8080/archives/2957
在Biobase基础包中,ExpressionSet是非常重要的类,因为Bioconductor设计之初是为了对基因芯片数据进行分析,而ExpressionSet正是Bioconductor为基因表达数据格式所定制的标准。它是所有涉及基因表达量相关数据在Bioconductor中进行操作的基础数据类型,比如affyPLM, affy, oligo, limma, arrayMagic等等。所以当我们学习Bioconductor时,第一个任务就是了解并掌握ExpressionSet的一切。
ExpressionSet的组成:
- assayData: 一个matrix类型或者environment类型数据。用于保存表达数据值。
当它是一个matrix时,它的行表示不同的探针组(probe sets)(也是features,总之是一个无重复的索引值)的值,它的列表示不同的样品。如果有行号或者列号的话,那么行号必须与featureData及phenoData中的行号一致,列号就是样品名。当我们使用exprs()方法时,就是调取的这个assayData的matrix。
当它是一个enviroment时,它必须有两个变量,一个就是与上一段描述一致的matrix,另一个就是epxrs,而这个exprs会响应exprs()方法,返回表达值。 - 头文件:用于描述实验平台相关的数据,其中包括phenoData, featureData,protocolData以及annotation等等。其中
phenoData是一个存放样品信息的data.frame或者AnnotatedDataFrame类型的数据。如果有行号的话,其行号必须与assayData的列号一致(也就是样品名)。如果没有行号,则其行数必须与assayData的列数一致。
featureData是一个存放features的data.frame或者AnnotatedDataFrame类型的数据。它的行数必须与assayData的行数一致。如果有行号的话,那么它的行号必须和assayData的行号一致。
annotation是用于存放芯片类型的字符串,比如hgu95av2之类。
protocolData用于存放设备相当的数据。它是AnnotatedDataFrame类型。它的维度必须与assayData的维度一致。 - experimentData: 一个MIAME类型的数据,它用于保存和实验设计相关的资料,比如实验室名,发表的文章,等等。那么什么是MIAME类呢?MIAME是Minimum Information About a Microarray Experiment的首字母缩写,它包括以下一些属性(slots):
- name: 字符串,实验名称
- lab: 字符串,实验室名称
- contact: 字符串,联系方式
- title: 字符串,一句话描述实验的内容
- abstract: 字符串,实验摘要
- url: 字符串,实验相关的网址
- samples: list类,样品的信息
- hybridizations: list类,杂交的信息
- normControls: list类,对照信息,比如一些持家基因(house keeping genes)
- preprocessing: list类,原始数据的预处理过程
- pubMedIds: 字符串,pubMed索引号
- others: list类,其它相关的信息
有了这些,所有实验相关的信息基本全备。
ExpressionSet继承了eSet类,属性基本和eSet保持一致。
那么,对于一个ExpressionSet,哪些属性是必须的?哪些有可能缺失呢?很显然,assayData是必须的,其它的可能会缺失,但是不能都缺失,因为那样的话就无法完成数据分析的工作。
对于ExpressionSet最重要的操作就是如何取出子集了。有时候在进行质量分析之后,我们对其中一些样品的数据不满意,想从已经实例化的ExpressionSet中抽取掉,或者我们希望对样品进行分组,都需要使用到Subset的概念。那么如何抽取子集呢?
我们可以象操作矩阵那样对其进行子集操作:vv <- exampleSet[1:5, 1:3]
使用它的一些属性来对其进行子集操作:males <- exampleSet[, exampleSet$gender == "Male"];
芯片探针注释基因ID或者symbol,并对每个基因挑选最大表达量探针
在R里面实现这个功能其实非常简单,难的是很多packages经常会出现安装问题,更有的人压根不看芯片平台是什么,芯片对应的package是什么,就开始到处发问,自学能力实在是堪忧!
我前面有写目前所有bioconductor支持的芯片平台对应关系:通过bioconductor包来获取所有的芯片探针与gene的对应关系
但那其实是一个很笨的办法,得到所有的各式各样的探针ID与基因的对应关系,以为它绕路了,正常情况只需要在GEO里面找到芯片对应基因关系即可,没必要下载那么多package的,但是这样做的好处也是很明显的, 对很多初学者来说,如果package能解决的话,就省心很多,比如下面这个转换关系:
suppressPackageStartupMessages(library(CLL))## 这个package自带了一个数据,是我们需要用的data(sCLLex) ## 这个数据里面有24个样本,分成两组,可以直接拿来测试差异基因分析library(hgu95av2.db) ## 一定要搞清楚自己的芯片是什么数据包exprSet=exprs(sCLLex) ##得到表达数据矩阵,但是矩阵的行名,是探针ID,无法理解,需要转换##首先你取出所有的探针ID,#这里可以用三种方法来得到symbol,或者得到entrezID也可以probeset=rownames(exprSet)Symbol=as.character(as.list(hgu95av2SYMBOL[probeset]))#annotate包提供 getSYMBOL( probeset ,"hgu95av2" )#还可以用lookUp函数 lookUp( probeset , "hgu95av2", "SYMBOL")#这些只是技巧而已啦a=cbind.data.frame(Symbol,exprSet)## 下面这个函数是对每个基因挑选最大表达量探针rmDupID <-function(a=matrix(c(1,1:5,2,2:6,2,3:7),ncol=6)){exprSet=a[,-1]rowMeans=apply(exprSet,1,function(x) mean(as.numeric(x),na.rm=T))a=a[order(rowMeans,decreasing=T),]exprSet=a[!duplicated(a[,1]),]#exprSet=apply(exprSet,2,as.numeric)exprSet=exprSet[!is.na(exprSet[,1]),]rownames(exprSet)=exprSet[,1]exprSet=exprSet[,-1]return(exprSet)}exprSet=rmDupID(a)
bioconductor中文社区招募站长
我已经构建好bioconductor中文社区的雏形,大家可以进去看看!
写在前面
突然发现我的bioconductor.cn这个域名都快要过期了!
哈哈,才想起一年前的计划到现在还没开始实施,实在不像我的风格,可能是水平到了一定程度吧,很多初级工作不像以前那样事无巨细的把关了。正好,借这个机会找几个朋友帮我一起完成这个bioconductor中文社区计划!
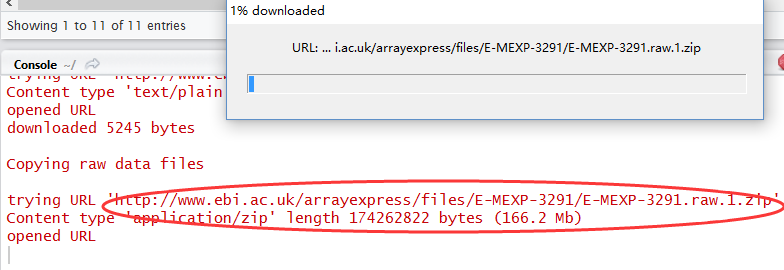
用R语言包从EBI的arrayexpress数据库里面下载芯片数据
rawset = ArrayExpress("E-MEXP-3291")
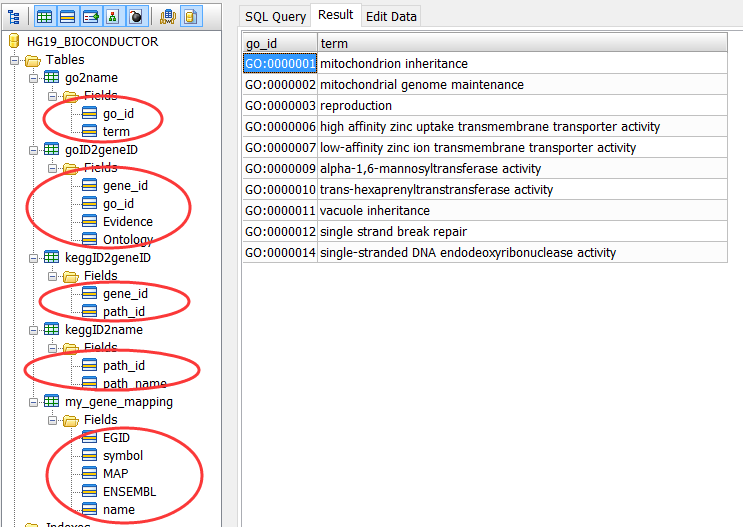
在R里面操作SQLite
library(org.Hs.eg.db )
把bioconductor的gene mapping信息上传到mysql数据库
R里面list对象和AnnDbBimap对象区别
AnnDbBimap对象是R里面的bioconductor系列包的基础对象,在探针数据里面会包装成ProbeAnnDbMap,跟go通路相关又是GOTermsAnnDbBimap对象。
但是它都是AnnDbBimap对象衍生过来的
主要存在于芯片系列的包和org系列的包,其实AnnDbBimap对象就是list对象的包装,比如下面这些例子:
ls("package:hgu133plus2.db")
| [1] "hgu133plus2" "hgu133plus2.db" "hgu133plus2_dbconn" |
| [4] "hgu133plus2_dbfile" "hgu133plus2_dbInfo" "hgu133plus2_dbschema" |
| [7] "hgu133plus2ACCNUM" "hgu133plus2ALIAS2PROBE" "hgu133plus2CHR" |
| [10] "hgu133plus2CHRLENGTHS" "hgu133plus2CHRLOC" "hgu133plus2CHRLOCEND" |
| [13] "hgu133plus2ENSEMBL" "hgu133plus2ENSEMBL2PROBE" "hgu133plus2ENTREZID" |
| [16] "hgu133plus2ENZYME" "hgu133plus2ENZYME2PROBE" "hgu133plus2GENENAME" |
| [19] "hgu133plus2GO" "hgu133plus2GO2ALLPROBES" "hgu133plus2GO2PROBE" |
| [22] "hgu133plus2MAP" "hgu133plus2MAPCOUNTS" "hgu133plus2OMIM" |
| [25] "hgu133plus2ORGANISM" "hgu133plus2ORGPKG" "hgu133plus2PATH" |
| [28] "hgu133plus2PATH2PROBE" "hgu133plus2PFAM" "hgu133plus2PMID" |
| [31] "hgu133plus2PMID2PROBE" "hgu133plus2PROSITE" "hgu133plus2REFSEQ" |
| [34] "hgu133plus2SYMBOL" "hgu133plus2UNIGENE" "hgu133plus2UNIPROT" |
那么我们可以随便挑选包中的一个数据分析一下
x <- hgu133plus2SYMBOL
xlist=as.list(x)
我们查看X对象,可知,它是object of class "ProbeAnnDbMap",而这个对象,就是常见的list对象,被包装了一下, 只有我们明白了它和list对象到底有什么区别,才算是真正搞懂了这一系列包
但是这个ProbeAnnDbMap对象,在GO等包里面会被包装成更复杂的对象-GOTermsAnnDbBimap,但是对他们的理解都大同小异。
我们先回顾一下在R语言里面的list的基础知识:
R 的 列表(list)是一个以对象的有序集合构成的对象。 列表中包含的对象又称为它的分量(components)。
分量可以是不同的模式或者类型, 如一个列表可以包括数值向量,逻辑向量,矩阵,复向量, 字符数组,函数等等
如果你会perl的话,可以把它理解成hash。
分量常常会被编号的(numbered),并且可以利用它来 访问分量
列表的分量可以被命名,这种情况下 可以通过名字访问。
特别要注意一下 Lst[[1]] 和 Lst[1] 的差别。 [[...]] 是用来选择单个 元素的操作符,而 [...] 是一个更为一般 的下标操作符。
因此前者得到的是列表 Lst 中的第一个对象, 并且含有分量名字的命名列表(named list)中分量的名字会被排除在外的。
后者得到的则是列表 Lst 中仅仅由第一个元素 构成的子列表。如果是命名列表, 分量名字会传给子列表的。
那么接下来我们就看看x和xlist的区别。
它们里面的数据都是affymetrix公司出品的人类的hgu133plus2芯片的探针ID与基因symbol的对应关系
如果我想拿到所有的探针ID,那么对于AnnDbBimap对象,需要用mappedkeys(x),对于普通的list对象,需要用names(xlist).
对于普通的list对象,如果我想看前几个元素,直接head就可以了,但是对于AnnDbBimap对象,数据被封装好了,需要先as.list,然后才能head
mapped_probes <- mappedkeys(x)
PID2=head(mapped_probes)
那么,如果我们想根据以下探针ID来查看它们在这些数据里面被对应着哪些基因symbol 呢?
> PID2 #这是一串探针ID,后面的操作都需要用的
[1] "1053_at" "117_at" "121_at" "1255_g_at" "1316_at" "1320_at"
如果是对于AnnDbBimap对象,我们可以用mget函数来操作,取多个探针的对应基因symbol
accnum <- mget(PID2, env=hgu133plus2ACCNUM);
gname <- mget(PID2, env=hgu133plus2GENENAME)
gsymbol <- mget(PID2, env=hgu133plus2SYMBOL)
mget函数返回的就是普通的list函数了,可以直接查看了。
如果是对于普通的list对象,我们想取多个探针的对应基因symbol也是非常简单的,xlist[PID2]即可。
那么我们不禁有问了,既然它们两个功能完全一样,何苦把它包装成一个对象了,我直接操作list对象不就好了,学那么多规矩干嘛?
所以,重点就来了
> length(mapped_probes)
[1] 42125
> length(names(xlist))
[1] 54675
看懂了吗?
但是,事实上用处也不大,你觉得下面这两个有区别吗?
SYMBOL <- AnnotationDbi::as.list(hgu133plus2SYMBOL)
SYMBOL <- SYMBOL[!is.na(SYMBOL)];
x <- hgu133plus2SYMBOL
mapped_probes <- mappedkeys(x)
SYMBOL <- AnnotationDbi::as.list(x[mapped_probes])
PS,在R里面创建一个list对象是非常简单的
The setNames() built-in function makes it easy to create a hash from given key and value lists.(Thanks to Nick K for the better suggestion.)
Usage: hh <- setNames(as.list(values), keys)
Example:
players <- c("bob", "tom", "tim", "tony", "tiny", "hubert", "herbert")
rankings <- c(0.2027, 0.2187, 0.0378, 0.3334, 0.0161, 0.0555, 0.1357)
league <- setNames(as.list(rankings), players)