Tag Archives: mysql
mysql的table居然有最大列限制
用GEMINI来探索vcf格式的突变数据
第一次听说这个软件,是一个香港朋友推荐的:http://davetang.org/muse/2016/01/13/getting-started-with-gemini/ 他写的很棒,但是我当初以为是一个类似于SQLite的数据库浏览模式,所以没在意。实际上,我现在仍然觉得这个软件没什么用!
软件官网有详细的介绍:https://gemini.readthedocs.io/en/latest/
而且提供丰富的教程:
We recommend that you follow these tutorials in order, as they introduce concepts that build upon one another.
- Introduction to GEMINI, basic variant querying and data exploration. html pdf
- Identifying de novo mutations underlying Mendelian disease html pdf
- Identifying autosomal recessive variants underlying Mendelian disease html pdf
- Identifying autosomal dominant variants underlying Mendelian disease html pdf
- Other GEMINI tools html pdf
软件本身并不提供注释,虽然它的功能的确包括注释,号称可以利用(ENCODE tracks, UCSC tracks, OMIM, dbSNP, KEGG, and HPRD.)对你的突变位点注释,比如你输入1 861389 . C T ,它告诉你这个突变发生在哪个基因,对蛋白改变如何?是否会产生某些疾病?
虽然它本身没有注释功能,但是它会调用snpEFF或者VEP进行注释,你需要自己先学习它们。
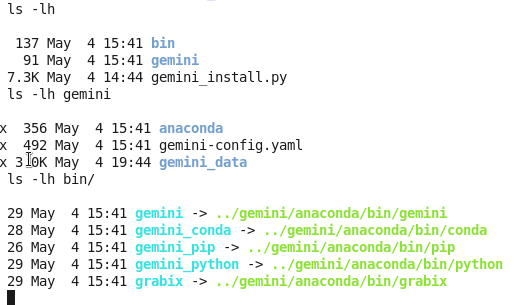
软件安装:
GEMINI是用python写的,有一个小脚本可以自动完成安装过程:
7.3K May 4 14:44 gemini_install.py
下载这个脚本,然后安装即可
wget https://github.com/arq5x/gemini/raw/master/gemini/scripts/gemini_install.py
python gemini_install.py $tools $data
PATH=$tools/bin:$data/anaconda/bin:$PATH
where $tools and $data are paths writable on your system.
我把$tools用的就是当前文件夹,$data也是当前文件夹下面的gemini文件夹。
这样就会在当前文件夹下面生成两个文件夹,bin是存储程序,gemini是存储数据用的,而且注意要把bin目录的全路径添加到环境变量!
输入数据:
我们可以直接下载软件作者提供的测试数据
首先是22号染色体的所有突变位点经过WEP注释的文件
然后是一个三口直接的突变ped格式数据
数据存放在亚马逊云,所有的教程pdf也在
http://s3.amazonaws.com/gemini-tutorials
如果是你自己的vcf文件,需要自己用VEP注释一下
运行命令:
结果解读:
产生是chr22.db就是一个数据库格式的文件,但是需要用gemini 来进行查询,个人认为,并没有多大意思!
你只要熟悉mySQL等SQL语言,完全可以自己来!
生信数据库的schema如何构建
大家分析生物信息学数据的时候必不可少的步骤就是利用各种公共资源对自己的数据进行注释。
这时候可能会用到mysql,把一些公共数据库本地化,方便使用,但是数据的下载已经存储到mysql等数据库中间会有很多值得玩味的事情。
我这里给大家指出一个还算比较标准的参考,就是bioconductor官方制作的数据库设计代码。
bioconductor官方注释方面的包(主要是各种ID的转换,KEGG或者GO这样的功能注释,基因信息注释,转录本,外显子起始终止等等)
目前为止,bioconductor是3.3版本,共896个包
大部分包都是以sqlite的数据库标准发布,所以建表语句是一样的。
所有代码见:https://github.com/Bioconductor-mirror/AnnotationDbi/blob/release-3.2/inst/DBschemas
部分代码如下:
CREATE TABLE metadata (
name VARCHAR(80) PRIMARY KEY,
value VARCHAR(255)
);
CREATE TABLE go_ontology (
ontology VARCHAR(9) PRIMARY KEY, -- GO ontology (short label)
term_type VARCHAR(18) NOT NULL UNIQUE -- GO ontology (full label)
);
CREATE TABLE go_term (
_id INTEGER PRIMARY KEY,
go_id CHAR(10) NOT NULL UNIQUE, -- GO ID
term VARCHAR(255) NOT NULL, -- textual label for the GO term
ontology VARCHAR(9) NOT NULL, -- REFERENCES go_ontology
definition TEXT NULL, -- textual definition for the GO term
FOREIGN KEY (ontology) REFERENCES go_ontology (ontology)
);
CREATE TABLE sqlite_stat1(tbl,idx,stat);
CREATE TABLE go_obsolete (
go_id CHAR(10) PRIMARY KEY, -- GO ID
term VARCHAR(255) NOT NULL, -- textual label for the GO term
ontology VARCHAR(9) NOT NULL, -- REFERENCES go_ontology
definition TEXT NULL, -- textual definition for the GO term
FOREIGN KEY (ontology) REFERENCES go_ontology (ontology)
);
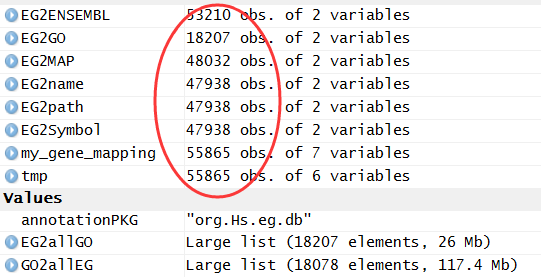
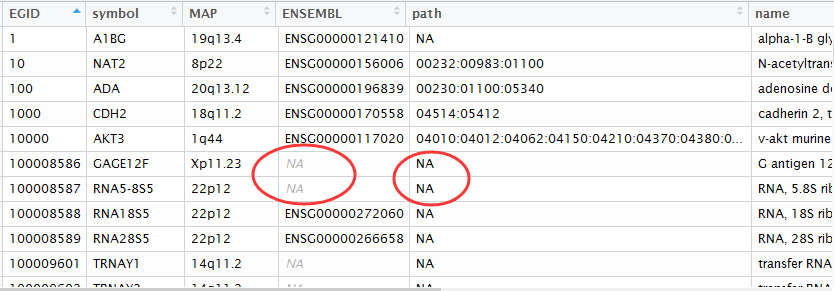
把bioconductor的gene mapping信息上传到mysql数据库
生物信息学学者学习mysql之路
来自于:https://www.biostars.org/p/474/#9095
for example, I would cite:
UCSC http://genome.ucsc.edu/FAQ/FAQdownloads#download29
ENSEMBL http://uswest.ensembl.org/info/data/mysql.html
GO http://www.geneontology.org/GO.database.shtml#mirrors
1000 Genomes: since June 16, 2011: http://www.1000genomes.org/public-ensembl-mysql-instance
mysql -h mysql-db.1000genomes.org -u anonymous -P 4272
Flybase has direct access to its postgres chado database.
http://flybase.org/forums/viewtopic.php?f=14&t=114
hostname: flybase.org port: 5432 username: flybase password: no password database name: flybase
e.g. psql -h flybase.org -U flybase flybase
mysql -h database.nencki-genomics.org -u public
mysql -h useastdb.ensembl.org -u anonymous -P 5306
是不是很简单呀,只有你认真的学习,其实这些应用的东西都还是蛮简单的。
ubuntu服务器解决方案第九讲-mysql和apache的安装
发现自己搞服务器遇到的困难还是蛮多的,所以记录了一下,给菜鸟们指个路。
ubuntu对生信菜鸟来说是最好用的linux服务器,没有之一,因为它有apt-get。
很多时候大家的服务器可能并不是想联网,只是想玩一下,或者只是因为生信的某些软件要求数据库,所以大家可能会单独安装mysql,或者想学习perl的CGI模块,需要apache。
ubuntu上安装mysql
非常简单只需要几条命令就可以完成。
1. sudo apt-get install mysql-server
2. sudo apt-get install mysql-client
3. sudo apt-get install libmysqlclient-dev
安装过程中会提示设置密码什么的,注意设置了不要忘了,安装完成之后可以使用如下命令来检查是否安装成功:
sudo netstat -tap | grep mysql
通过上述命令检查之后,如果看到有mysql 的socket处于 listen 状态则表示安装成功。
登陆mysql数据库可以通过如下命令:
mysql -u root -p
-u 表示选择登陆的用户名, -p 表示登陆的用户密码,上面命令输入之后会提示输入密码,此时输入密码就可以登录到mysql。
Ubuntu上安装Apache
在Ubuntu上安装Apache,有两种方式:1 使用开发包的打包服务,例如使用apt-get命令;2 从源码构建Apache。本文章将详细描述这两种不同的安装方式。
方法一:使用开发包的打包服务——apt-get
安装apache,在命令行终端中输入一下命令:
$ sudo apt-get install apache2
安装完成后,重启apache服务,在命令行终端中输入一下命令:
$ sudo /etc/init.d/apache2 restart
可能会出现的问题1: NameVirtualHost *:80 has no VirtualHosts,
出现上述问题的原因:定义了多个NameVirtualHost,故将/etc/apache2/ports.conf中的NameVirtualHost *:80注释掉即可。
可能会出现的问题2: Could not reliably determine the server's fully qualified domain name, using 127.0.1.1 for ServerName
原因:
根据提示,无法可靠的确定服务器的有效域名,使用127.0.1.1作为服务器域名。应此,在下面的测试中,应该使用127.0.1.1,而不是127.0.0.1!
解决:
$ vim /etc/apache2/httpd.conf,在文件中添加:
ServerName localhost:80,再次重启apache2,就可以使用127.0.0.1来访问web服务器啦!
测试:
在浏览器里输入http://localhost或者是http://127.0.0.1,如果看到了It works!,那就说明Apache就成功的安装了,Apache的默认安装,会在/var下建立一个名为www的目录,这个就是Web目录了,所有要能 过浏览器访问的Web文件都要放到这个目录里。