Tag Archives: KEGG
下载所有的酶的信息,并且解析好
之前我提到过kegg数据库里面的有些pathway下面没有对应任何基因,当时我还在奇怪,怎么会有这样的通路呢?
然后,我随机挑选了其中一条通路(hsa01000),进行查看,发现正好是所有的酶的信息。
好奇怪,不明白为什么kegg要列出所有的酶信息
http://www.genome.jp/kegg-bin/get_htext?hsa01000
下载htext格式的酶的信息
798K Dec 15 2015 hsa01000.keg
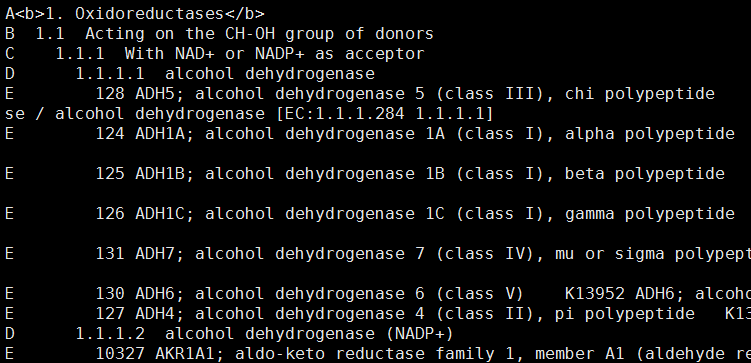
查看文件,发现也是层级非常清楚的结构,D已经是最底级别的酶了,而E对应的基因是属于该酶的。
简单统计一下,发现跟酶相关的基因有3688个,而最底级别的酶有5811个,应该会持续更新的
如果想做成kegg那样的基因与酶对应表格,也是非常简单的!
国际系统分类法按酶促反应类型,将酶分成六个大类:
1、氧化还原酶类(oxidoreductases) 催化底物进行氧化还原反应的酶类。包括电子或氢的转移以及分子氧参加的反应。常见的有脱氢酶、氧化酶、还原酶和过氧化物酶等。
2、转移酶类(transferases) 催化底物进行某些基团转移或交换的酶类,如甲基转移酶、氨基转移酶、转硫酶等。
3、水解酶类(hydrolases)催化底物进行水解反应的酶类。如淀粉酶、粮糖苷酶、蛋白酶等。
4、裂解酶类(lyases)或裂合酶类(synthases) 催化底物通过非水解途径移去一个基团形成双键或其逆反应的酶类,如脱水酶、脱羧酸酶、醛缩酶等。如果催化底物进行逆反应,使其中一底物失去双键,两底物间形成新的化学键,此时为裂合酶类。
5、异构酶类(isomerases)催化各种同分异构体、几何异构体或光学异构体间相互转换的酶类。如异构酶、消旋酶等。
6、连接酶类(ligases)或合成酶类(synthetases)催化两分子底物连接成一个分子化合物的酶类。
上述六大类酶用EC(enzyme commission)加1.2.3.4.5.6编号表示,再按酶所催化的化学键和参加反应的基团,将酶大类再进一步分成亚类和亚-亚类,最后为该酶在这亚-亚类中的排序。如α淀粉酶的国际系统分类纩号为:EC3.2.1.1
EC3——Hydrolases 水解酶类
EC3.2——Glycosylases 转葡糖基酶亚类
EC3.2.l——Glycosidases 糖苷酶亚亚类i.e.enzymes hydmlyzing O-and S-glycosyl compound即能水解O-和S-糖基化合物
EC3.2.1.1 Alpha-amylase, α-淀粉酶
值得注意的是,即使是同一名称和EC编号,但来自不同的物种或不同的组织和细胞的同一种酸,如来自动物胰脏、麦芽等和枯草杆菌BF7658的α-淀粉酶等,它们的一级结构或反应机制可解不同,它们虽然都能催化淀扮的水解反应,但有不同的活力和最适合反应条件。
可以按照酶在国际分类编号或其推荐名,从酶手册(Enzyme Handbook)、酶数据库中检索到该酶的结构、特性、活力测定和Km值等有用信息。著名的手册和数据库有:
手册:
1、Schomburg,M.Salzmann and D.Stephan:Enzyme Handbook 10 Volumes
2、美国Worthington Biochemical Corporation:Enzyme Manual
(http://www.worthington-biochem.com/index/manual.htm/)
数据库:
l、德国BRENDA:Enzyme Database(http://www.brenda wnzymes.org)
2、Swissprot:EXPASYENZYME Enzyme nomenclature database (http://www.expasy.org/enzyme/)
3、IntEnz:Integrated relational Enzyme database (http://www.ebi.ac.uk/mtenz)
下载最新版的KEGG信息,并且解析好
打开官网:http://www.genome.jp/kegg-bin/get_htext?hsa00001+3101
http://www.genome.jp/kegg-bin/get_htext#A1 (这个好像打不开)
可以在里面找到下载链接
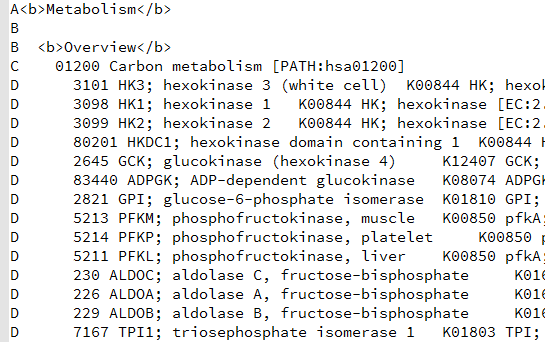
下载得到文本文件,可以看到里面的结构层次非常清楚,
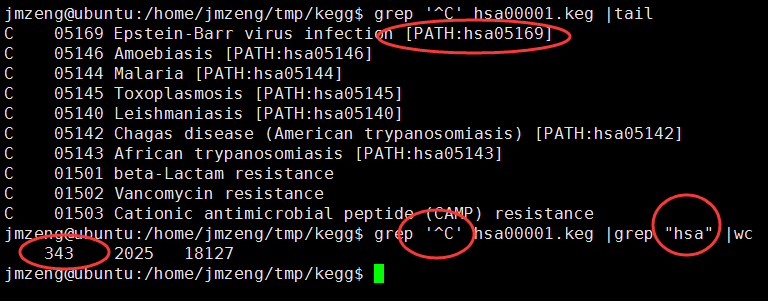
C开头的就是kegg的pathway的ID所在行,D开头的就是属于它的kegg的所有的基因
A,B是kegg的分类,总共是6个大类,42个小类
grep ^A hsa00001.keg
A<b>Metabolism</b>
A<b>Genetic Information Processing</b>
A<b>Environmental Information Processing</b>
A<b>Cellular Processes</b>
A<b>Organismal Systems</b>
A<b>Human Diseases</b>
也可以看到,到目前为止(2015年12月8日20:26:57),共有343个kegg的pathway信息啦
接下来我们就把这个信息解析一下:
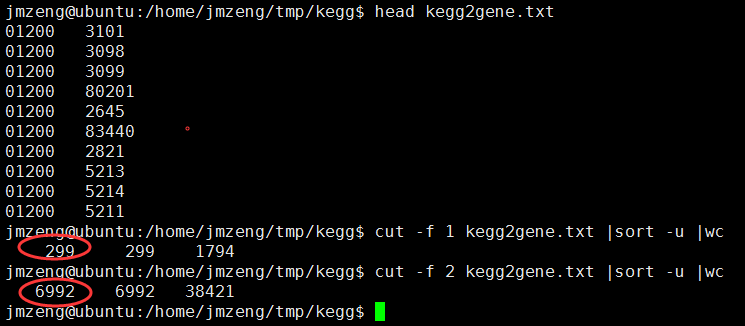
perl -alne '{if(/^C/){/PATH:hsa(\d+)/;$kegg=$1}else{print "$kegg\t$F[1]" if /^D/ and $kegg;}}' hsa00001.keg >kegg2gene.txt
这样就得到了
但是我发现了一个问题,有些通路竟然是没有基因的,我不是很明白为什么?
C 04030 G protein-coupled receptors [BR:hsa04030]
C 01020 Enzyme-linked receptors [BR:hsa01020]
C 04050 Cytokine receptors [BR:hsa04050]
C 03310 Nuclear receptors [BR:hsa03310]
C 04040 Ion channels [BR:hsa04040]
C 04031 GTP-binding proteins [BR:hsa04031]
那我们来看看kegg数据库更新的情况吧。
首先我们看org.Hs.eg.db这个R包里面自带的数据
Date for KEGG data: 2011-Mar15
org.Hs.egPATH has 5869 entrez genes and 229 pathways
2015年八月我用的时候是 6901 entrez genes and 295 pathways
现在是299个通路,6992个基因
所以这个更新其实很缓慢的,所以大家还在用DAVID这种网络工具做kegg的富集分析结果也差不大!
转录组-GO和KEGG富集的R包clusterProfiler
PS: 请不要在问我关于这个包的任何问题,直接联系Y叔,我就两年前用过一次而已,再也没有用过。
Y叔的包更新太频繁了,这个教程已经作废,请不要再照抄了,可以去我们论坛看新的教程:http://www.biotrainee.com/thread-1084-1-1.html
一:下载安装该R包
clusterProfiler是业界很出名的YGC写的R包,非常通俗易懂,也很好用,可以直接根据cuffdiff等找差异的软件找出的差异基因entrez ID号直接做好富集的所有内容; Continue reading