表达芯片大家最熟悉的当然是affymetrix系列芯片啦,而且分析套路很简单,直接用R的affy包,就可以把cel文件经过RMA或者MAS5方法得到表达矩阵。illumina出厂的芯片略微有点不一样,它的原始数据有3个层级,一般拿到的是Processed data (示例), 当仍然需要一系列的统计学方法才能提取到表达矩阵。我比较喜欢用bioconductor,所以下面讲一讲如何用lumi包来处理这个芯片数据!
Tag Archives: 芯片
illumina的bead 系列表达芯片扫盲
表达芯片大家最熟悉的当然是affymetrix系列芯片啦,而且分析套路很简单,直接用R的affy包,就可以把cel文件经过RMA或者MAS5方法得到表达矩阵。illumina出厂的芯片略微有点不一样,它的原始数据有3个层级,一般拿到的是Processed data (示例), 当仍然需要一系列的统计学方法才能提取到表达矩阵。接下来我们首先讲一讲illumina的bead 系列表达芯片基础知识吧: Continue reading
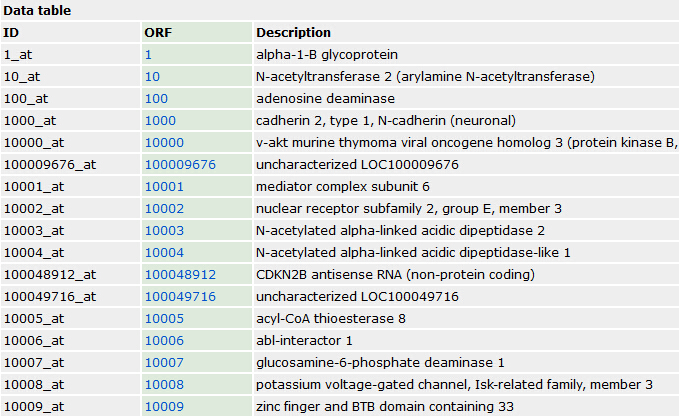
芯片探针注释基因ID或者symbol,并对每个基因挑选最大表达量探针
在R里面实现这个功能其实非常简单,难的是很多packages经常会出现安装问题,更有的人压根不看芯片平台是什么,芯片对应的package是什么,就开始到处发问,自学能力实在是堪忧!
我前面有写目前所有bioconductor支持的芯片平台对应关系:通过bioconductor包来获取所有的芯片探针与gene的对应关系
但那其实是一个很笨的办法,得到所有的各式各样的探针ID与基因的对应关系,以为它绕路了,正常情况只需要在GEO里面找到芯片对应基因关系即可,没必要下载那么多package的,但是这样做的好处也是很明显的, 对很多初学者来说,如果package能解决的话,就省心很多,比如下面这个转换关系:
suppressPackageStartupMessages(library(CLL))## 这个package自带了一个数据,是我们需要用的data(sCLLex) ## 这个数据里面有24个样本,分成两组,可以直接拿来测试差异基因分析library(hgu95av2.db) ## 一定要搞清楚自己的芯片是什么数据包exprSet=exprs(sCLLex) ##得到表达数据矩阵,但是矩阵的行名,是探针ID,无法理解,需要转换##首先你取出所有的探针ID,#这里可以用三种方法来得到symbol,或者得到entrezID也可以probeset=rownames(exprSet)Symbol=as.character(as.list(hgu95av2SYMBOL[probeset]))#annotate包提供 getSYMBOL( probeset ,"hgu95av2" )#还可以用lookUp函数 lookUp( probeset , "hgu95av2", "SYMBOL")#这些只是技巧而已啦a=cbind.data.frame(Symbol,exprSet)## 下面这个函数是对每个基因挑选最大表达量探针rmDupID <-function(a=matrix(c(1,1:5,2,2:6,2,3:7),ncol=6)){exprSet=a[,-1]rowMeans=apply(exprSet,1,function(x) mean(as.numeric(x),na.rm=T))a=a[order(rowMeans,decreasing=T),]exprSet=a[!duplicated(a[,1]),]#exprSet=apply(exprSet,2,as.numeric)exprSet=exprSet[!is.na(exprSet[,1]),]rownames(exprSet)=exprSet[,1]exprSet=exprSet[,-1]return(exprSet)}exprSet=rmDupID(a)
关于芯片平台GPL15308和GPL570
它们虽然被GEO数据标记了不同的ID号,但是其实都是一种芯片,都是昂飞公司的U133++2芯片,分析过芯片数据的人肯定不会陌生了
http://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GPL15308
事实上,这个平台应该是GPL570,但是被CCLE数据库给稍微变通了一下,就给了一个GPL15308的标签,平台主页也写的很清楚,它的探针ID是伪ID,其实就是entrez gene ID
| 本来这个芯片设计的是五万多个探针,最后只剩下了18926个基因 |
| This array is identical to GPL570 but the data were analyzed with a custom CDF Brainarray Version 15, hgu133plus2hsentrezg. |
拷贝数变异检测芯片介绍
通过与哈佛大学合办的Broad研究所合作,SNP6.0芯片在数据准确性和一致性方面达到了新的高度。相应推出的Genotyping Console用来处理SNP6.0芯片数据和全基因组遗传分析及质量控制。
产品特点:
1.涵盖超过1,800,000个遗传变异标志物:包括超过906,600个SNP和超过946,000个用于检测拷贝数变化(CNV,Copy Number Variation)的探针;
2.SNP和CNV两种探针高密度且均匀地分布在整个基因组,不仅可以用于SNP基因精确分型,还可用于拷贝数变异CNV的研究;
3.744,000个探针平均分配到整个基因组上,用来发现未知的拷贝数变异区域;
4.可用于Copy-neutral LOH/UPD检测,亲子鉴定,纯合性分析、血缘关系鉴定、遗传病或其它疾病的研究。
affymetix的基因表达芯片数据差异基因分析
我主要是看了一个差异分析的教程,讲的非常详细,全面,我先简单列出这个教程,然后再贴出我的代码
基因芯片(Affymetrix)分析2:芯片数据预处理
基因芯片(Affymetrix)分析3:获取差异表达基因
基因芯片(Affymetrix)分析4:GO和KEGG分析
基因芯片(Affymetrix)分析5:聚类分析
Bioconductor系列之hgu95av2.db
这个包主要都是芯片数据处理方面的应用,本来我是懒得看的,但是我无意中浏览了一下readme,发现它挺适合被作为数据库的典范来学习。
source("http://bioconductor.org/biocLite.R")
biocLite("org.Hs.eg.db")
biocLite("hgu95av2.db")
还是老办法安装了hgu95av2.db之后用ls可以查看这个包里面有36个映射数据,都是把芯片探针ID号转为其它36种主流ID号的映射而已。
library(hgu95av2.db)
> ls("package:hgu95av2.db")
[1] "hgu95av2" "hgu95av2ACCNUM" "hgu95av2ALIAS2PROBE"
[4] "hgu95av2CHR" "hgu95av2CHRLENGTHS" "hgu95av2CHRLOC"
[7] "hgu95av2CHRLOCEND" "hgu95av2.db" "hgu95av2_dbconn"
[10] "hgu95av2_dbfile" "hgu95av2_dbInfo" "hgu95av2_dbschema"
[13] "hgu95av2ENSEMBL" "hgu95av2ENSEMBL2PROBE" "hgu95av2ENTREZID"
[16] "hgu95av2ENZYME" "hgu95av2ENZYME2PROBE" "hgu95av2GENENAME"
[19] "hgu95av2GO" "hgu95av2GO2ALLPROBES" "hgu95av2GO2PROBE"
[22] "hgu95av2MAP" "hgu95av2MAPCOUNTS" "hgu95av2OMIM"
[25] "hgu95av2ORGANISM" "hgu95av2ORGPKG" "hgu95av2PATH"
[28] "hgu95av2PATH2PROBE" "hgu95av2PFAM" "hgu95av2PMID"
[31] "hgu95av2PMID2PROBE" "hgu95av2PROSITE" "hgu95av2REFSEQ"
[34] "hgu95av2SYMBOL" "hgu95av2UNIGENE" "hgu95av2UNIPROT"
但是用这个包自带的函数capture.output(hgu95av2())可以看到这些映射并没有囊括我们标准的hg19版本的2.3万个基因,也就说这个芯片设计的探针只有1.1万个左右。但它根据已有的org.Hs.eg.db来构建了它自己芯片独有的数据包,这样就显得更加正式规范化了,这启发我们研发人员在做一些市场产品的同时也可以把自己的研究成果通主流数据库数据形式结合起来,更易让他人接受。
> capture.output(hgu95av2())
[1] "Quality control information for hgu95av2:"
[2] ""
[3] ""
[4] "This package has the following mappings:"
[5] ""
[6] "hgu95av2ACCNUM has 12625 mapped keys (of 12625 keys)"
[7] "hgu95av2ALIAS2PROBE has 33755 mapped keys (of 103735 keys)"
[8] "hgu95av2CHR has 11540 mapped keys (of 12625 keys)"
[9] "hgu95av2CHRLENGTHS has 93 mapped keys (of 93 keys)"
[10] "hgu95av2CHRLOC has 11474 mapped keys (of 12625 keys)"
[11] "hgu95av2CHRLOCEND has 11474 mapped keys (of 12625 keys)"
[12] "hgu95av2ENSEMBL has 11460 mapped keys (of 12625 keys)"
[13] "hgu95av2ENSEMBL2PROBE has 9814 mapped keys (of 28553 keys)"
[14] "hgu95av2ENTREZID has 11543 mapped keys (of 12625 keys)"
[15] "hgu95av2ENZYME has 2125 mapped keys (of 12625 keys)"
[16] "hgu95av2ENZYME2PROBE has 786 mapped keys (of 975 keys)"
[17] "hgu95av2GENENAME has 11543 mapped keys (of 12625 keys)"
[18] "hgu95av2GO has 11245 mapped keys (of 12625 keys)"
[19] "hgu95av2GO2ALLPROBES has 17214 mapped keys (of 18826 keys)"
[20] "hgu95av2GO2PROBE has 12920 mapped keys (of 14714 keys)"
[21] "hgu95av2MAP has 11519 mapped keys (of 12625 keys)"
[22] "hgu95av2OMIM has 10541 mapped keys (of 12625 keys)"
[23] "hgu95av2PATH has 5374 mapped keys (of 12625 keys)"
[24] "hgu95av2PATH2PROBE has 228 mapped keys (of 229 keys)"
[25] "hgu95av2PMID has 11529 mapped keys (of 12625 keys)"
[26] "hgu95av2PMID2PROBE has 372382 mapped keys (of 432400 keys)"
[27] "hgu95av2REFSEQ has 11506 mapped keys (of 12625 keys)"
[28] "hgu95av2SYMBOL has 11543 mapped keys (of 12625 keys)"
[29] "hgu95av2UNIGENE has 11533 mapped keys (of 12625 keys)"
[30] "hgu95av2UNIPROT has 11315 mapped keys (of 12625 keys)"
[31] ""
[32] ""
[33] "Additional Information about this package:"
[34] ""
[35] "DB schema: HUMANCHIP_DB"
[36] "DB schema version: 2.1"
[37] "Organism: Homo sapiens"
[38] "Date for NCBI data: 2014-Sep19"
[39] "Date for GO data: 20140913"
[40] "Date for KEGG data: 2011-Mar15"
[41] "Date for Golden Path data: 2010-Mar22"
[42] "Date for Ensembl data: 2014-Aug6"
这个hgu95av2.db所加载的36个包都是一种特殊的对象,但是可以把它当做list来操作,是一种类似于hash的对应表格,其中keys是独特的,但是value可以有多个。
既然是类似于list,那我就简单讲几个小技巧来操作这些数据对象。所有的操作都要用as.list()函数来把数据展现出来
> as.list(hgu95av2ENZYME[1])
$`1000_at`
[1] "2.7.11.24"
可以看到这样就提取出来了hgu95av2ENZYME的第一个元素,key是`1000_at`,它所映射的value是 "2.7.11.24"这个酶。
同理对list取元素的三个操作在这里都可以用
> as.list(hgu95av2ENZYME['1000_at'])
$`1000_at`
[1] "2.7.11.24"
> as.list(hgu95av2ENZYME$'1000_at')
[[1]]
[1] "2.7.11.24"
然而这只是list的操作,我们还有一个函数专门对这个对象来取元素,那就是get函数,取多个元素用mget,类似于as.list(hgu95av2ENZYME[1:10])
用函数mget(probes_id,hgu95av2ENZYME)就可以根据自己的probes_id这个向量来选取数据了,当然也要用as.list()来展示出来。
值得一提的是这里有个非常重要的函数是revmap,可以把我们的key和value调换位置。
因为我们的数据对象里面的对应关系不是一对一,而是一对多,例如一个基因往往有多个go通路信息,例如这个就有十几个
as.list(hgu95av2GO$'1000_at')
$`GO:0000165`
$`GO:0000165`$GOID
[1] "GO:0000165"
$`GO:0000165`$Evidence
[1] "TAS"
$`GO:0000165`$Ontology
[1] "BP"
$`GO:0000186`
$`GO:0000186`$GOID
[1] "GO:0000186"
$`GO:0000186`$Evidence
[1] "TAS"
$`GO:0000186`$Ontology
[1] "BP"
一层层的数据结构非常清晰,但是数据太多,所以它给了一个toTable函数来格式化,就是把一对多的list压缩成表格。
> toTable(hgu95av2GO[1])
probe_id go_id Evidence Ontology
1 1000_at GO:0000165 TAS BP
2 1000_at GO:0000186 TAS BP
3 1000_at GO:0000187 TAS BP
4 1000_at GO:0002224 TAS BP
5 1000_at GO:0002755 TAS BP
6 1000_at GO:0002756 TAS BP
7 1000_at GO:0006360 TAS BP
------------------------------------------------------------
81 1000_at GO:0004707 NAS MF
82 1000_at GO:0001784 IEA MF
83 1000_at GO:0005524 IEA MF
这样就非常方便的查看啦。
当然对这个数据对象还有很多实用的操作。length(),Llength(),Rlength(),keys(),Lkeys(),Rkeys()等等,还有count.mappedkeys(),mappedLkeys(),还有个比较特殊的isNA来判断是否有些keys探针没有对应任何信息。
而且以上这些函数不仅可以用来获取数据对象的信息,还可以修改这个数据对象。
Lkeys(hgu95av2CHR) 可以查看这个对象有12625个探针。
而> Rkeys(hgu95av2CHR)
[1] "19" "12" "8" "14" "3" "2" "17" "16" "9" "X" "6" "1" "7" "10" "11"
[16] "22" "5" "18" "15" "Y" "20" "21" "4" "13" "MT" "Un"
可以看到这些探针分布在不同的染色体上面,如果我这时候给
x=hgu95av2CHR
Rkeys(x) = c("1","2")
toTable(x)可以看到这时候x只剩下1946个探针了,就是1,2号染色体上面的基因了。
然后可以一个个看这些函数的用法,其实就是SQL的增删改查的基本操作而已。
> length(x)
[1] 12625
> Llength(x)
[1] 12625
> Rlength(x)
[1] 2
> head(keys(x))
[1] "1000_at" "1001_at" "1002_f_at" "1003_s_at" "1004_at" "1005_at"
> head(Lkeys(x))
[1] "1000_at" "1001_at" "1002_f_at" "1003_s_at" "1004_at" "1005_at"
> head(Rkeys(x))
[1] "1" "2"
> count
count.fields count.mappedLkeys countOverlaps counts<-
count.links count.mappedRkeys countQueryHits countSubjectHits
count.mappedkeys countMatches counts
> count.mappedkeys(x)
[1] 1946
> count.mappedLkeys(x)
[1] 1946
> count.mappedRkeys(x)
[1] 2