首先要明白,需要下载什么?
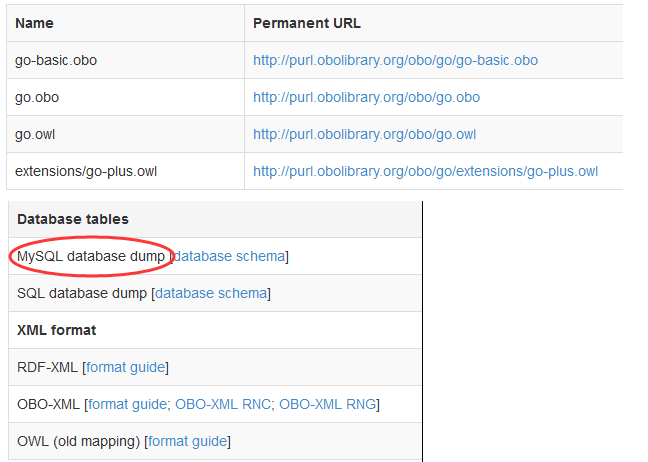
要下载四万多条GO记录的详细信息(http://purl.obolibrary.org/obo/go/go-basic.obo)
要下载GO与GO之间的关系(http://archive.geneontology.org/latest-termdb/go_daily-termdb-tables.tar.gz)
要下载GO与基因之间的对应关系!(物种)(ftp://ftp.ncbi.nlm.nih.gov/gene/DATA)
去官网!
http://geneontology.org/page/download-ontology
grep '\[Term\]' go-basic.obo |wc
43992 43992 307944
版本的区别!刚才我们下载的GO共有43992条,而以前的版本才38804条
GO与GO之间的关系
对应关系也在更新
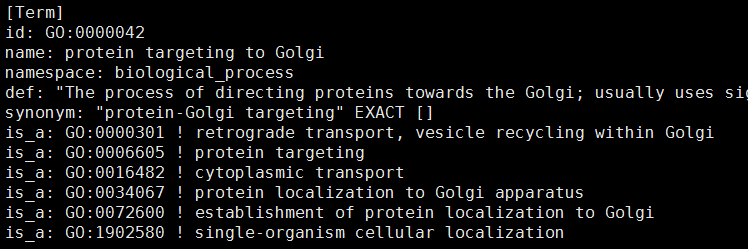
> as.list(GOBPPARENTS['GO:0000042'])
$`GO:0000042`
is_a is_a is_a is_a
"GO:0000301" "GO:0006605" "GO:0016482" "GO:0072600"
library(org.Hs.eg.db)
library(GO.db)
> tmp=toTable(org.Hs.egGO) ##这个只包括基因与最直接的go的对应关系
> dim(tmp)
[1] 213101 4
> tmp2=toTable(org.Hs.egGO2ALLEGS) #这个是所有的基因与go的对应关系
> dim(tmp2)
[1] 2218968 4
基因与GO的对应关系也在更新
grep '^9606' gene2go |wc -l ### ##这个只包括基因与最直接的go的对应关系
269063
ftp://ftp.informatics.jax.org/pub/reports/index.html#go
ftp://ftp.ncbi.nlm.nih.gov/gene/DATA
ftp://ftp.informatics.jax.org/pub/reports/index.html#go