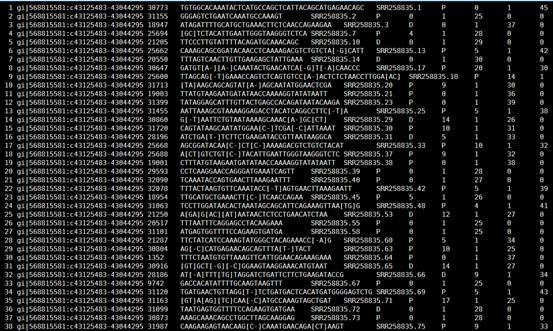
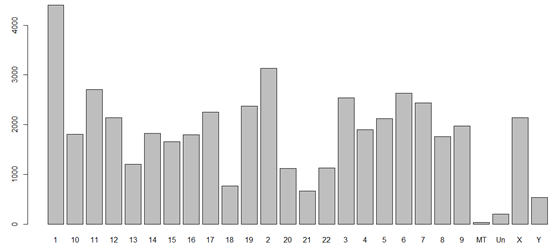
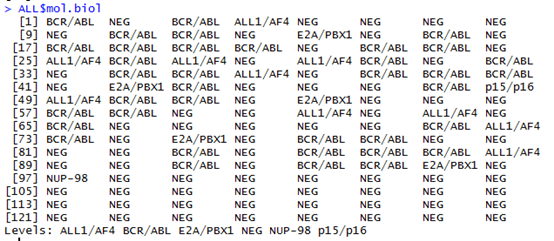
CHROM(chromosome):染色体
POS - position:参考基因组variant碱基位置,如果是INDEL(插入缺失),位置是INDEL的第一个碱基位置
ID - identifier: variant的ID。比如在dbSNP中有该SNP的id,则会在此行给出;若没有,则用’.'表示其为一个novel variant。
REF - reference base(s):参考碱基,染色体上面的碱基,必须是ATCGN中的一个,N表示不确定碱基
ALT - alternate base(s):与参考序列比较发生突变的碱基
QUAL - quality: Phred格式(Phred_scaled)的质量值,表 示在该位点存在variant的可能性;该值越高,则variant的可能性越大;计算方法:Phred值 = -10 * log (1-p) p为variant存在的概率; 通过计算公式可以看出值为10的表示错误概率为0.1,该位点为variant的概率为90%。
FILTER - _filter status: 使用上一个QUAL值来进行过滤的话,是不够的。GATK能使用其它的方法来进行过滤,过滤结果中通过则该值为”PASS”;若variant不可靠,则该项不为”PASS”或”.”。
INFO - additional information: 这一行是variant的详细信息,具体如下:
DP-read depth:样本在这个位置的reads覆盖度。是一些reads被过滤掉后的覆盖度。 DP4:高质量测序碱基,位于REF或者ALT前后
MQ:表示覆盖序列质量的均方值RMS Mapping Quality
FQ:phred值关于所有样本相似的可能性
AF1: AF(Allele Frequency) 表示Allele的频率,AF1为第一个ALT allele 发生频率的可能性评估
AC1:AC表示Allele(等位基因)的数目,AC1为对第一个ALT allele count的最大可能性评估
AN:AN(Allele Number) 表示Allele的总数目
IS:插入缺失或部分插入缺失的reads允许的最大数量
AC:AC(Allele Count) 表示该Allele的数目
G3:ML 评估基因型出现的频率
HWE:chi^2基于HWE的测试p值和G3
CLR:在受到或者不受限制的情况下基因型出现可能性log值
UGT:最可能不受限制的三种基因型结构
CGT:最可能受限制三种基因型的结构
PV4:四种P值得误差,分别是(strand、baseQ、mapQ、tail distance bias)
INDEL:表示该位置的变异是插入缺失
PC2:非参考等位基因的phred(变异的可能性)值在两个分组中大小不同
PCHI2:后加权chi^2,根据p值来测试两组样本之间的联系
QCHI2:Phred scaled PCHI2.
PR:置换产生的一个较小的PCHI2
QBD:Quality by Depth,测序深度对质量的影响
RPB:序列的误差位置(Read Position Bias)
MDV:样本中高质量非参考序列的最大数目
VDB:Variant Distance Bias,RNA序列中过滤人工拼接序列的变异误差范围
GT:样品的基因型(genotype)。两个数字中间用’/'分 开,这两个数字表示双倍体的sample的基因型。0 表示样品中有ref的allele; 1 表示样品中variant的allele; 2表示有第二个variant的allele。因此: 0/0 表示sample中该位点为纯合的,和ref一致; 0/1 表示sample中该位点为杂合的,有ref和variant两个基因型; 1/1 表示sample中该位点为纯合的,和variant一致。
GQ:基因型的质量值(Genotype Quality)。Phred格式(Phred_scaled)的质量值,表示在该位点该基因型存在的可能性;该值越高,则Genotype的可能性越 大;计算方法:Phred值 = -10 * log (1-p) p为基因型存在的概率。
GL:三种基因型(RR RA AA)出现的可能性,R表示参考碱基,A表示变异碱基
DV:高质量的非参考碱基
SP:phred的p值误差线
PL:指定的三种基因型的质量值(provieds the likelihoods of the given genotypes)。这三种指定的基因型为(0/0,0/1,1/1),这三种基因型的概率总和为1。和之前不一致,该值越大,表明为该种基因型的可能 性越小。 Phred值 = -10 * log (p) p为基因型存在的概率。
FORMAT 和 BC1-1-base.sorted.bam:这两行合起来提供了’ BC1-1-base′这个sample的基因型的信息。’ BC1-1-base′代表这该名称的样品,是由BAM文件中的@RG下的 SM 标签决定的。