这是发布在bioconductor平台上面的一个数据库文件,可以通过R里面下载安装并使用,非常方便。而且用的是数据库存储方式,所以搜索起来也是非常快速。
这个包里面有28个主流数据资料文件,这样我们可以用select函数根据我们自己的ID在这28个数据库里面随意转换自己想要的信息!!!
当然我本人是比较喜欢直接下载原文件,然后写脚本自己进行各种数据直接的转换。
首先我们加载这个数据包,可以看到这个数据包依赖于很多其它的包,如果是第一次安装。会耗时很长!
用这个函数,可以看到这个org.Hs.eg.db数据对象里面包含着各大主流数据库的数据,一般人都比较熟悉的entrez ID 和ensembl 数据库的ID。
keytypes(org.Hs.eg.db)
## [1] "ENTREZID" "PFAM" "IPI" "PROSITE"
## [5] "ACCNUM" "ALIAS" "ENZYME" "MAP"
## [9] "PATH" "PMID" "REFSEQ" "SYMBOL"
## [13] "UNIGENE" "ENSEMBL" "ENSEMBLPROT" "ENSEMBLTRANS"
## [17] "GENENAME" "UNIPROT" "GO" "EVIDENCE"
## [21] "ONTOLOGY" "GOALL" "EVIDENCEALL" "ONTOLOGYALL"
## [25] "OMIM" "UCSCKG"
然后,我们用select函数,就可以把任意公共数据库的数据进行一一对应了。
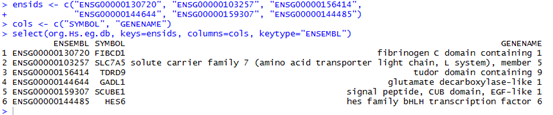
ensids <- c("ENSG00000130720", "ENSG00000103257", "ENSG00000156414",
"ENSG00000144644", "ENSG00000159307", "ENSG00000144485")
cols <- c("SYMBOL", "GENENAME")
select(org.Hs.eg.db, keys=ensids, columns=cols, keytype="ENSEMBL")
比如说,我们有几个ensembl的基因ID号。然后我们想找它所对应的gene名和缩略词简称,就通过select函数来搞定即可!
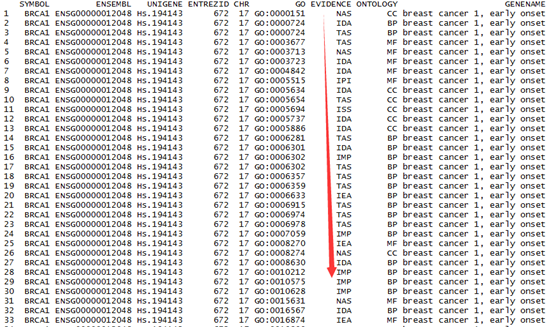
select(org.Hs.eg.db, keys="BRCA1", columns=c("ENSEMBL","UNIGENE","ENTREZID","CHR","GO","GENENAME"), keytype="SYMBOL")
这样得到了这个BRCA1基因的大部分信息,只是它的GO条目太多了,看得有点乱。