> library(org.Hs.eg.db)
载入需要的程辑包:AnnotationDbi载入需要的程辑包:stats4载入需要的程辑包:GenomeInfoDb载入需要的程辑包:S4Vectors载入需要的程辑包:IRanges载入程辑包:‘AnnotationDbi’The following object is masked from ‘package:GenomeInfoDb’: species载入需要的程辑包:DBI
1、人共有多少个entrez id的基因呢?
x <- org.Hs.egENSEMBLTRANS
# Get the entrez gene IDs that are mapped to an Ensembl ID
mapped_genes <- mappedkeys(x)
# Convert to a list
xx <- as.list(x[mapped_genes])
length(x)
[1] 47721
可知共有47721个基因都是有entrez ID号的
2、能对应转录本ID的基因有多少个呢?
length(xx)
[1] 20592
可以看到共有20592个基因都是有转录本的!
2、能对应ensembl的gene ID的基因有多少个呢?
x <- org.Hs.egENSEMBL
# Get the entrez gene IDs that are mapped to an Ensembl ID
mapped_genes <- mappedkeys(x)
# Convert to a list
xx <- as.list(x[mapped_genes])
> length(x)
[1] 47721
> length(xx)
[1] 26019
可以看到只有26019是有ensembl的gene ID的
3、那么基因对应的转录本分布情况如何呢?
table(unlist(lapply(xx,length)))
可以看出绝大部分的基因都是20个转录本一下的,但也有极个别基因居然有高达两百个转录本,很可怕!
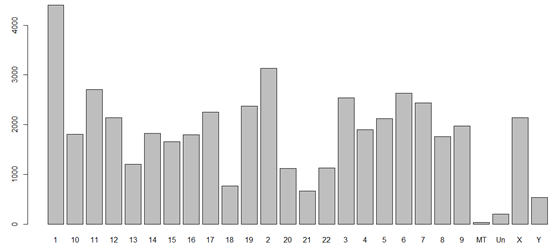
4、那么基因在染色体的分布情况如何呢?
x <- org.Hs.egCHR
# Get the entrez gene identifiers that are mapped to a chromosome
mapped_genes <- mappedkeys(x)
# Convert to a list
xx <- as.list(x[mapped_genes])
> length(x)
[1] 47721
> length(xx)
[1] 47232
可以看到有接近五百个基因居然是没有染色体定位信息的!!!
table(unlist(xx))
用barplot函数可视化一下,如图
6、那么有多多少基因是有GO注释的呢?
x <- org.Hs.egGO
# Get the entrez gene identifiers that are mapped to a GO ID
mapped_genes <- mappedkeys(x)
# Convert to a list
xx <- as.list(x[mapped_genes])
length(xx)
[1] 18229
> length(x)
[1] 47721
可以看到只有18229个基因是有go注释信息的。
那么基因被注释的go的分布如何呢?
可以看到大部分的基因都是只有30个go的,但是某些基因特别活跃,高达197个go注释。
还有kegg和omin数据库的我就不写了!