这一讲是miRNA-seq数据分析的分水岭,前面的5讲说的是读文献下载数据比对然后计算表达量,属于常规的流程分析,一般在公司测序之后都可以拿到分析结果,或者文献也会给出下载结果。但是单纯的分析一个样本意义不大,一般来说,我们做研究都是针对于不同状态下的miRNA表达量差异分析,然后做注释,功能分析,网络分析,这才是重点,也是难点。我这里就直接拿文献处理好的miRNA表达量来展示如何做下游分析,首先就是差异分析啦: Continue reading
Tag Archives: DESeq
用R语言的DESeq2包来对RNA-seq数据做差异分析
我以前写过DESeq,以及过时了:http://www.bio-info-trainee.com/867.html
正好准备筹集bioconductor中文社区,我写简单讲一下DESeq2这个包如何用!
用DESeq进行差异分析的源代码
要保证当前文件夹下面有了742KO1.count等4个文件,就是用htseq对比对的bam文件进行处理后的输出文件
library(DESeq)
#加载数据
K1=read.table("742KO1.count",row.names=1)
K2=read.table("743KO2.count",row.names=1)
W1=read.table("740WT1.count",row.names=1)
W2=read.table("741WT2.count",row.names=1)
#列名
data=cbind(K1,K2,W1,W2)
#如果是htseq的结果,则删除data最后四行
n=nrow(data)
data=data
[c language="(-n+4:-n),"][/c]
#如果是bedtools的结果,取出统计个数列和行名
kk1=cbind(K1$V5)
rownames(kk1)=rownames(K1)
K1=kk1
#差异分析
colnames(data)=c("K1","K2","W1","W2")
type=rep(c("K","W"),c(2,2))
de=newCountDataSet(data,type)
de=estimateSizeFactors(de)
de=estimateDispersions(de)
res=nbinomTest(de,"K","W")
#res就是我们的表达量检验结果
到这里,理论上差异基因的分析已经结束啦!后面只是关于R的bioconductor包的一些简单结合使用而已
library(org.Mm.eg.db)
tmp=select(org.Mm.eg.db, keys=res$id, columns=c("ENTREZID","SYMBOL"), keytype="ENSEMBL")
#合并res和tmp
res=merge(tmp,res,by.x="ENSEMBL",by.y="id",all=TRUE)
#go
tmp=select(org.Mm.eg.db, keys=res$ENSEMBL, columns="GO", keytype="ENSEMBL")
ensembl_go=unlist(tapply(tmp[,2],as.factor(tmp[,1]),function(x) paste(x,collapse ="|"),simplify =F))
#为res加入go注释,
res$go=ensembl_go[res$ENSEMBL]#为res加入一列go
#写入all——data
all_res=res
write.csv(res,file="all_data.csv",row.names =F)
uniq=na.omit(res)#删除无效基因
sort_uniq=uniq[order(uniq$padj),]#按照矫正p值排序
#写入排序后的all_data
write.csv(res,file="all_data.csv",row.names =F)
#标记上下调基因
sort_uniq$up_down=ifelse(sort_uniq$baseMeanA>sort_uniq$baseMeanB,"up","down")
#交换上下调基因列位置
final_res=sort_uniq[,c(12,1:11)]
#写出最后数据
write.csv(final_res,file="final_annotation_gene_bedtools_sort_uniq.csv",row.names =F)
#然后挑选出padj值小于0.05的数据来做富集
tmp=select(org.Mm.eg.db, keys=sort_uniq[sort_uniq$padj<0.05,1], columns="ENTREZID", keytype="ENSEMBL")
diff_ENTREZID=tmp$ENTREZID
require(DOSE)
require(clusterProfiler)
diff_ENTREZID=na.omit(diff_ENTREZID)
ego <- enrichGO(gene=diff_ENTREZID,organism="mouse",ont="CC",pvalueCutoff=0.05,readable=TRUE)
ekk <- enrichKEGG(gene=diff_ENTREZID,organism="mouse",pvalueCutoff=0.05,readable=TRUE)
write.csv(summary(ekk),"KEGG-enrich.csv",row.names =F)
write.csv(summary(ego),"GO-enrich.csv",row.names =F)
R语言DESeq找差异基因
一:安装并加装该R包
安装就用source("http://bioconductor.org/biocLite.R") ;biocLite("DESeq")即可,如果安装失败,就需要自己下载源码包,然后安装R模块。
二.所需要数据
它的说明书指定了我们一个数据
source("http://bioconductor.org/biocLite.R") ;biocLite("pasilla")
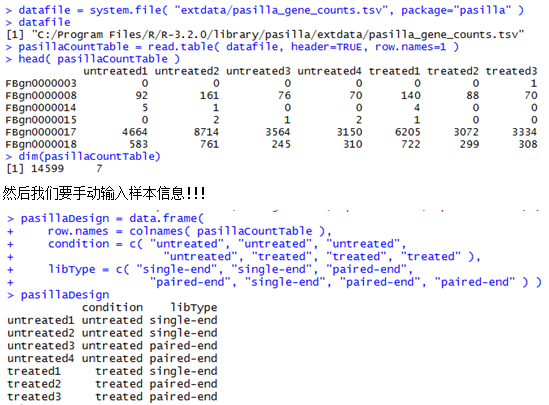
安装了pasilla这个包之后,在这个包的安装目录就可以找到一个表格文件,就是我们的DESeq需要的文件。
C:\Program Files\R\R-3.2.0\library\pasilla\extdata\pasilla_gene_counts.tsv
说明书原话是这样的
The table cell in the i-th row and the j-th column of the table tells how many reads have been mapped to gene i in sample j.
一般我们需要用htseq-count这个程序对我们的每个样本的sam文件做处理计数,并合并这样的数据
下面这个是示例数据,第一列是基因ID号,后面的每一列都是一个样本。
de = newCountDataSet( pasillaCountTable, condition ) #根据我们的样本因子把基因计数表格读入成一个cds对象,这个newCountDataSet函数就是为了构建对象!
对我们构建好的de对象就可以直接开始找差异啦!非常简单的几步即可
de=estimateSizeFactors(de)
de=estimateDispersions(de)
res=nbinomTest(de,"K","W") #最重要的就是这个res表格啦!
uniq=na.omit(res)
我这里是对4个样本用htseq计数后的文件来做的,贴出完整代码吧
library(DESeq)
#首先读取htseq对bam或者sam比对文件的计数结果
K1=read.table("742KO1.count",row.names=1)
K2=read.table("743KO2.count",row.names=1)
W1=read.table("740WT1.count",row.names=1)
W2=read.table("741WT2.count",row.names=1)
data=cbind(K1,K2,W1,W2)
data=data[-c(43630:43634),]
#把我们的多个样本计数结果合并起来成数据框,列是不同样本,行是不同基因
colnames(data)=c("K1","K2","W1","W2")
type=rep(c("K","W"),c(2,2))
#构造成DESeq的对象,并对分组样本进行基因表达量检验
de=newCountDataSet(data,type)
de=estimateSizeFactors(de)
de=estimateDispersions(de)
res=nbinomTest(de,"K","W")
#res就是我们的表达量检验结果
library(org.Mm.eg.db)
tmp=select(org.Mm.eg.db, keys=res$id, columns="GO", keytype="ENSEMBL")
ensembl_go=unlist(tapply(tmp[,2],as.factor(tmp[,1]),function(x) paste(x,collapse ="|"),simplify =F))
#首先输出所有的计数数据,加上go注释信息
all_res=res
res$go=ensembl_go[res$id]
write.csv(res,file="all_data.csv",row.names =F)
#然后输出有意义的数据,即剔除那些没有检测到表达的基因
uniq=na.omit(res)
sort_uniq=uniq[order(uniq$padj),]
write.csv(sort_uniq,file="sort_uniq.csv",row.names =F)
#然后挑选出padj值小于0.05的差异基因数据来做富集,富集用的YGC的两个包,在我前面的博客已经详细说明了!
tmp=select(org.Mm.eg.db, keys=sort_uniq[sort_uniq$padj<0.05,1], columns="ENTREZID", keytype="ENSEMBL")
diff_ENTREZID=tmp$ENTREZID
require(DOSE)
require(clusterProfiler)
diff_ENTREZID=na.omit(diff_ENTREZID)
ego <- enrichGO(gene=diff_ENTREZID,organism="mouse",ont="CC",pvalueCutoff=0.01,readable=TRUE)
ekk <- enrichKEGG(gene=diff_ENTREZID,organism="mouse",pvalueCutoff=0.01,readable=TRUE)
write.csv(summary(ekk),"KEGG-enrich.csv",row.names =F)
write.csv(summary(ego),"GO-enrich.csv",row.names =F)