我前面写到过如何把数据库写到mysql,但是发现其实msyql并不方便,需要连接数据库什么的,如果发布一个离线小网页,这时候sqlite的优点就显示出来了!
基础代码很简单:
library(RSQLite)
sqlite <- dbDriver("SQLite")
con <- dbConnect(sqlite,"hg19_bioconductor.sqlite") # makes a new file
suppressMessages(library(org.Hs.eg.db))
kegg2ID=toTable(org.Hs.egPATH)
#[1] "gene_id" "path_id"
dbWriteTable(con,'keggID2geneID',kegg2ID,row.name=F,overwrite=T)
具体代码,可以看我的github主页:https://github.com/jmzeng1314/my-R/blob/master/3-get-hg19-gene-mapping/get-hg19-gene-mapping-bioconductor.R
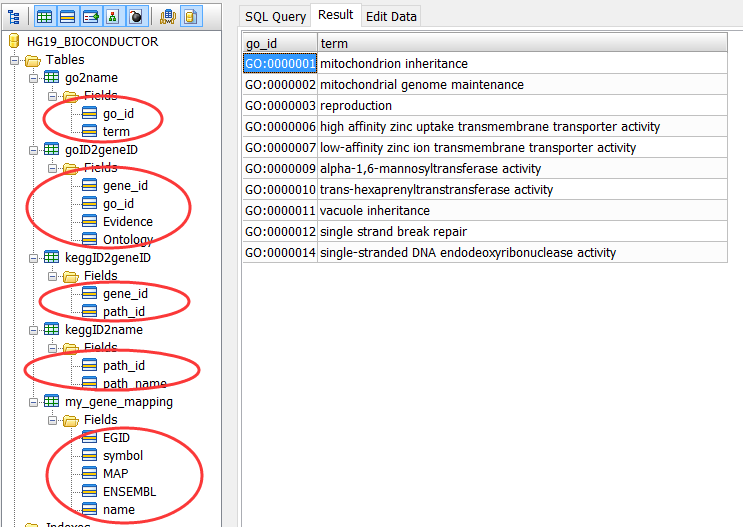
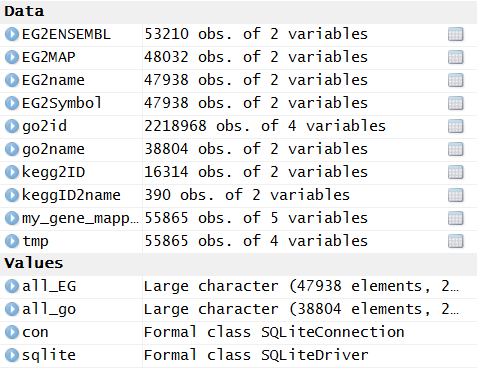
做出来的数据,如下,就是几个table存储在文件里面!
最后这些数据都保存在了当前工作目录下的hg19_bioconductor.sqlite文件里面!
library(KEGG.db)
library(GO.db)
library(org.Hs.eg.db )
很好用,跟mysql一样!