这个包跟GEOquery区别不是很大,只不过一个是正对NCBI的GEO数据库,一个是针对EBI的arrayexpress数据库,只有对写自动化脚本的人来说才有需求,一般个人分析者都是自己去数据库主页里面查找,然后拿到下载链接,一个个下载。
从EBI的arrayexpress数据库里面下载芯片数据:
update to 2016-3-1 11:41:27
63890 experiments
1912744 assays
40.53 TB of archived data 数据量还是蛮大的
所有的data,都可以在ftp服务器里面下载:ftp://ftp.ebi.ac.uk/pub/databases/arrayexpress/data/experiment/BUGS/
根据ID号很整齐的储存着。
也可以用一个R语言包:ArrayExpress R package
2009年,那个时候R语言用的人很少,这个简单的包都可以发文章,现在看来简直不可思议!
其实大部分数据都是跟GEO数据库对应的:比如https://www.ebi.ac.uk/arrayexpress/experiments/E-GEOD-55645/ 对应于:GEO - GSE55645
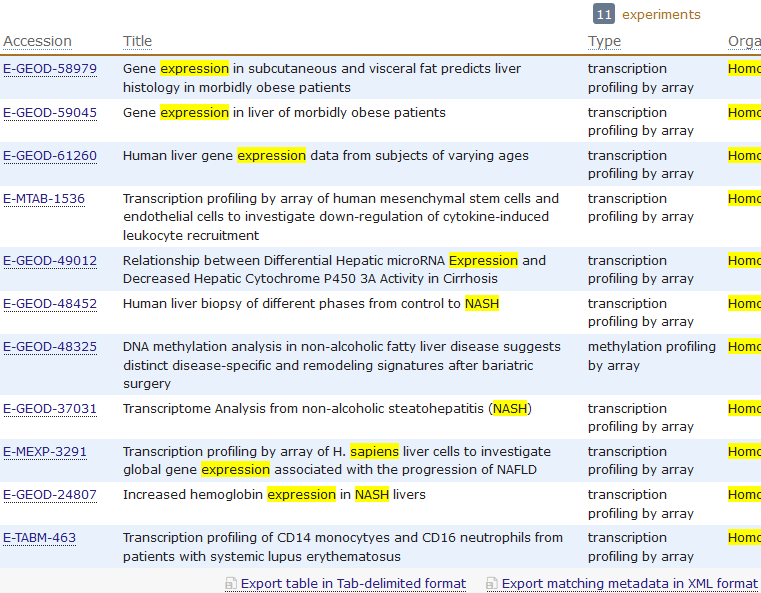
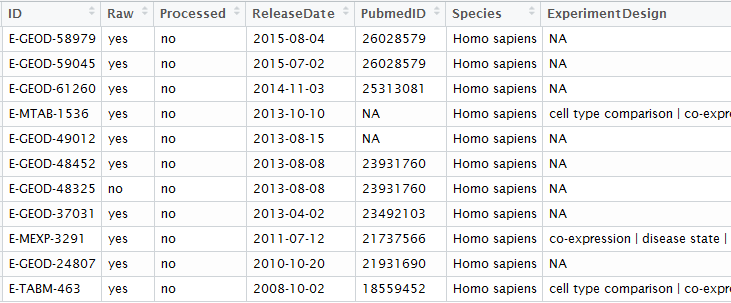
比如对NASH表达数据查找:https://www.ebi.ac.uk/arrayexpress/search.html?query=NASH++expression 30条结果里面只有4条是arrayexpress数据库独有的!
source("https://bioconductor.org/biocLite.R")
biocLite("ArrayExpress")
library(ArrayExpress)
如果用R语言,搜索如下:
可以用sets = queryAE(keywords = "NASH+expression", species = "homo+sapiens")
效果是一样的!
下载数据用:
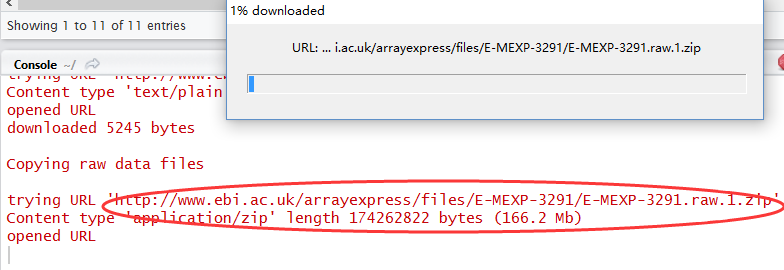
back = getAE("E-MEXP-3291")
下载其实也就是里面存储了链接,直接调用R语言的下载函数即可!
一般没必要下载原始测序文件,直接用下面这个函数就可以得到一个数据对象,可以直接得到表达矩阵和实验的metadata
rawset = ArrayExpress("E-MEXP-3291")