讲到这里,我们的自学CHIP-seq分析系列教程就告一段落了,当然,我会随时查漏补缺,根据读者的反馈来更新着系列教程。其实可视化这已经是一个比较复杂的方向了,不仅仅是针对于CHIP-seq数据。可视化本身是发文章的先决条件,而让人一目了然图片也说明了数据分析人员对数据本身的理解。我这里就列出一些目录和一些工具,和ppt。这个主要靠大家自学了,而且我博客空间有限,就不上传一大堆图片了,大家随便找一些经典的paper里面都会有很多可视化分析。
首先强烈推荐两个网页版工具,针对找到的peaks可视化:
然后再推荐一个哈佛刘小乐实验室出品的软件,也是专门为了作图http://liulab.dfci.harvard.edu/CEAS/usermanual.html
还有一个java工具:也可以可视化CHIP-seq的peaks结果EXPANDER (EXpression Analyzer and DisplayER) is a java-based tool for analysis of gene expression data.http://acgt.cs.tau.ac.il/expander/help/ver7.0Help/html/Input_Data_.htm
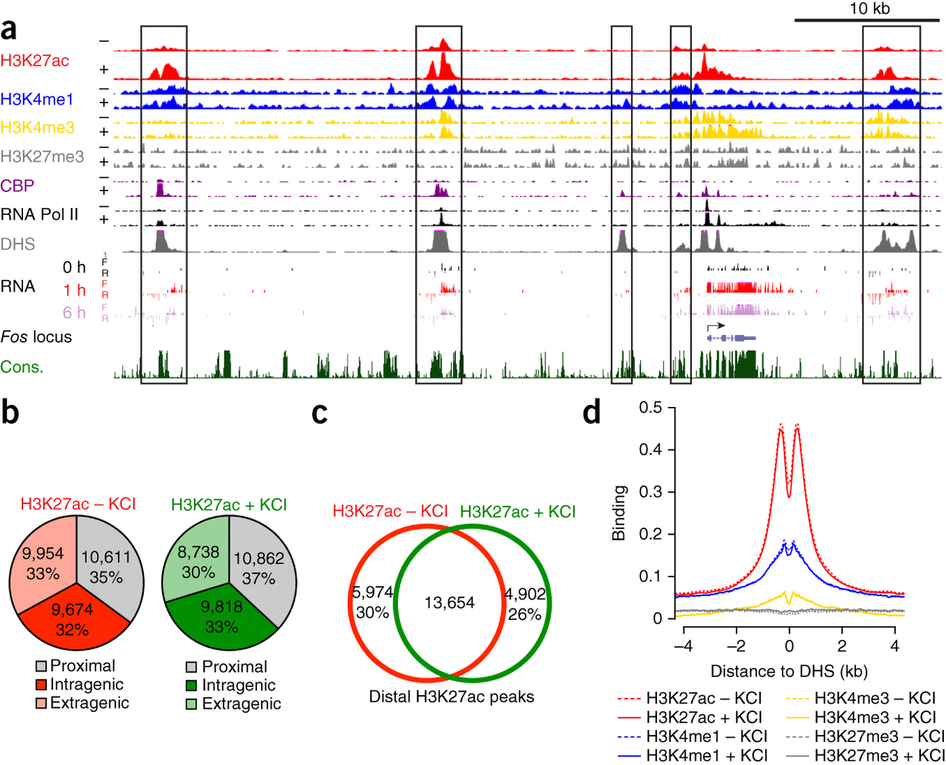
然后来随意上传一张图片吧
然后我所了解的图片大概有下面这些,都是有专门的软件,甚至自己写脚本也可以做的:
peaks长度分布柱状图
每个peak的测序情况可视化(IGV,sushi)
测序reads在全基因组各个染色体的分布(Chromosome ideograms)
reads相对基因位置分布统计
peaks相对基因位置分布统计
reads在基因组位置分布统计(染色体分开作图)
peaks在基因组位置分布统计(染色体分开作图)
统计peaks在各种基因组区域(基因上下游,5,3端UTR,启动子,内含子,外显子,基因间区域,microRNA区域)分布情况,条形图和饼图均可
Peak与转录起始位点距离的分析(曲线图和热图)
Average ChIP-Seq Gene Profile
ChIP-Seq Browser Tracks with Peak Calling
visualizes how ChIP regions are distributed over the genome along with their scores or peak heights.
可视化比较whole tiled or mappable regions + whole regions 这两种区域在全基因组各个染色体的百分比(百分比横向条形图)以及在各种genomic features的分布(百分比条形图)
display the average ChIP enrichment signals around TSS and TTS of genes, respectively(一般会把基因分成TOP10%,BOTTOM10%和ALL)
Since exon and intron lengths highly vary from gene to gene, CEAS groups exons (or introns) into multiple classes by length 看它们上面的 ChIP enrichment signals 分布情况
the average ChIP signal profiles on top 10 % , middle 10 %, and bottom 10 % of expressed genes
最后总结一下
其实有个国外的哥们也写过类似的自学教程:
一个实际的CHIP-seq数据分析例子: http://www.biologie.ens.fr/~mthomas/other/chip-seq-training/
CHIP-seq pipeline : http://www.slideshare.net/COST-events/chipseq-data-analysis
然后大家一定要看这个ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia. http://www.ncbi.nlm.nih.gov/pubmed/22955991