一般来讲,我们对测序数据进行QC,就三个大的方向:Quality trimming, Adapter removal, Contaminant filtering,当我们是双端测序数据的时候,去除接头时,也会丢掉太短的reads,就容易导致左右两端测序文件reads数量不平衡,有一个比较好的软件能解决这个问题,我比较喜欢的是cutadapt软件的PE模式来去除接头!尤其是做基因组或者转录组de novo 组装的时候,尤其要去掉接头,去的干干净净!
cutadapt是经典的python软件,但是因为我的linux服务器有点问题 ,可能是root权限问题,没有用pip install cutadapt 安装成功,我懒得搞这些了,其实可以自己去下载cutadapt的源码,然后进入源码文件夹里面 python setup.py install --user 到自己的 ~/.local/bin下面。
所以我用conda安装了cutadapt软件,http://www.bio-info-trainee.com/1906.html 所以我需要 python ~/miniconda2/pkgs/cutadapt-1.10-py27_0/bin/cutadapt --help 才能调用这个软件,不过,问题不大,我也就是试用一下。 Continue reading
Category Archives: 基础软件
用sickle软件来对双端测序数据过滤低质量reads
一般来讲,我们对测序数据进行QC,就三个大的方向:Quality trimming, Adapter removal, Contaminant filtering,当我们是双端测序数据的时候,去除低质量的reads就容易导致左右两端测序文件不平衡,有一个比较好的软件能解决这个问题,而且软件使用非常简单! Continue reading
大型基因组拼装的乐高软件之MaSuRCA assembler使用指南
本文转载自 生信技能树 论坛特约作者Mint 的 MaSuRCA assembler 软件指导书,非常符合我博客的风格,也正式开启了我博客的转载之路。(前面的近400篇文章都是本人原创,手打,但是精力有限,以后文章更新频率会大大降低,但是会引入不少 技能树论坛特约作者的 好文!) Continue reading
用Miniconda,Bioconda来安装常见的生物信息学软件
 https://bioconda.github.io/
https://bioconda.github.io/用 SHRiMP 来比对color space的数据
0=blue, 1=green, 2=orange, 3=red) or as characters A/C/G/T (A=blue, C=green, G=orange, T=red).我们通常称为csfastq格式。使用CEAS软件来对CHIP-seq的peaks进行
用网页版工具GREAT来对CHIP-seq的peaks进行下游功能分析
用网页版工具ChIPseek来可视化CHIP-seq的peaks结果
用R包BayesPeak来对CHIP-seq数据call peaks
BayesPeak也是peaks caller家族一员,用的人也不少,我这次也试了一下,因为是R的bioconductor系列包,所以直接在R里面安装就好,但是有几个点需要注意,我比对的基因组不只是Chr1~22,X,Y,M,还有一些contig和scaffold,需要在bam文件里面去除的,而且BayesPeak比较支持读取BED文件,可以直接转为GRanges对象,虽然它号称可以使用多核,但是计算速度还是非常慢。 Continue reading
用PeakRanger软件来对CHIP-seq数据call peaks
此文专门讲这个软件如何用,但是跟我以前写的软件说明书又不大一样,主要是因为我用MACS2这个软件call peaks并没有达到预期的结果,所以就多使用了几个软件,其中PeakRanger尤其值得一提,安装特别简单,而且处理数据的速度特别快,结果也非常容易理解,更重要的是它给出一个网页版的报告,里面有所有找到的符合要求的peaks的可视化图片!!!! Continue reading
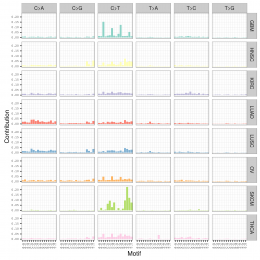
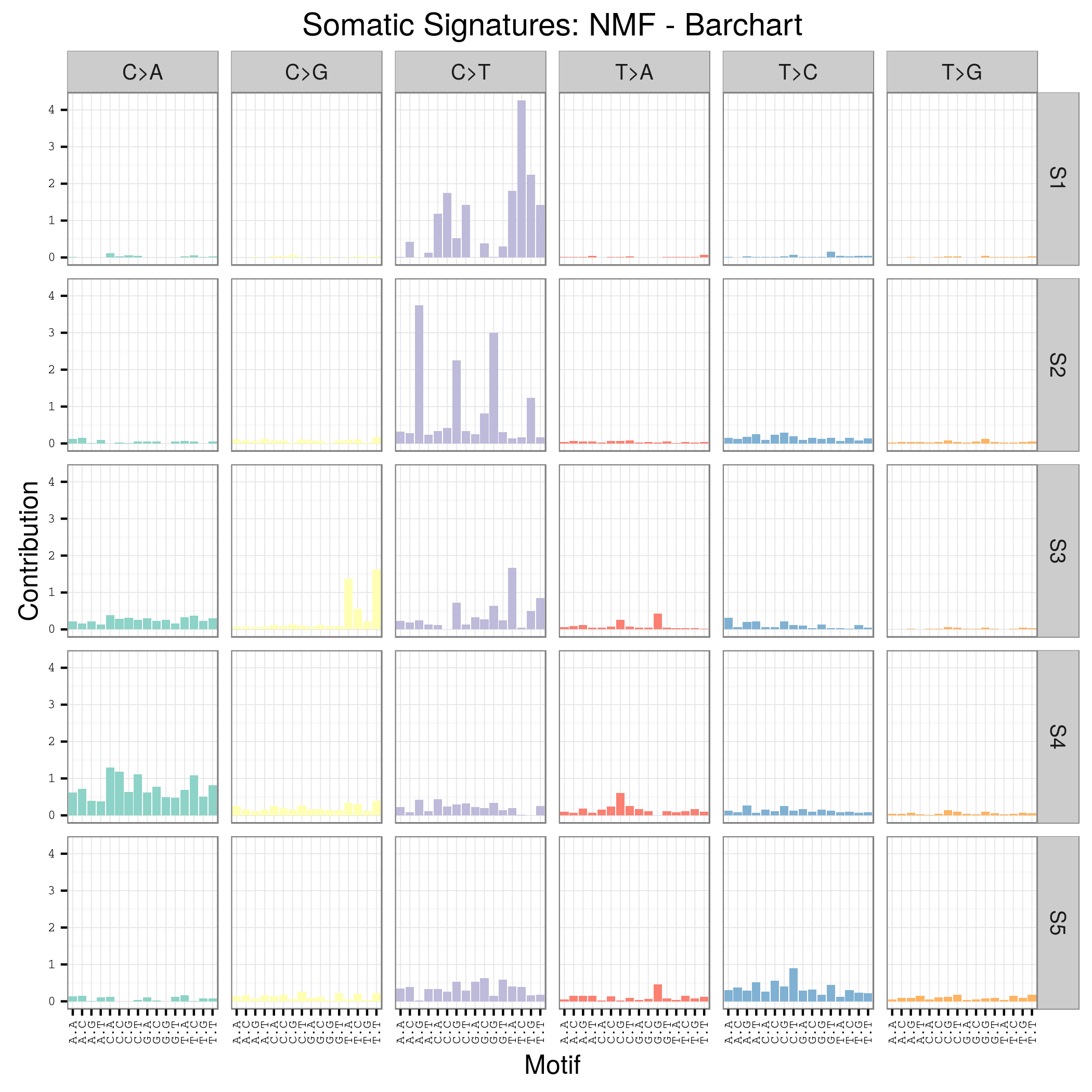
用SomaticSignatures包来解析maf突变数据获得mutation signature
mutation signature这个概念提出来还不久,我看了看文献,最早见于2013年的一篇nature文章,主要是用来描述癌症患者的somatic mutation情况的。
首先要自己分析癌症样本数据,拿到somatic mutation,TCGA计划发展到现在已经有非常多的somatic mutation结果啦,大家可以自行选择感兴趣的癌症数据拿来研究,解析一下mutation signature 。
我这里给大家推荐一个工具,是R语言的Bioconductor系列包中的一个,SomaticSignatures
其实它的说明书写的非常详细了已经,如果你理解了mutation signature的概念,很容易用那个包,其实你自己写一个脚本也是非常任意的,就是根据mutation的位置在基因组中找到它的前后一个碱基,然后组成三碱基突变模式,最后统计一下那96种突变模式的分布状况!
我这里简单讲一讲这个包如何用吧!
首先下载并加载几个必须的包:
然后根据突变数据做好一个GRanges对象,这个可以看我以前的博客
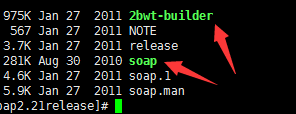
华大soap系列的比对软件
也不知道是什么原因,对国产软件总是提不起兴趣,所以尽管SOAP系列都已经发展到了十几个软件了,我依然没有去试用一下。
# download a test reference genome (TAIR9 Chromosome 1)
wgethttp://biocluster.ucr.edu/~tbackman/query.fastq# download some test Illumina reads from Arabidopsis
运行命令:
2bwt-builder genome.fasta
# create binary of reference genome
soap -a query.fastq -D genome.fasta.index -o output.soap
# align query to genome and store output
结果解读:
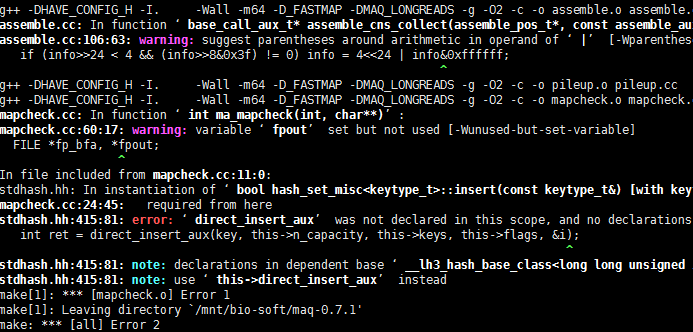
很老的比对软件maq
tar jxvf software.tar.bz2cd software./configure --prefix=$pathmakemake test
# download a test reference genome (TAIR9 Chromosome 1)
wgethttp://biocluster.ucr.edu/~tbackman/query.fastq# download some test Illumina reads from Arabidopsis
运行命令:
maq # inspect command line options
maq fasta2bfa genome.fasta genome.bfa
# create binary of reference genome
maq fastq2bfq query.fastq readBinary.bfq
# create a binary of dataset
maq match out.map genome.bfa readBinary.bfq
# align query to genome and store output
结果解读:
out.map肯定不是sam格式的。哈哈,这个软件我无法安装,换了好几系统也没成功,如果是太老了,很多库文件却是。我也懒得去解决了。这种报错,对我这样的非计算机专业来说,简直是天书!用samr包对芯片数据做差异分析
本来搞差异分析的工具和包就一大堆了,而且limma那个包已经非常完善了,我是不准备再讲这个的,正好有个同学问了一下这个包,我就随手测试了一下,顺便看看它跟limma有什么差异没有!手痒了就记录了测试流程!
学习一个包其实非常简单,就是找到包的官网看看说明书即可!说明书链接
用GEMINI来探索vcf格式的突变数据
第一次听说这个软件,是一个香港朋友推荐的:http://davetang.org/muse/2016/01/13/getting-started-with-gemini/ 他写的很棒,但是我当初以为是一个类似于SQLite的数据库浏览模式,所以没在意。实际上,我现在仍然觉得这个软件没什么用!
软件官网有详细的介绍:https://gemini.readthedocs.io/en/latest/
而且提供丰富的教程:
We recommend that you follow these tutorials in order, as they introduce concepts that build upon one another.
- Introduction to GEMINI, basic variant querying and data exploration. html pdf
- Identifying de novo mutations underlying Mendelian disease html pdf
- Identifying autosomal recessive variants underlying Mendelian disease html pdf
- Identifying autosomal dominant variants underlying Mendelian disease html pdf
- Other GEMINI tools html pdf
软件本身并不提供注释,虽然它的功能的确包括注释,号称可以利用(ENCODE tracks, UCSC tracks, OMIM, dbSNP, KEGG, and HPRD.)对你的突变位点注释,比如你输入1 861389 . C T ,它告诉你这个突变发生在哪个基因,对蛋白改变如何?是否会产生某些疾病?
虽然它本身没有注释功能,但是它会调用snpEFF或者VEP进行注释,你需要自己先学习它们。
软件安装:
GEMINI是用python写的,有一个小脚本可以自动完成安装过程:
7.3K May 4 14:44 gemini_install.py
下载这个脚本,然后安装即可
wget https://github.com/arq5x/gemini/raw/master/gemini/scripts/gemini_install.py
python gemini_install.py $tools $data
PATH=$tools/bin:$data/anaconda/bin:$PATH
where $tools and $data are paths writable on your system.
我把$tools用的就是当前文件夹,$data也是当前文件夹下面的gemini文件夹。
这样就会在当前文件夹下面生成两个文件夹,bin是存储程序,gemini是存储数据用的,而且注意要把bin目录的全路径添加到环境变量!
输入数据:
我们可以直接下载软件作者提供的测试数据
首先是22号染色体的所有突变位点经过WEP注释的文件
然后是一个三口直接的突变ped格式数据
数据存放在亚马逊云,所有的教程pdf也在
http://s3.amazonaws.com/gemini-tutorials
如果是你自己的vcf文件,需要自己用VEP注释一下
运行命令:
结果解读:
产生是chr22.db就是一个数据库格式的文件,但是需要用gemini 来进行查询,个人认为,并没有多大意思!
你只要熟悉mySQL等SQL语言,完全可以自己来!
用VEP对vcf格式的突变数据进行注释
VEP是国际三大数据库之一的ENSEMBL提供的,也是非常主流和方便,但它是基于perl语言的,所以在模块方面可能会有点烦人。跟snpEFF一样,也是对遗传变异信息提供更具体的注释,而不仅仅是基于位点区域和基因。如果你熟悉外显子联盟这个数据库EXAC(ExAC.r0.3.sites.vep.vcf.gz),你可以下载它所有的突变记录数据,看看它对每个变异位点到底注释了些什么,它就是典型的用VEP来注释的。 Continue reading
用snpEFF对vcf格式的突变数据进行注释
这个软件比较重要,尤其是对做遗传变异相关研究的,很多人做完了snp-calling后喜欢用ANNOVAR来进行注释,但是那个注释还是相对比较简单,只能得到该突变位点在基因的哪个区域,那个基因这样的信息,如果想了解更具体一点,就需要更加功能化的软件了,snpEFF就是其中的佼佼者,而且是java平台软件,非常容易使用!而且它的手册写的非常详细:http://snpeff.sourceforge.net/SnpEff_manual.html Continue reading
用oligo包来读取affymetix的基因表达芯片数据-CEL格式数据
前面讲到affy处理的芯片平台是有限的,一般是hgu 95系列和133系列,[HuGene-1_1-st] Affymetrix Human Gene 1.1 ST Array这个平台虽然也是affymetrix公司的,但是affy包就无法处理 了,这时候就需要oligo包了!
oligo包是R语言的bioconductor系列包的一个,就一个功能,读取affymetix的基因表达芯片数据-CEL格式数据,处理成表达矩阵!!!
用affy包读取affymetix的基因表达芯片数据-CEL格式数据
Affymetrix的探针(proble)一般是长为25碱基的寡聚核苷酸;探针总是以perfect match 和mismatch成对出现,其信号值称为PM和MM,成对的perfect match 和mismatch有一个共同的affyID。
CEL文件:信号值和定位信息。
CDF文件:探针对在芯片上的定位信息
affy包是R语言的bioconductor系列包的一个,就一个功能,读取affymetix的基因表达芯片数据-CEL格式数据,处理成表达矩阵!!!
用limma包的voom函数来对RNA-seq数据做差异分析
limma真不愧是最流行的差异分析包,十多年过去了,一直是芯片数据处理的好帮手。
现在又可以支持RNA-seq数据,我赶紧试用了一下!
我下面只讲用法,大家看代码就明白了!