这个TCGA表达数据的多项应用系列帖子是应群里朋友的要求来写的,你们也可以继续提需求,我会接着写下去,其实从TCGA数据库里面下载到了数据之后,后面的所有分析都跟TCGA没有半毛钱关系了,大家要有这个想法,别三两句就问TCGA数据怎么分析,http://www.bio-info-trainee.com/?s=TCGA&submit=Search 本系列最后会形成一个shiny版本的交互式表达数据查询,处理,绘图,统计的网页APP。
我这里偷懒一下了,直接下载GEO里面的TCGA的表达数据,而不是去TCGA的官网里面下载:
它处理了目前(大概是2015年6月)TCGA收集的所有癌症样本的mRNA表达数据,并且统一处理成了count和RPKM两种表达量形式。 GEO地址:http://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE62944
下载的文件,首先用R连接mysql把RPKM形式的表达数据写到数据库啦!
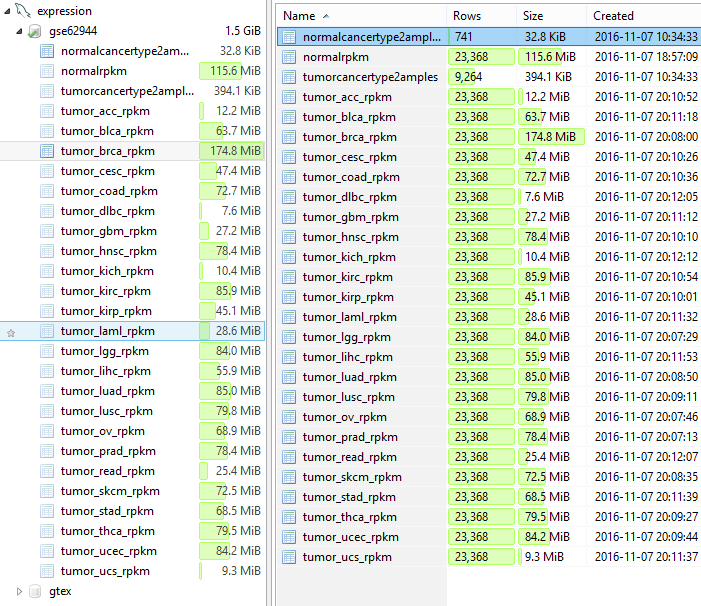
很明显,我们需要下载正常样本和癌症样本的ID和组织对应表格,还有他们的RPKM矩阵,是压缩包格式的txt文档,然后用R语言链接mysql写入,至于为什么要写入的mysql,主要是因为R里面读取文件太慢了,而我后面的一系列表达数据的分析都是高频的,不可能每次都打开R,然后读取几个G的数据,大家看下面的表格就应该明白,我把正常组织样本的表达数据写到了一个表格里面,而把癌症根据tissue来分开存放,主要是因为mysql有最大列数限制,而我们这个GSE62944里面有近万的样本了。
数据下载就是在GEO的supplement里面找链接,写入到mysql,首先需要你自己安装了mysql,请看我在论坛写的教程:http://www.biotrainee.com/thread-389-1-1.html
可以看到R其实就是通过R的包来调用系统的mysql程序,往mysql里面写入数据,而我们的表达数据还比较大,所以会耗费半小时左右。
后面一系列的分析,都是基于此,所以你要想看懂后面的帖子,必须要用我的代码下载好数据,并导入到自己的mysql里面。(我代码在:https://github.com/jmzeng1314/myTCGA/blob/master/expression/mRNA/GSE62944/GSE62944data2mysql.R)
作为一个合格的生信工程师,有些东西,你不得不学习,抱歉!