最近在忙一些chip-seq的数据分析项目,它的可视化展现比较复杂一点,自己写程序将会耗费挺长时间的,就想着利用现成的工具,前面试用了deeptools,挺好 的,但是有点慢,是python程序,如下:
现在换一个R程序,这个非常快速,而且绘图个人觉得稍微美观一点,大家也可以都试试看。
首先软件的github里面有源代码,然后作者还四处宣讲这个包的神奇之处,下面的ppt非常言简意赅的描述了它的功能和强大之处。
安装超级简单啦,只需要去Google的云盘里下载软件和测试数据咯
cd ~/biosoft
mkdir ngsplot && cd ngsplot
## download by yourself :https://drive.google.com/drive/folders/0B1PVLadG_dCKN1liNFY0MVM1Ulk
tar -zxvf ngsplot-2.61.tar.gz
tar zxvf ngsplot.eg.bam.tar.gz ## 测试数据非常给力,清楚的说明了,CHIP-seq数据分析-可视化需要什么样的数据。
cp ../ngsplot/example/config.example.txt ./ ## 在后面的测试代码需要用
echo 'export PATH=/home/jianmingzeng/biosoft/ngsplot/ngsplot/bin:$PATH' >>~/.bashrc
echo 'export NGSPLOT=/home/jianmingzeng/biosoft/ngsplot/ngsplot' >>~/.bashrc
source ~/.bashrc
## 需要你的服务器安装好R,并且你自己手动安装好这几个包。
install.packages("doMC", dep=T)
install.packages("caTools", dep=T)
install.packages("utils", dep=T)
source("http://bioconductor.org/biocLite.R")
biocLite( "BSgenome" )
biocLite( "Rsamtools" )
biocLite( "ShortRead" )
使用非常简单,看懂ngs.plot.r的用法即可,一个命令就出图了,如果这个出图不满意,就用replot.r重新选择参数绘制一个新的图!
前提是自己下载好了基因组文件,本软件自带hg19,其余的基因组有:https://github.com/shenlab-sinai/ngsplot/wiki/SupportedGenomes , 但是都放在Google云盘里面,所以需要翻墙才能下载的: https://drive.google.com/drive/folders/0B1PVLadG_dCKNEsybkh5TE9XZ1E
测序数据如下:
有了这些测试数据,而且软件里面还自带了测试代码:
ngs.plot.r -G hg19 -R tss -C hesc.H3k4me3.1M.bam -O k4.test
ngs.plot.r -G hg19 -R tss -C config.example.txt -O encode1M.k4k27
如果需要对多个bam文件画图,就根据作者定义的规则来设置好config.example.txt 文件即可
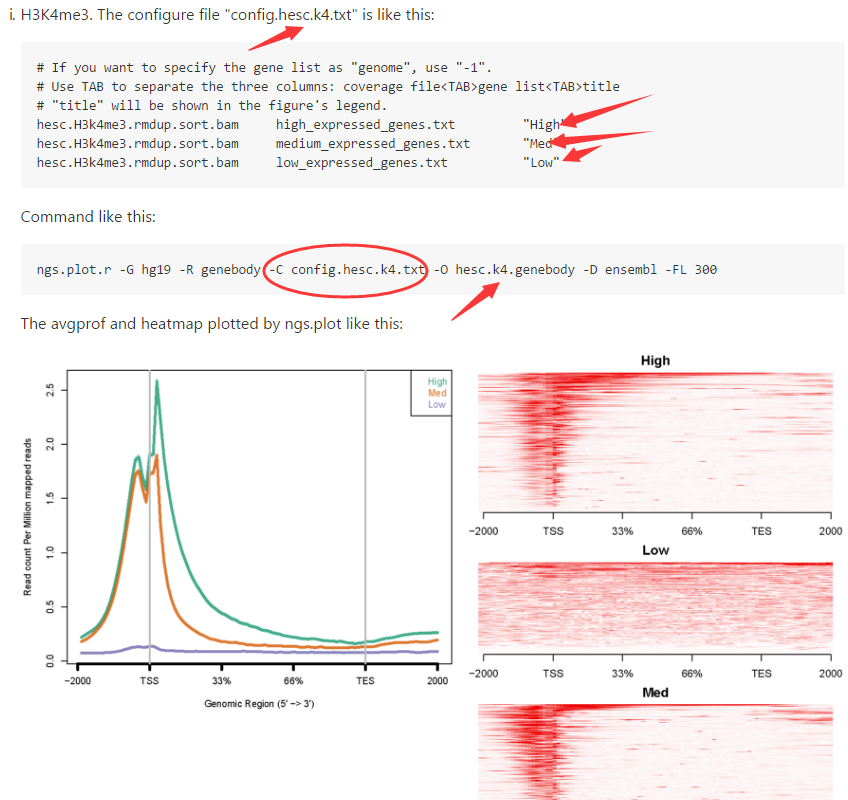
如果你对上面的图不满意,可以用replot.r 来重新根据上面的参数来画图。
replot.r prof -I k4.test.zip -O k4.replot -SE 0 -MW 9 -H 0.3
replot.r heatmap -I encode1M.k4k27.zip -O k4k27.replot -GO hc -RR 80
除了以tss来画图,还可以根据genebody或者其它: tss, tes, genebody, exon, cgi, enhancer, dhs or bed
ngs.plot.r -G hg19 -R genebody -F rnaseq -C hesc.RNAseq.1M.bam -O encode1M.rnaseq
ngs.plot.r -G hg19 -R tss -C hesc.H3k4me3.1M.bam:hesc.Input.500K.bam -O k4vsInp
轻轻松松get到作者的意图,然后拿自己的数据就可以做同样的分析图片了!
当然,如果你领悟力比较差,慢慢读人家的github上面的readme吧,实在是太简单了,我都不知道需要我讲什么。
而且运行速度还特快!
当然,也可能是它这个测试文件本来就很小的原因。
The genome files can be found in this Google drive folder: ngs.plot genome folder. A list of the available genomes is listed in this Wiki: SupportedGenomes. A brief list is here (not all): "human (hg18, hg19), chimpanzee (panTro4), rhesus macaque (rheMac2), mouse (mm9, mm10), rat (rn4, rn5), cow (bosTau6), chicken (galGal4), zebrafish (Zv9), drosophila (dm3), Caenorhabditis elegans (ce6, ceX), Saccharomyces cerevisiae (sacCer2, sacCer3), Schizosaccharomyces pombe (Asm294), Arabidopsis thaliana (TAIR10), Zea mays (AGPv3), rice (IRGSP-1.0)".