请不要直接拷贝我的代码,需要自己理解,然后打出来,思考我为什么这样写代码。
软件请用最新版,尤其是samtools等被我存储在系统环境变量的,考虑到读者众多,一般的软件我都会自带版本信息的!
我用两个小时,不代表你是两个小时就学会,有些朋友反映学了两个星期才 学会,这很正常,没毛病,不要异想天开两个小时就达到我的水平。
本次讲解选取的文章是为了探索PRC1,PCR2这样的蛋白复合物,不是转录因子或者组蛋白的CHIP-seq,请注意区别!
这是一个系列帖子,你可以先看:
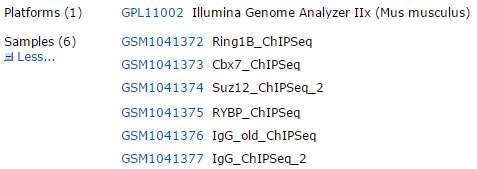
文章是:RYBP and Cbx7 define specific biological functions of polycomb complexes in mouse embryonic stem cells
RYBP and Cbx7都是Polycomb repressive complex 1 (PRC1)的组分:
所以用脚本在ftp里面批量下载即可:
下载地址很容易获取啦!
ls *sra |while read id; do ~/biosoft/sratoolkit/sratoolkit.2.6.3-centos_linux64/bin/fastq-dump --split-3 $id;done
图片丢失~~~~~
因为我用fastqc看了看数据质量,代码如下:
ls *fastq |xargs ~/biosoft/fastqc/FastQC/fastqc -t 10
发现3端质量有点问题,我就用了-3 5 --local参数,
首先用bowtie2软件把测序得到的fastq文件比对到mm10参考基因组上面
~/biosoft/bowtie/bowtie2-2.2.9/bowtie2 -p 6 -3 5 --local -x ~/reference/index/bowtie/mm10 -U SRR620204.fastq| samtools sort -O bam -o ring1B.bam
~/biosoft/bowtie/bowtie2-2.2.9/bowtie2 -p 6 -3 5 --local -x ~/reference/index/bowtie/mm10 -U SRR620205.fastq| samtools sort -O bam -o cbx7.bam
~/biosoft/bowtie/bowtie2-2.2.9/bowtie2 -p 6 -3 5 --local -x ~/reference/index/bowtie/mm10 -U SRR620206.fastq| samtools sort -O bam -o suz12.bam
~/biosoft/bowtie/bowtie2-2.2.9/bowtie2 -p 6 -3 5 --local -x ~/reference/index/bowtie/mm10 -U SRR620207.fastq| samtools sort -O bam -o RYBP.bam
~/biosoft/bowtie/bowtie2-2.2.9/bowtie2 -p 6 -3 5 --local -x ~/reference/index/bowtie/mm10 -U SRR620208.fastq| samtools sort -O bam -o IgGold.bam
~/biosoft/bowtie/bowtie2-2.2.9/bowtie2 -p 6 -3 5 --local -x ~/reference/index/bowtie/mm10 -U SRR620209.fastq| samtools sort -O bam -o IgG.bam
接下来需要对bam文件进行简单过滤,包括未比对的和multiple比对的,但是我比较懒,就直接用MACS2软件来call peaks啦!
nohup ~/.local/bin/macs2 callpeak -c ../IgGold.bam -t ../suz12.bam -m 10 30 -p 1e-5 -f BAM -g mm -n suz12 2>suz12.masc2.log &
nohup ~/.local/bin/macs2 callpeak -c ../IgGold.bam -t ../ring1B.bam -m 10 30 -p 1e-5 -f BAM -g mm -n ring1B 2>ring1B.masc2.log &
nohup ~/.local/bin/macs2 callpeak -c ../IgG.bam -t ../cbx7.bam -m 10 30 -p 1e-5 -f BAM -g mm -n cbx7 2>cbx7.masc2.log &
nohup ~/.local/bin/macs2 callpeak -c ../IgG.bam -t ../RYBP.bam -m 10 30 -p 1e-5 -f BAM -g mm -n RYBP 2>RYBP.masc2.log &
大家可以看到RYBP这个CHIP-seq我几乎得不到peaks,哪怕是换了一个control,除非我不用任何control!我用IGV看了看,这个RYBP的确很诡异,我怀疑是作者上传数据出错了!
而且作者在GEO给的PEAKS个数如下:
2754 GSE42466_Cbx7_peaks_10.txt
6982 GSE42466_Ring1b_peaks_10.txt
6872 GSE42466_RYBP_peaks_5.txt
8054 GSE42466_Suz12_peaks_10.txt
首先对这些bam文件批量转换成bw文件。然后批量画图
ls ../*bam |while read id
do
file=$(basename $id )
sample=${file%%.*}
echo $sample
bamCoverage -b $id -o $sample.bw ## 这里有个参数,-p 10 --normalizeUsingRPKM
computeMatrix reference-point --referencePoint TSS -b 10000 -a 10000 -R ~/annotation/CHIPseq/mm10/ucsc.refseq.bed -S $sample.bw --skipZeros -o matrix1_${sample}_TSS.gz --outFileSortedRegions regions1_${sample}_genes.bed
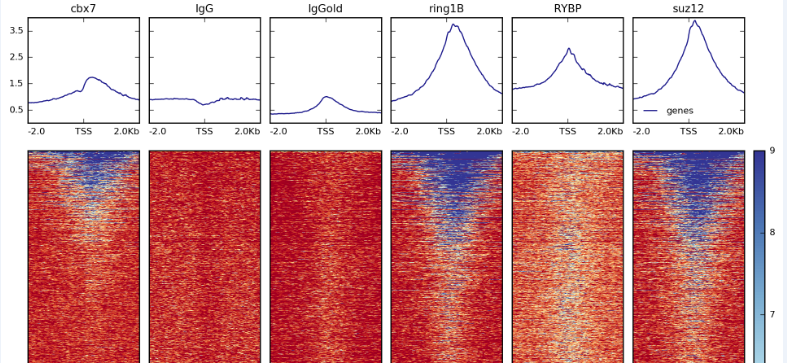
plotHeatmap -m matrix1_${sample}_TSS.gz -out ${sample}.png
done
然后整合所有的chipseq的bam文件,画基因的TSS附近的profile和heatmap图
computeMatrix reference-point -p 10 --referencePoint TSS -b 2000 -a 2000 -S ../*bw -R ~/annotation/CHIPseq/mm10/ucsc.refseq.bed --skipZeros -o tmp4.mat.gz
plotHeatmap -m tmp4.mat.gz -out tmp4.merge.png
plotProfile --dpi 720 -m tmp4.mat.gz -out tmp4.profile.pdf --plotFileFormat pdf --perGroup
plotHeatmap --dpi 720 -m tmp4.mat.gz -out tmp4.merge.pdf --plotFileFormat pdf
最后整合所有的chipseq的bam文件,画基因的genebody附近的profile和heatmap图
computeMatrix scale-regions -p 10 -S ../*bw -R ~/annotation/CHIPseq/mm10/ucsc.refseq.bed -b 3000 -a 3000 -m 5000 --skipZeros -o tmp5.mat.gz
plotHeatmap -m tmp5.mat.gz -out tmp5.merge.png
plotProfile --dpi 720 -m tmp5.mat.gz -out tmp5.profile.pdf --plotFileFormat pdf --perGroup
plotHeatmap --dpi 720 -m tmp5.mat.gz -out tmp5.merge.pdf --plotFileFormat pdf
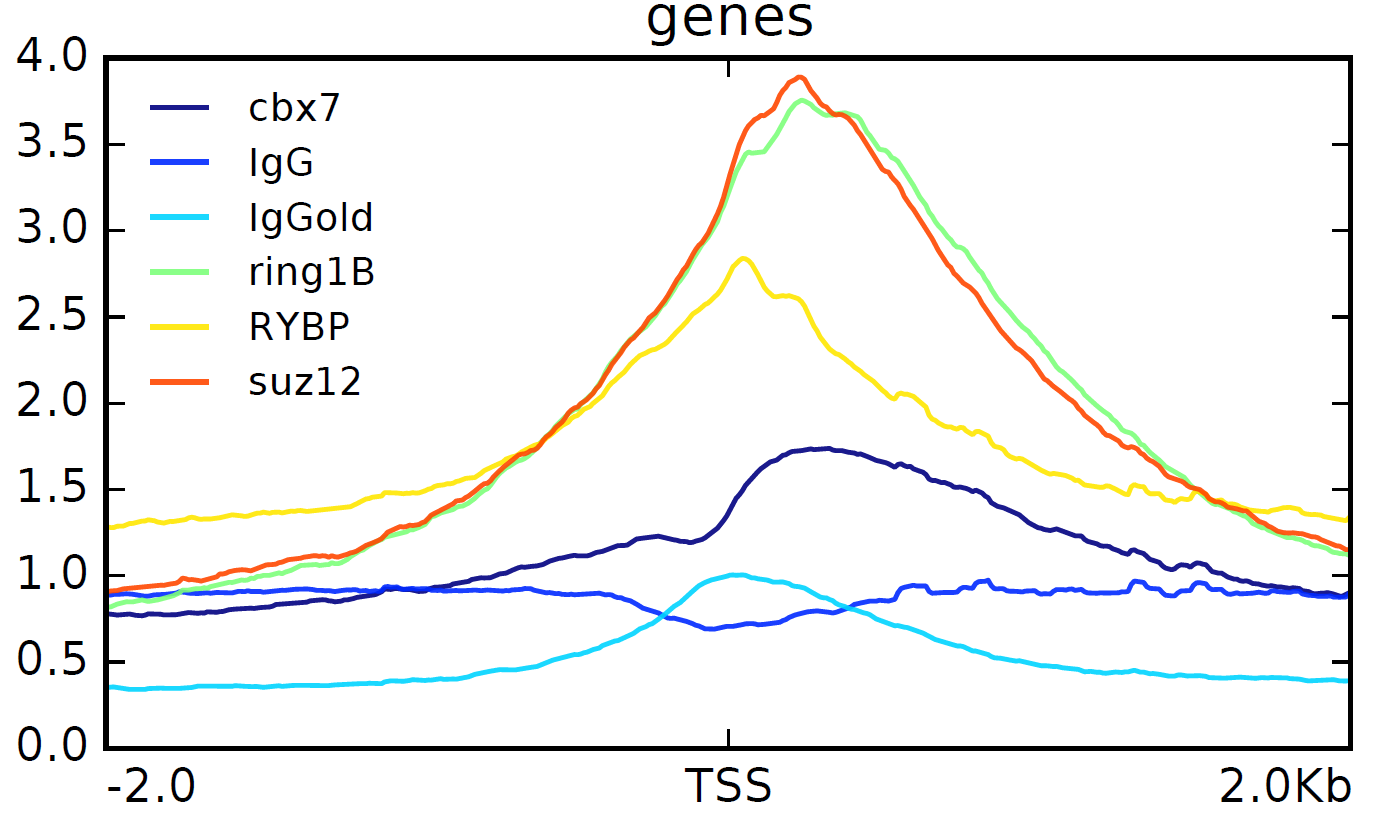
下面是输出的图的例子,我只放了tss附近的!
上图可以看到RYBP的peaks的中点在TSS处,而其它peaks都在TSS下游一点点。
用Sequential ChIP (re-ChIP)实验的确可以看到RYBP和CBX7的peaks有重合。
这篇文章一直翻来覆去说 这些CHIP-seq实验的peaks的交叉情况:
PRC1的组分异常复杂,包括 Cbx (Cbx2, Cbx4, Cbx6, Cbx7, or Cbx8); Ring1A or Ring1B; PHC (PHC1, PHC2, or PHC3); PCGF (PCGF1, PCGF2, PCGF3, PCGF4, PCGF5, or PCGF6); and RYBP or YAF2.
其中,a Ring1A/B E3 ligase subunit that monoubiquitinates histone H2A at lysine 119 (H2AK119ub)
但不是说都必须要有,而是它们的组合,形成了各种各样的PRC1,但是都统一叫做PRC1。
比如在mouse的ESCs里面,就有两种PRC1,它们的 Cbx7 or RYBP 是不可能共存的!我们可以把它们分别叫做, Cbx7-PRC1, RYBP-PRC1Cbx7 的功能是把 Ring1B 招募到染色质上面,是必须的。它结合的基因多参与 early-lineage commitment of ESCs.
RYBP 可以增强PRC1的酶活性,它结合大基因多参与,regulation of metabolism and cell-cycle progression
RYBP 结合的基因要比 CBX7 结合的基因表达量高。 因为CBX7结合的同时,会招募PRC2这个抑制marker。
而PRC2 deposits the histone H3 lysine 27 trimethyl repressive mark (H3K27me3) through the Ezh1/2 histone methyltransferase enzymes.如何描述它们这些peaks的交叉情况呢?
We observed an overlap of RYBP peaks (3,918 in total) with 14%, 42%, and 37% of Cbx7, Ring1B, and Suz12 peaks, respectively
Moreover, although more than 90% of Cbx7 peaks contained Ring1B and Suz12, 20% were also bound by RYBP
尽管RYBP and Cbx7 在大部分情况下都是互相排斥的,但是也在少部分基因组区域存在共定位的现象。Ring1B / Suz12的peaks情况可以被 Cbx7 和 RYBP 的peaks情况说明:
RYBP and Cbx7 都有的地方,有着高Ring1B/Suz12
Cbx7 but not RYBP的地方,Ring1B/Suz12会稍微低一点
RYBP but not Cbx7的地方,Ring1B/Suz12会更低一点
RYBP and Cbx7 都没有的地方,Ring1B/Suz12就最少!RYBP的peaks的中点在TSS处,而其它peaks都在TSS下游一点点。
用Sequential ChIP (re-ChIP)实验的确可以看到RYBP和CBX7的peaks有重合。而且RYBP还有一些peaks是其它PRC1所没有的,说明它可以独立于PRC1发挥作用H2AK119ub 与 Ring1B/Suz12正相关,但是与RYBP只有25.7%交叉,与CBX7有着72%交叉,所以可以把 PRC1 target genes分成3类:
a first set with Cbx7/Ring1B/H2AK119ub; ~~~~GO/KEGG分析,
a second that contains RYBP and lower levels of Ring1B/H2AK119ub
a third set cobound by RYBP/Cbx7/Ring1B and that also contains H2AK119ub.
然后这些所有的gene list都可以拿去做GO/KEGG分析,看看是不是有什么biological meaning !
genes co-occupied by Ring1B/Cbx7/RYBP and H2AK119ub are involved in system development.
genes containing RYBP/Ring1B/H2AK119ub, but not Cbx7, have a strong association with the M phase of the meiotic cycle and cellular metabolism
genes with Cbx7/Ring1B/H2AK119ub are involved in developmental processes and mesoderm specification,
those containing RYBP/Cbx7/Ring1B/H2AK119ub predominantly represent the ectodermal fate and, to a lesser extent, mesoderm and endoderm fates
超过700的基因有 RYBP/Cbx7/Ring1B的peaks,所以作者敲除Cbx7 看看 RYBP的peaks是否会变化,但是没有做CHIP-seq,只是做了ChIP-qPCR
下面这个结论很重要:
Overall, our ChIP-seq analysis allowed us to identify five types of genes according to the occupancy of PRC1 and PRC2: those with
(1) Ring1B/Cbx7/RYBP and Suz12 (725 genes);
(2) Ring1B/Cbx7/Suz12, but not RYBP (1,527 genes);
(3) Ring1B/RYBP/Suz12, but not Cbx7 (861 genes);
(4) only Ring1B and Suz12 (1,694 genes); or
(5) RYBP but no Polycomb proteins (1,674)