用rmarkdown写教程真心非常方便,尤其是R语言相关的,比如一些R包的应用,或者一些可视化,或者一些统计,下面我简单列出一些我以前写过的,图文并茂,关键是还非常省心,不需要排版,不需要上传图片,整理图片。
一般来说看链接最后的文件名就知道这篇文章讲的是什么了:
首先是几个R包的讲解:
http://www.bio-info-trainee.com/ ... software/limma.html
http://www.bio-info-trainee.com/ ... oftware/DESeq2.html
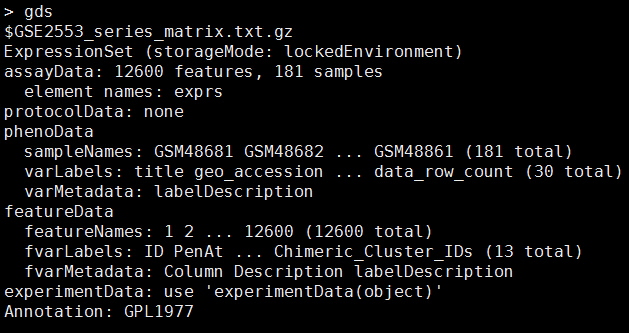
http://www.bio-info-trainee.com/ ... tware/GEOquery.html
http://www.bio-info-trainee.com/ ... are/limma_voom.html
当然,一些并不是bioconductor的包我也会写教程, 偶尔:
http://www.bio-info-trainee.com/ ... oftware/GOplot.html
http://www.bio-info-trainee.com/ ... ftware/Rcircos.html
http://www.bio-info-trainee.com/ ... software/limma.html
http://www.bio-info-trainee.com/ ... oftware/DESeq2.html
http://www.bio-info-trainee.com/ ... tware/GEOquery.html
http://www.bio-info-trainee.com/ ... are/limma_voom.html
当然,一些并不是bioconductor的包我也会写教程, 偶尔:
http://www.bio-info-trainee.com/ ... oftware/GOplot.html
http://www.bio-info-trainee.com/ ... ftware/Rcircos.html