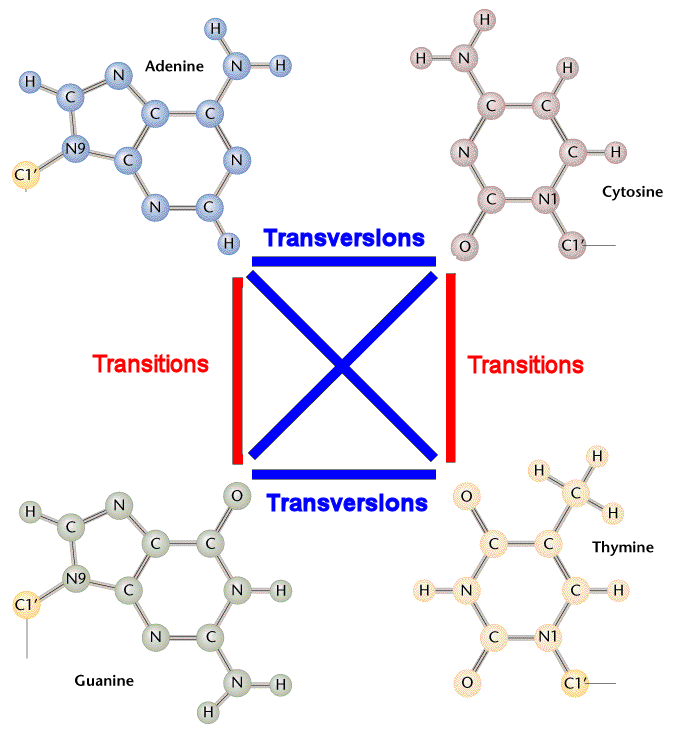
DNA分子中某一个碱基为另一种碱基置换,导致DNA碱基序列异常,是基因突变的一种类型。可分为转换和颠换两类。转换(transitions)是同类碱基的置换(AT→GC及GC→AT),颠换(transversions) 是不同类碱基的置换(AT→TA或CG,GC→CG或TA。
DNA substitution mutations are of two types. Transitions are interchanges of two-ring purines (A G) or of one-ring pyrimidines (C T): they therefore involve bases of similar shape. Transversions are interchanges of purine for pyrimidine bases, which therefore involve exchange of one-ring and two-ring structures.
我们在分析driver mutation的时候会区分各种点突变:
- 1. CpG transitions
- 2. CpG transversions
- 3. C:G transitions
- 4. C:G transversions
- 5. A:T transitions
- 6. A:T transversions
那么,我们有64种密码子,每种密码子都会有9种突变可能,我们如何得到一个所有的突变可能的分类并且打分表格呢?
类似于下面这样的表格:共576行!!!
head category.acgt
AAA>AAT 2 A T 6
AAA>AAC 2 A C 6
AAA>AAG 2 A G 5
AAA>ATA 1 A T 6
AAA>ACA 1 A C 6
AAA>AGA 1 A G 5
AAA>TAA 0 A T 6
AAA>CAA 0 A C 6
AAA>GAA 0 A G 5
AAT>AAA 2 T A 6
tail category.acgt
GGC>GGG 2 C G 2
GGG>AGG 0 G A 3
GGG>TGG 0 G T 4
GGG>CGG 0 G C 4
GGG>GAG 1 G A 3
GGG>GTG 1 G T 4
GGG>GCG 1 G C 4
GGG>GGA 2 G A 3
GGG>GGT 2 G T 4
GGG>GGC 2 G C 4
我本来以为这是一件很简单的事情,写起来,才发现好麻烦
里面用到的一个函数如下:就是判断突变属于上述六种的哪一种!
参考:https://www.mun.ca/biology/scarr/Transitions_vs_Transversions.html
https://en.wikipedia.org/wiki/Transversion
http://www.uvm.edu/~cgep/Education/Mutations.html
点突变,也称作单碱基替换(single base substitution),指由单个碱基改变发生的突变。
可以分为转换(transitions)和颠换(transversions)两类。
转换:嘌呤和嘌呤之间的替换,或嘧啶和嘧啶之间的替换。
颠换:嘌呤和嘧啶之间的替换。
方便理解下面再附上一张示意图,如下: