学员的微信交流群有小伙伴提到了一个单细胞转录组数据挖掘文章:《Single cell RNA-seq data and bulk gene profiles reveal a novel signature of disease progression in multiple myeloma》,Zeng et al. Cancer Cell Int (2021) ,有意思的是文献重新分析的数据集是 GSE118900,Multiple myeloma(多发性骨髓瘤)是一种影响血液的癌症,主要涉及浆细胞(plasma cells)。浆细胞是一种白血细胞,负责产生抗体,这些抗体帮助身体对抗感染和疾病。在多发性骨髓瘤中,骨髓内的浆细胞会发生异常,变得异常增生并形成肿瘤。这种异常的浆细胞,称为骨髓瘤细胞,它们会取代骨髓中正常的血细胞,导致以下问题: Continue reading
Author Archives: ulwvfje
为什么要添加我的微信好友(今年这形势,劝大家还是多做一手准备吧!)
早些日子我在《生信技能树》发布了 我应该不需要你来教我如何赚钱 的吐槽小短文,那个时候微信通讯录其实限制的是5000好友数量,时至今日这个上限提高到了1万人, 但是我的微信仍然是满员了。非常尴尬,所以我前些天又删除了三千多人,算起来是可以再撑个两三年啦。 Continue reading
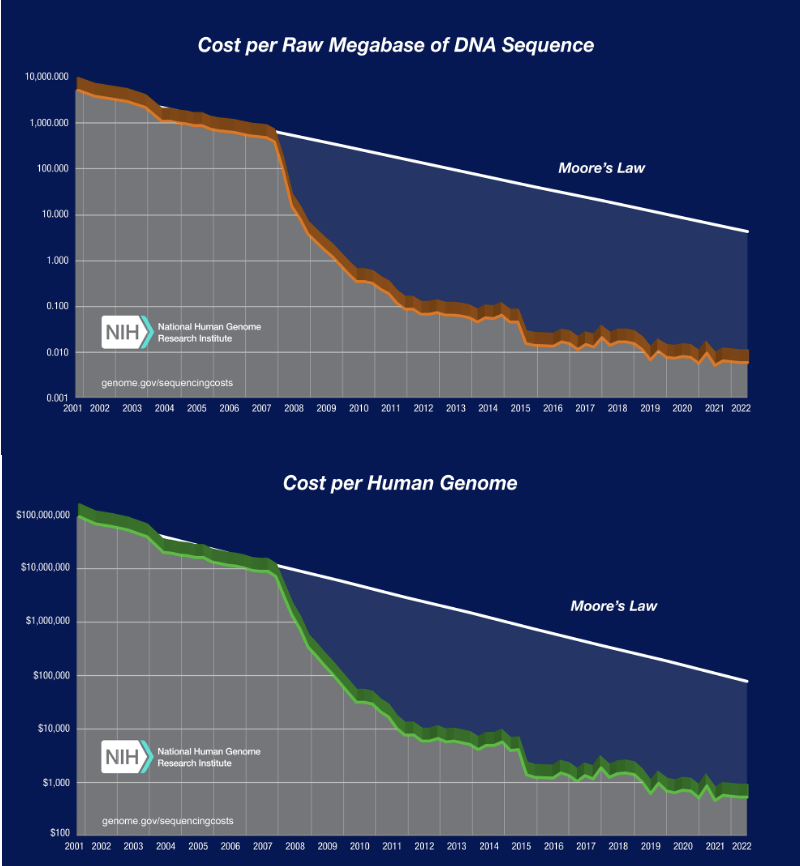
为什么都在卷转录组测序的价格呢
通篇文章没有测序数据你敢信?
看到了一个新闻:贵州茅台改善酒精性肝纤维化的文章被撤回。
我才注意到居然有这样的研究:2020年6月15日,Food Science & Nutrition 在线发表题为“Kweichow Moutai ameliorates alcohol-induced liver fibrosis in mice by targeting the NFκB pathway”的研究论文,该研究表明茅台可诱导肝星状细胞(HSC)凋亡,抑制胶原沉积,减轻肝纤维化。我简单的看了看文献的pdf,都是一些实验结果,最简单的测序都没有,我看都懒得看,就让人工智能大模型帮我简单的读了一下: Continue reading
数据框的列名居然会影响绘图
我们的生物信息学马拉松授课非常注重于编程基础,一般来说完成了两周的r编程语言的互动式授课之后我仍然是会建议大家完成我的b站视频学习和对应的100个r练习题; Continue reading
使用人工智能优化一个数据库文件批量下载脚本
学完了生信技能树的马拉松授课,算是掌握了跟人工智能对话的基础,比如前些日子看到的练习题:PDX小鼠模型的单细胞样品定量能选择人类参考基因组吗,是需要下载这个CRA010501数据集里面的单细胞转录组样品的fq文件,然后走cellranger定量流程,选择人类以及小鼠参考基因组,各自走一遍定量流程,然后两个表达量矩阵分开做一下降维聚类分群哈, 看看背后是否有一些被忽略的生物学现象。
需要使用的数据库文件批量下载脚本是 : Continue reading
十多万个细胞数量就顶不住了吗
最近有小伙伴使用我们的共享服务器(2024的共享服务器交个朋友福利价仍然是800)后“抱怨”到居然读取一个单细胞转录组公共数据集,就十多万个细胞数量而已,就顶不住了,如下所示的文件 : Continue reading
生信数据分析可靠与否是一个伪命题
多种场合我都是高举生物信息学数据分析的靠谱的大旗,详见前面的推文:离大谱了,生信转湿实验?。仍然每次跟网友们的讨论的结局都是一地鸡毛,大家总会是把生物信息学数据分析跟常见的临床数据挖掘预后模型那些水文混为一谈。 Continue reading
生信人该如何找工作
众所周知,现在(2024年)是经济下行现阶段,基本上就很少有公司不裁员,更别说主动招聘。而且最近三五年的硕博士扩招又非常严重,进一步造就了 “僧多粥少”的地狱级别就业难度! Continue reading
生信工程师如何刻意体验完整的商业闭环
接触过一些各地科研服务公司的ngs产品销售,因为他们是面向各个课题组的一线接触人员,所以经常会遇到有课题组老师询问生物信息学数据处理相关的问题,所以销售小伙伴有职业困惑就是是否该花时间学一学简单的数据处理技能呢,比如使用R语言画个图,清洗一下数据等等。 Continue reading
生信工程师可以有意识的培养自己的老板思维
打工思维通常指的是一种被动的工作态度,员工仅仅完成分配的任务,而不关心公司的整体发展。这种思维模式可能会限制个人的职业发展,因为它缺乏主动性和创新性。
相反,具备老板思维的员工更能够主动承担责任,展现出领导潜力,从而在职场上获得更多的机会和更好的发展。
生信工程师可以有意识地培养自己的老板思维,主要是因为具备这种思维能够帮助他们在职业生涯中更好地理解业务、提高解决问题的能力、增强领导力和团队协作能力,从而提升个人竞争力和市场价值。以下是一些原因和建议: Continue reading
生信工程师都值得体验的小成本创业模式
曾经年少轻狂的你,可能有一个创业梦,作为生信工程师的我们也不例外。
但是大家如果不是在2010以及2017这两个关键的年份创业就做科研服务,就很难再开始了。更何况,作为普通生信工程师的你肯定是没有大资金,没办法组建团队,分工合作,这个时候你可以试试看这个小成本创业模式:经营好自媒体并且尝试线上接单: Continue reading
生物信息学的未来十年的趋势展望
2023年7月,第十届“数学、计算机与生命科学交叉研究”青年学者论坛在北京举行,来自数学、计算机与生命科学交叉领域的院士专家、优秀中青年学者与广大学生参加会议,共同交流生物信息学的前沿进展,展望生物信息学科的发展未来。与会专家们经过讨论, 确定了生物信息学的未来十年预计将由以下几个关键趋势和挑战定义: Continue reading
如何批量解压当前文件夹下面的全部压缩包
学员提问了一个小鼠的单细胞数据集,GSE232481,研究者公开了表达量矩阵文件,但是文件压缩包非常奇怪: Continue reading
如何理解shell脚本运行时候的标准输出和标准错误的信息
我们的生物信息学马拉松授课里面的转录组实战环节介绍了批量一次性的针对全部的样品运行一个shell脚本的实战演练,代码如下所示: Continue reading
如何加速一个简单的R代码绘制箱线图
在进行人工智能大模型的答疑的时候,演示了一个简单的表达量芯片数据集:GSE66175,但是它的样品数量确实是有点多,所以一个简单的R代码绘制箱线图都耗时很久,如下所示:
```r Continue reading
如何看一堆基因在各个单细胞亚群是否有差异呢?
安排学徒复现一个新鲜出炉的阿兹海默症的单细胞文章:《Characterisation of premature cell senescence in Alzheimer’s disease using single nuclear transcriptomics》: Continue reading
如何让更多人宣传你的科研成果
人工智能大模型也不能挽救一个破碎的文件啊(脊髓,神经单细胞标准数据集)
答读者问的时候,我演示了一下使用人工智能大模型来试图解决R文件读取的问题(2022-GSE172167-脊髓损伤),可以看到作者给出来了如下所示的文件 : Continue reading
人工智能大模型的好处之提取基因名字
2017的一个文章:《Meta-signature of human endometrial receptivity: a meta- analysis and validation study of transcriptomic biomarkers》 做了很多独立的公共数据集里面的表达量矩阵分组后的差异分析,然后使用rra整合算法拿到了可靠的上下调基因,但是隐藏在下面的图里面: Continue reading