发表在Nat Med. 2017 April的文章HRDetect is a predictor of BRCA1 and BRCA2 deficiency based on mutational signatures ,其研究发现了一种新的类似于BRCA的突变特征,普遍存在于约20%的乳腺癌患者中,具有这种突变的乳腺癌患者或许同样能从PARP抑制剂靶向治疗中受益。并不需要一定是BRCA1/2基因本身的突变。
这样就从理论基础上面极大的扩充了从PARP抑制剂靶向范围,临床意义深远。
背景介绍
大约1-5%的乳腺癌归因于BRCA1或BRCA2的遗传突变,并选择性敏感于(ADP-核糖)聚合酶(PARP)抑制剂;
BRCA1/2为抑癌基因,参与同源重组缺陷(homologous recombination deficiency, HRD)修复,具有BRCA1/2突变者其乳腺癌、卵巢癌的发病风险显著提高。
同源重组(HR)通路是涉及多种蛋白相互作用的复杂过程,随着基因检测技术的发展已经鉴定多个相关基因,临床上多基因panel检测除包括中高外显基因PALB2、ATM和CHEK2、BARD1、RAD51D,还涉及一些尚未明确增加乳腺癌风险的HR相关基因如NBN、MRE11A、RAD50、RAD51C。
携带BRCA1/2突变或其他导致HRD的突变的肿瘤对PARP(Poly (ADP-ribose) polymerase)抑制剂尤其敏感,疗效可用“合成致死”效应来解释。那么,何为“合成致死”呢?指当基因A功能缺失时,细胞依赖B基因生存,此时抑制B基因将导致细胞死亡,单独的A基因功能缺失或单独的B基因抑制细胞仍可正常存活。
在2016年,有 560 个BRCA 病人的WGS数据发布,本文研究就大篇幅重新复用了这个数据集。
在2018年,有一个全局筛查BRCA1基因所有SNV是否影响细胞生存的文章,我在文献阅读年计划里面分享过:https://zhuanlan.zhihu.com/p/49563795
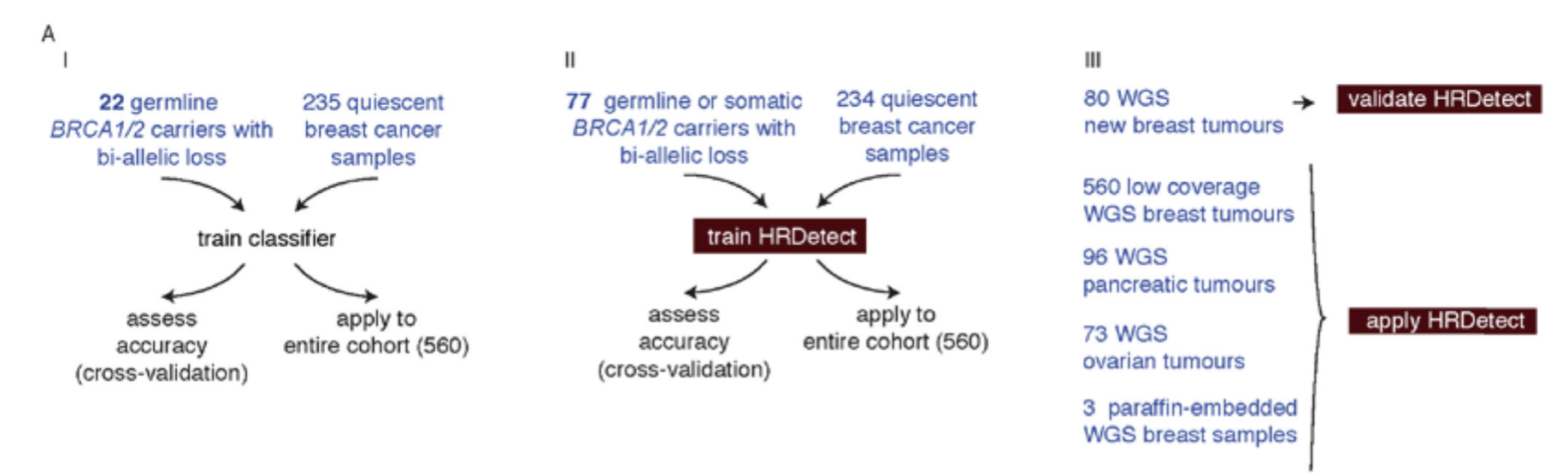
首先得到HRDetect基因集
这个560 个BRCA 病人的WGS数据集里面,24个带有BRCA1 (5) and BRCA2 (19)突变,其中22个很特殊

使用 supervised learning lasso logistic regression model 和 An iterative ten-fold nested cross-validation strategy 最大化的得到这22个病人,也就是BRCA1/BRCA2-deficient cancers 和 训练集的 235 sporadic breast cancers 的差异,包括5个参数:
- microhomology-mediated indels
- the HRD index
- base substitution Signature 3
- Rearrangement Signature 3
- Rearrangement Signature 5
在另外一个数据集验证:a larger, better-powered training set comprising 77 samples (22 known germline, 33 new germline diagnoses, 22 somatic)
最后再回到560 个BRCA 病人的WGS数据测试,HRDetect评分曲线依据BRCA1/2缺陷可能性呈显著的S型分布,不仅识别出已知的22名BRCA阳性患者,还在剩余538个肿瘤样本中识别出90个BRCA缺陷(区分BRCA缺陷与否的阈值设定为0.7)的患者,预测BRCA高水平缺陷型的比例高达20%。
这3个分析流程图如下:
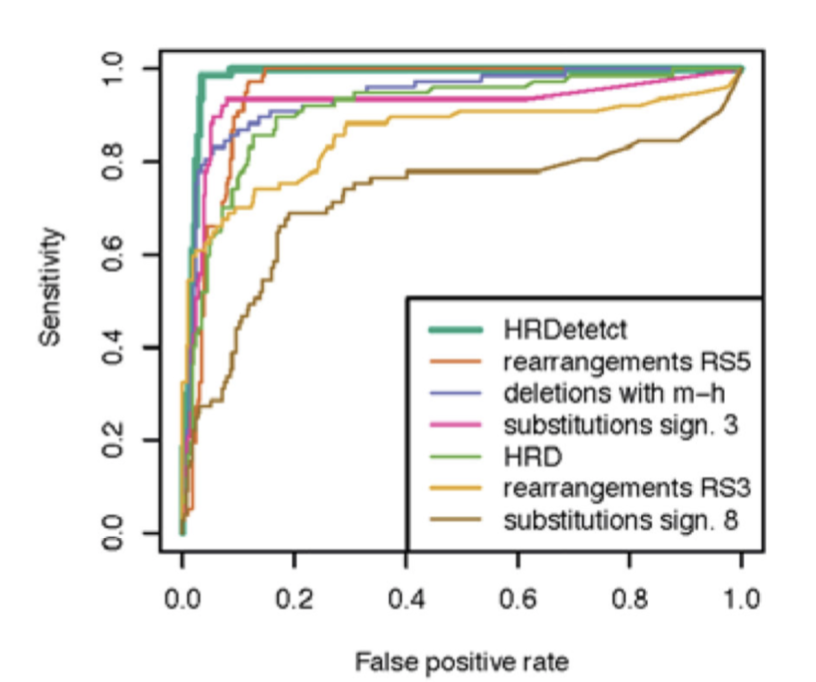
HRDetect was re-applied on the cohort of 560 breast cancers and showed excellent performance as revealed by a receiver operating characteristic (ROC) curve with an area under the ROC curve (AUC) of 0.98
如下所示:
这个算法依赖于WGS数据,这样临床应用就大打折扣。计算(HRD) index score
这个 homologous recombination deficiency(HRD) index score 如下图,可以看到 非常明显的把560个WGS测序的乳腺癌患者区分开来了
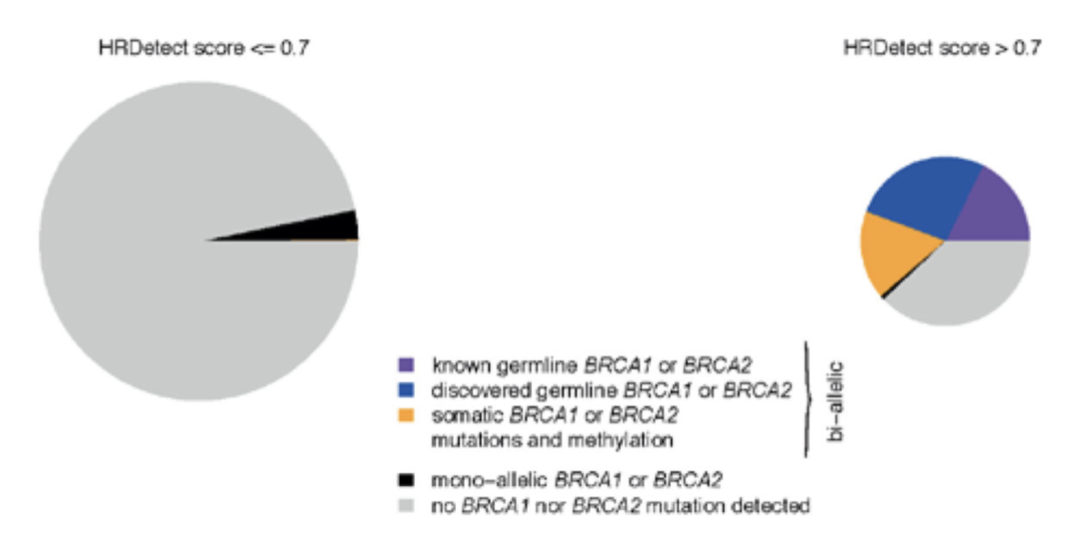
显著增加了 BRCA1/BRCA2 deficient 病人
如果仅仅是从germline BRCA1/BRCA2 null 突变角度来看这560个WGS测序的乳腺癌患者,就22个病人是符合PARP抑制剂靶向范围,但是如果使用研究者定义的HRDetect reveals an additional 33 germline, 22 somatic and 47 tumours where no mutation was detected
这样,受益的患者由22扩充到了124 (22%) BRCA1/BRCA2 deficient tumours.其它数据集验证
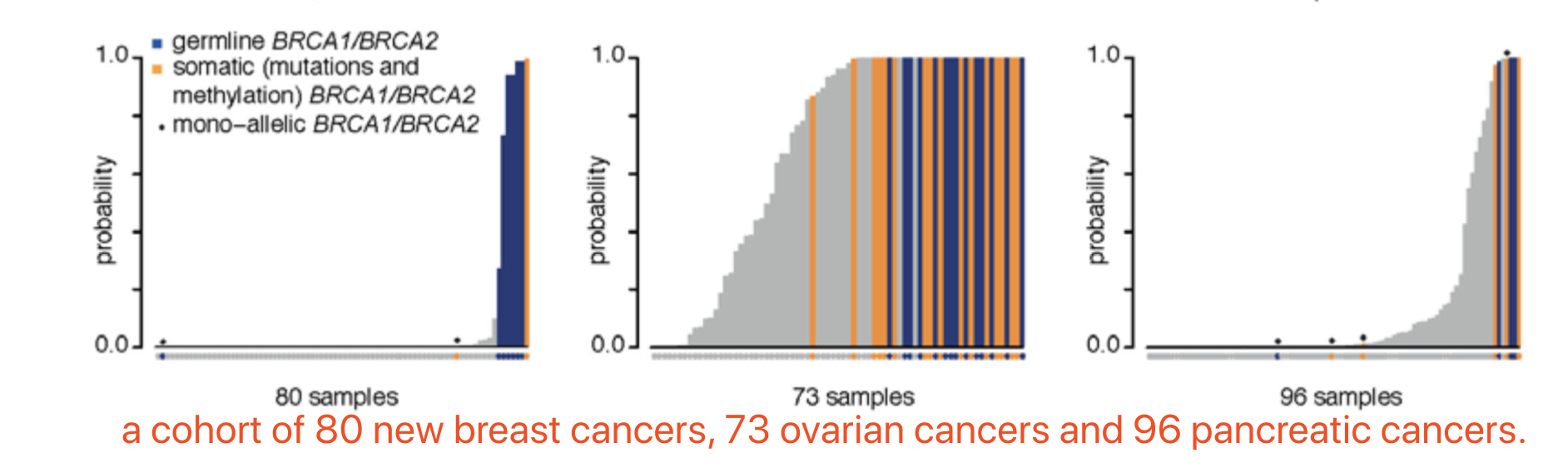
HRDetect was applied to a new cohort of WGS breast cancers from 80 women (Figure 4, Supplementary Table 5) with mainly ER positive, HER2 negative breast cancer as a validation exercise. HRDetect successfully identified 1 germline BRCA1 and 5 BRCA2 mutation carriers (4 germline and 1 somatic) with associated loss of the wild type allele.
在The ovarian cancer cohort comprised 73 samples.数据集表现优异,HRDetect has a sensitivity of detecting BRCA1/BRCA2 null cancers approaching 100% and has uncovered 16 additional patients as HR deficient.
在The pancreatic cancer cohort comprised 96 samples.数据集同样表现优异,Thus, HRDetect had a sensitivity approaching 100% in this pancreatic cancer cohort and identified 5 additional patients with potential HR deficiency.
如下图:
低深度测序数据验证
随机抽样把原始的高深度WGS数据 (30- to 40-fold) 模拟为低深度 (10-fold, range 9.9 to 10.5) WGS 数据继续检测 HRDetect的表现。
如下所示:
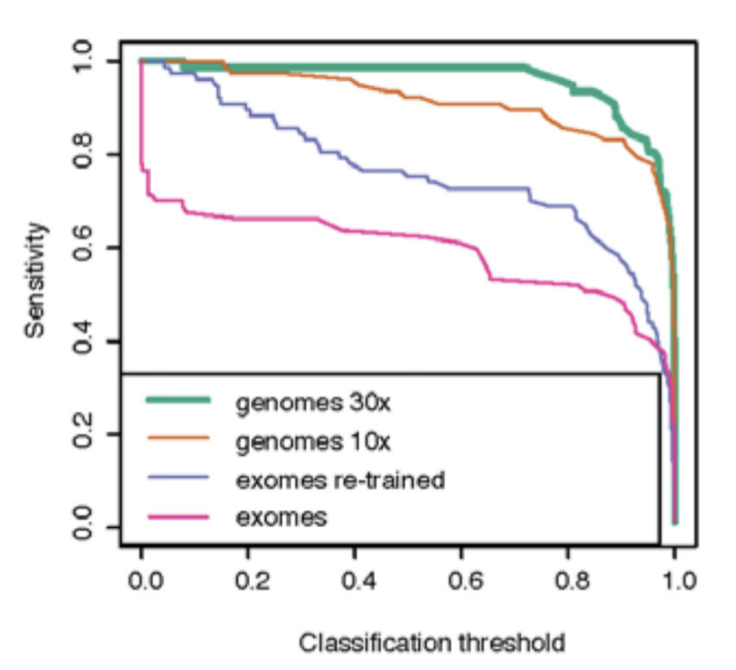
FFPE样本数据验证
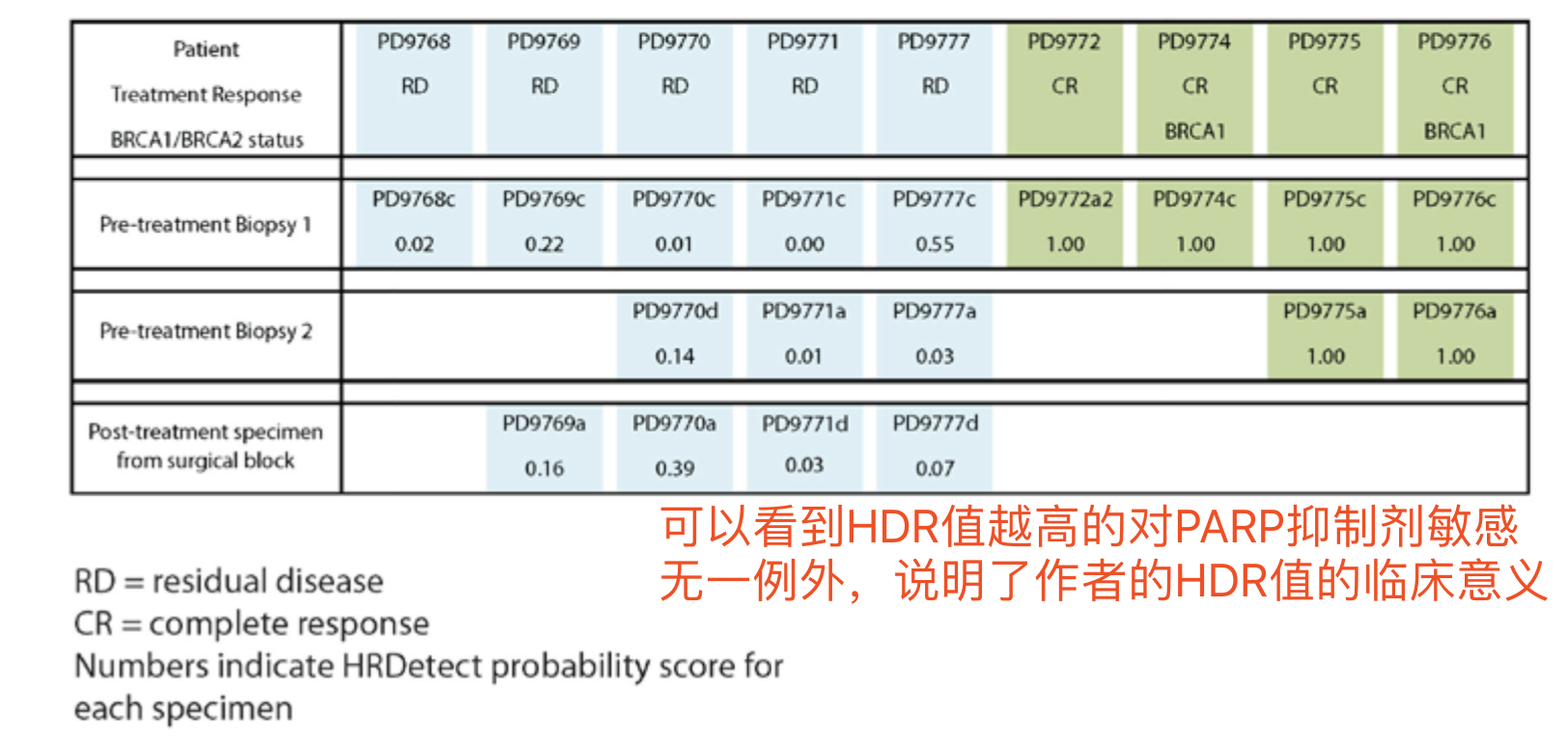
因为大量医院的癌症患者的肿瘤部位其实是FFPE形式,所以有必要测试研究者的HRDetect基因集在那样的数据里面的表现形式,作者从9个TNBC患者提取了18个DNA样品进行WGS测序,包括(14 needle
biopsies and four post-operative tumour block specimens) ,他们这些病人都是接受了PARP抑制剂的治疗,也说明了研究者提出的HRDetect基因集的有效性。
总结
HRDetect识别BRCA1/2类似突变特征的灵敏度高达99%,而且该模型同样适用于来自穿刺活检的小样品和手术期间取出的肿瘤大样品中。这表明该模型可以在临床早期第一次进行活检时就开始使用,有助于指导癌症患者的早期治疗。
识别了一系列可能对PARP抑制剂有响应的乳腺癌患者。为了将这些结果推广到临床治疗上,还需要进一步开展癌症基因组测序和临床试验。”