我以为我写完了R包终极解决方案! 之后,应该不会再有任何关于R包安装的问题产生了,但仔细回过头来看才发现,我介绍的都是如何从CRAN或者bioconductor里面安装正规发布的包,但是有很多人开发的是自己私人的包,而我们有的确非常需要用怎么办??这个时候就需要下载别人开发的包来安装了。比如我R包地址见github:https://github.com/jmzeng1314/humanid
首先你必须确定这个包是干净的,没有危险,然后要确定你的确需要这个包,因为大多数是时候你其实只需要他包里面一个函数即可。如果确定需要安装,就安装一个git软件吧,然后git clone https://github.com/jmzeng1314/humanid.git 这样就把这个R包下载到了自己指定的目录,或者如果你懒得安装git软件,直接在github网页里面下载成zip格式的压缩包也行。
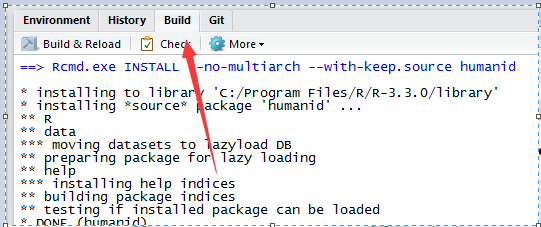
下载的R包里面有一个.Rproj后缀的文件,可以自己双击打开,在Rstudio里面就可以点击build安装这个R包了,如图:
安装完毕后就会自动加载这个包,然后就可以看到它里面的各种函数和数据的!你已经成功的接收了别人的代码啦!
那么是不是安装好了这个包,你就可以使用它了呢?其实不然,如果是已经发表的正规的包,一般会写好完全的依赖关系,所以在你安装过程中,会提示你不停地安装各种包,但是我的包没有,只有在你运行我但是的时候,我才会报错,告诉你你需要安装某某包!的确有点傻,因为我懒得去写依赖关系,或者说,我还没有学到!
> keggAnno()
Show Traceback
Rerun with Debug
Error in loadNamespace(name) : there is no package called ‘DT’
>
这样做其实也有好处,你无论如何都是可以把我的包安装上的,虽然你可能安装上了也无法使用。
> install.packages('DT')
trying URL 'https://cran.rstudio.com/bin/windows/contrib/3.3/DT_0.2.zip'
Content type 'application/zip' length 950203 bytes (927 KB)
downloaded 927 KB
package ‘DT’ successfully unpacked and MD5 sums checked
The downloaded binary packages are in
C:\Users\jimmy\AppData\Local\Temp\RtmpoT4VWl\downloaded_packages
>
很容易安装好了DT这个包,我的函数就可以使用啦!keggAnno()这个函数默认运行成功是没有提示的,但是你可以查看你当前目录,的确多了一个kegg注释文件,可以把你感兴趣的基因批量注释到KEGG数据库。