这是TCGA数据挖掘系列文章之一,是安德森癌症研究中心的Han Liang主导的,纯粹的生物信息学数据分析文章。
文章题目是:comprehensive characterization of molecular differences in cancer between male and female patients.
研究意义:癌症病人的性别对肿瘤发生,扩散的意义不言而喻。不仅仅是因为很多癌症本来就是有性别特异性,比如卵巢癌之于女性、前列腺癌之于男性。即使对于其它并非性别特异性的癌症种类,男女病人在肿瘤发生,扩散,以及治疗阶段的反应也大不一样。但是以前对这样分子机理研究的很有限,一般集中在某些性别相关的分子pattern,比如非小细胞肺癌女性患者的EGFR突变,但那些研究要么就局限于单一的基因,要么局限于单一的数据类型,或者研究单一的癌症。严重缺乏一个全面的,系统的分析癌症患者的性别差异。而且TCGA数据库的出现让这一个研究变成了可能,这也就是本文章的出现的原因。
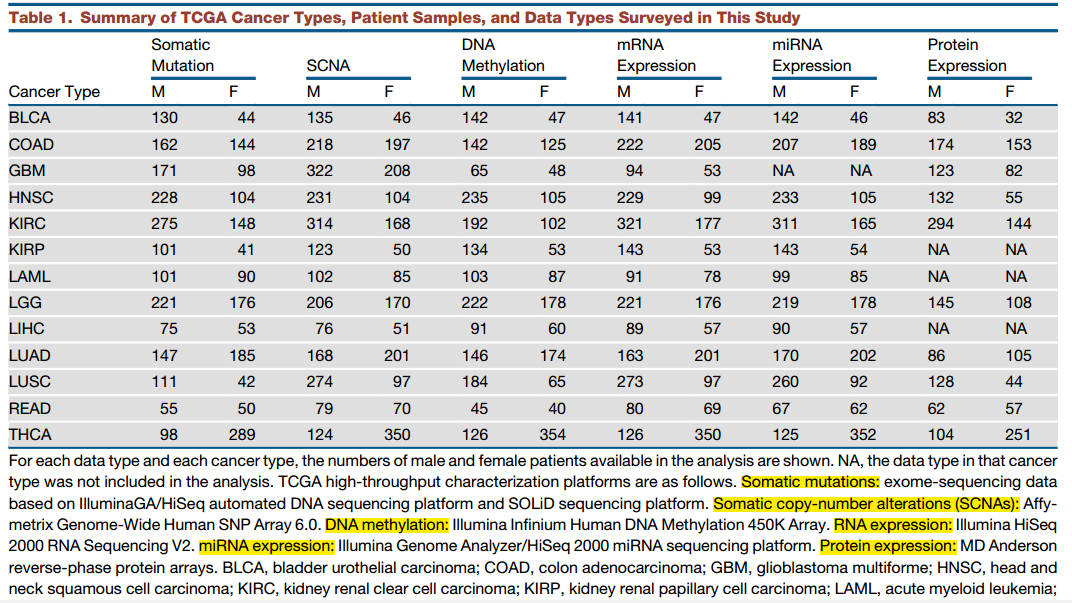
数据挖掘的对象:
如表所示,涉及到13种癌症,TCGA的六种数据()都用上了,因为是2016年,所以数据量也比较全面了。
还有他们的临床信息,也结合起来分析,具体样本个数,以及癌症种类分布见下表。
六种数据分别是:
全外显子组的somatic突变数据,
affymetrix的snp6.0芯片的拷贝数变异数据,
人甲基化450K芯片的DNA甲基化数据,
RNA-seq的mRNA表达量数据,
miRNA的表达量数据,
蛋白表达数据。
文章对这些数据做了6个方面的分析:
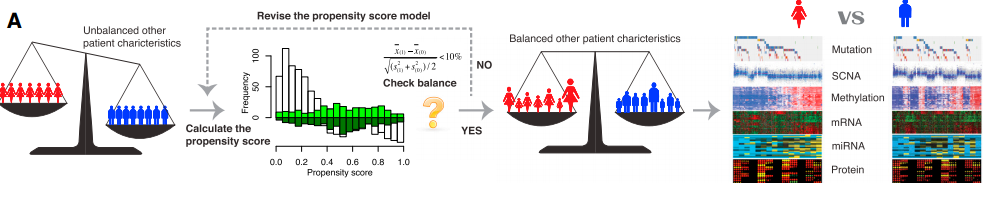
一是对各个样本进行权重矫正
这个偏统计学了,大家可以自己去看原理,主要是为了排除除性别外的其它影响因素( sex, age at diagnosis, smoking status, tumor stage, and histology subtype),采用了一种叫做propensity score.的统计学方法来矫正这些共影响因子,这一统计方法是上世纪80年代提出了的,被广泛应用于clinical research, economics, and social sciences。
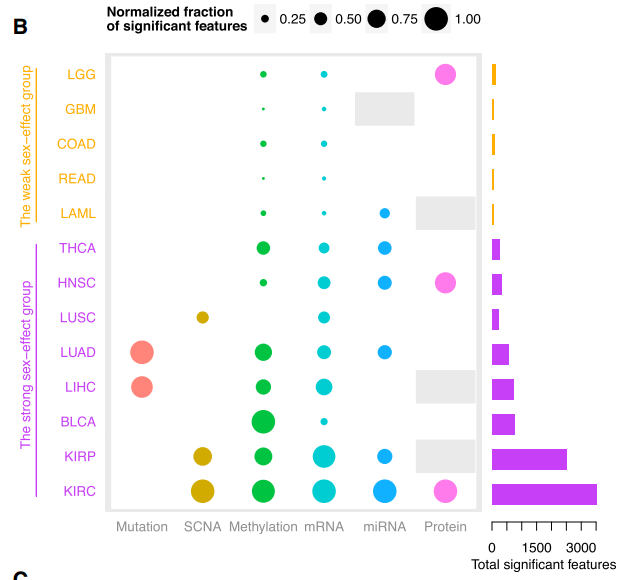
二是用六种数据结合起来把癌症根据性别影响分成两类
其中一类受性别影响较弱,是LGG, GBM, COAD, READ, and LAML
另一类受性别影响较强,包括THCA, HNSC, LUSC, LUAD, LIHC, BLCA, KIRP, and KIRC
并且提出一个sex-bias index 的概念来描述他们的差异 defined on the basis of the ratio of new cases of female and male patients
受性别影响较弱那几个癌种的男性与女性患者比较起来差异特征很少(44–104, mean 67)
受性别影响较弱那几个癌种的男性与女性患者比较起来差异特征很少(44–104, mean 67)
而受性别影响较强那几个癌种的男性与女性患者比较起来差异特征很多(240–3,521, mean 1,112)
看下面的图可知,这两组差异非常显著。而定义的差异特征是非常重要的概念,对6种数据,差异特征都不一样,下面会具体讲到。
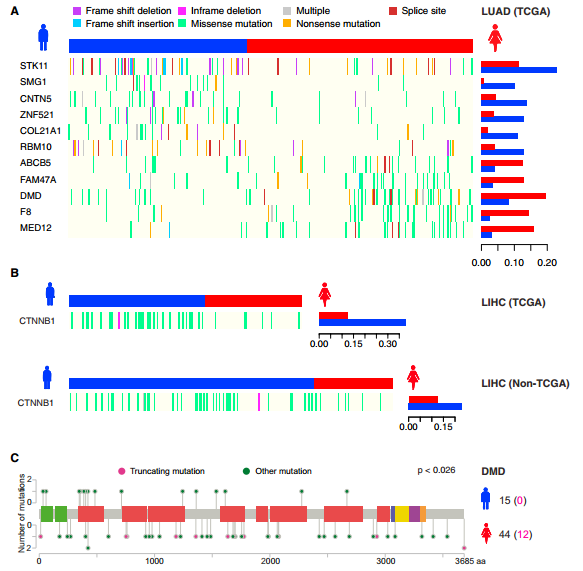
三是单独拿somatic mutation数据来分析
作者是直接从Firehose (http://gdac.broadinstitute.org) 里面下载了所有的上面列出的样本的MAF突变数据,一般TCGA记录的MAF突变数据就是他们已经分析好的somatic mutation数据。作者只分析了non-silent mutations,只考虑那些突变频率(基于这个文章的群体)大于5%的位点,而且去掉了somatic mutation个数超过1000的个体,男女之间用费谢尔精确检验来计算差异显著度。
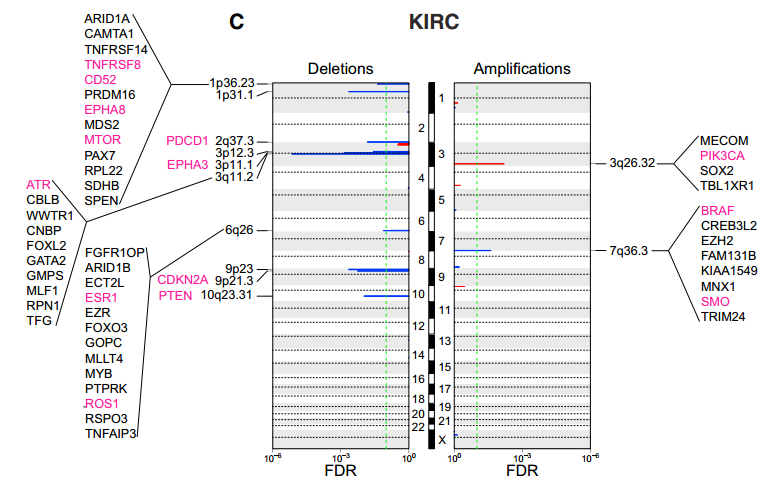
四是单独拿somatic的CNV数据来分析
这个分析也很简单,还是直接从Firehose (http://gdac.broadinstitute.org) 里面下载了所有的上面列出的样本的CNV数据,然后每个癌种都分男女分别跑一下GISTIC这个软件,得到somatic的拷贝数变异数据库,GISTIC软件是基于matlab的,在我的博客有详细介绍该软件如何使用。
把GISTIC的结果,包括focal and arm-level amplifications/deletions都进行了信息的生物学解释,哪些基因很重要,哪些通路很重要,都详细的描述了,这个需要作者具有渊博的生物学背景知识,而不是数据分析技巧了。
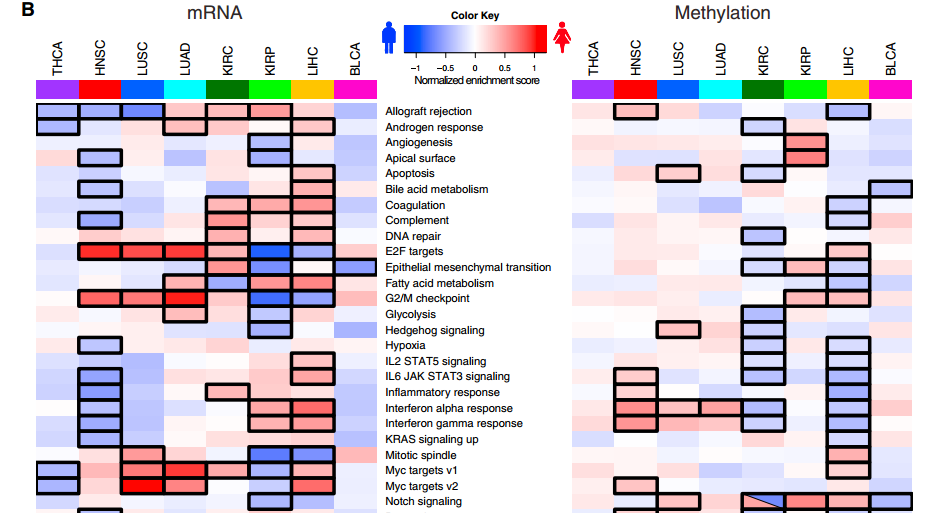
五是结合4种表达量数据来分析
分析完突变数据,然后开始分析表达数据,作者把4种表达量数据综合起来分析了,包括甲基化位点表达数据,mRNA,miRNA和蛋白的表达数据。前两个是从TCGA data portal里面下载的,后两个是从Firehose里面下载的。
其中mRNA表达数据,基于RSEM的表达值,分析表达数据差异的时候,还做了GSEA分析。
也研究了miRNA调控,用miRTarBase数据库来验证miRNA的target,或者通过TargetScan, miRanda and miRDB 数据库来预测
表达数据一般用热图来可视化,然后重点讲几个通路,为什么在癌症这么相关呢?为什么男女差异这么大呢?等等
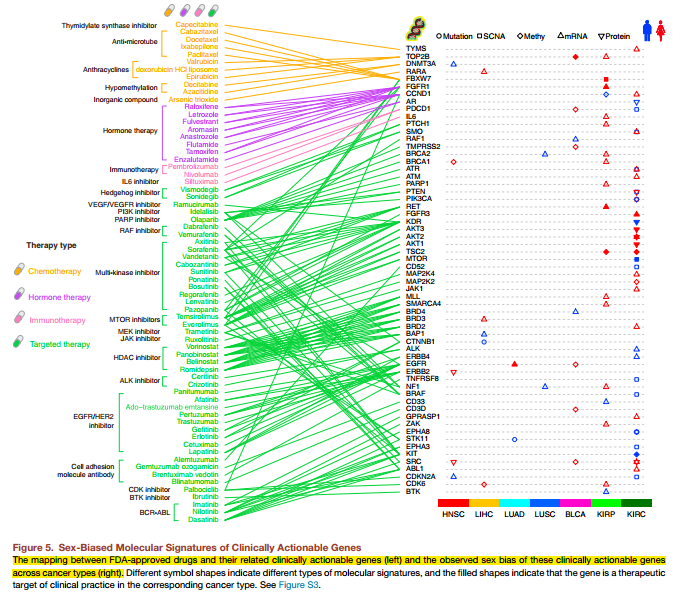
六是根据自己的分组来探索一些临床指标以及药物可能的影响。
这个算是本文比较新颖的地方了,作者从FDA批准的一些癌症相关药物里面找到了这些药物作用的基因,然后把这些基因跟有性别差异的基因进行交叉比较。
这个研究意义非凡,因为现在对癌症病人用药都是一视同仁,不会考虑到性别的差异,而我们的分析恰恰证明了癌症患者的性别差异还是蛮大的,为了更好的治疗,这些必须考虑进去。比如SRC这个基因在HNSC这个癌症患者里面,女性比男性显著高表达。
下面这个高大上的图说明了一切,但想真搞明白,不是一天两天的事情。