有小伙伴在交流群问:自己的目标基因在做完表达量差异分析后发现它并不在上下调列表里面,感觉有点惶恐,如果是真实现象,那么:
- 基因表达量无显著差异:这是最直接的可能性。在你研究的条件或处理下,该基因的表达量可能并未发生显著改变。
- 样本差异大:如果你的样本间的差异过大,可能会影响到差异表达分析的结果。例如,如果一部分样本中该基因表达量上调,而另一部分样本中该基因表,所以导致数据分析结果是无差异。
实际上,绝大部分情况下,仅仅是因为我们对数据的了解不够。这样的疑惑很容易发生在没有太多数据处理经验的初学者身上,他们过渡依赖于标准流程和“师兄师姐”传递下来的代码,或者说太依赖各种参数和阈值,不敢大胆的反抗软件分析的结果。。。。
比如2019年Science Advances上发表的一项新研究,标题是:《ITGA5 inhibition in pancreatic stellate cells attenuates desmoplasia and potentiates efficacy of chemotherapy in pancreatic cancer》初步得到了整合素α5 (ITGA5)在胰腺癌发挥癌基因的作用,也就是说它在癌症里面相对于癌旁来说是恶性高表达的,而且表达量越高病人预后就越差。。。
可以看到,这个研究的作者为了说明整合素α5 (ITGA5)在胰腺癌发挥癌基因的作用,使用了生存分析图加上表达量箱线图,而且都达到了统计学显著性。
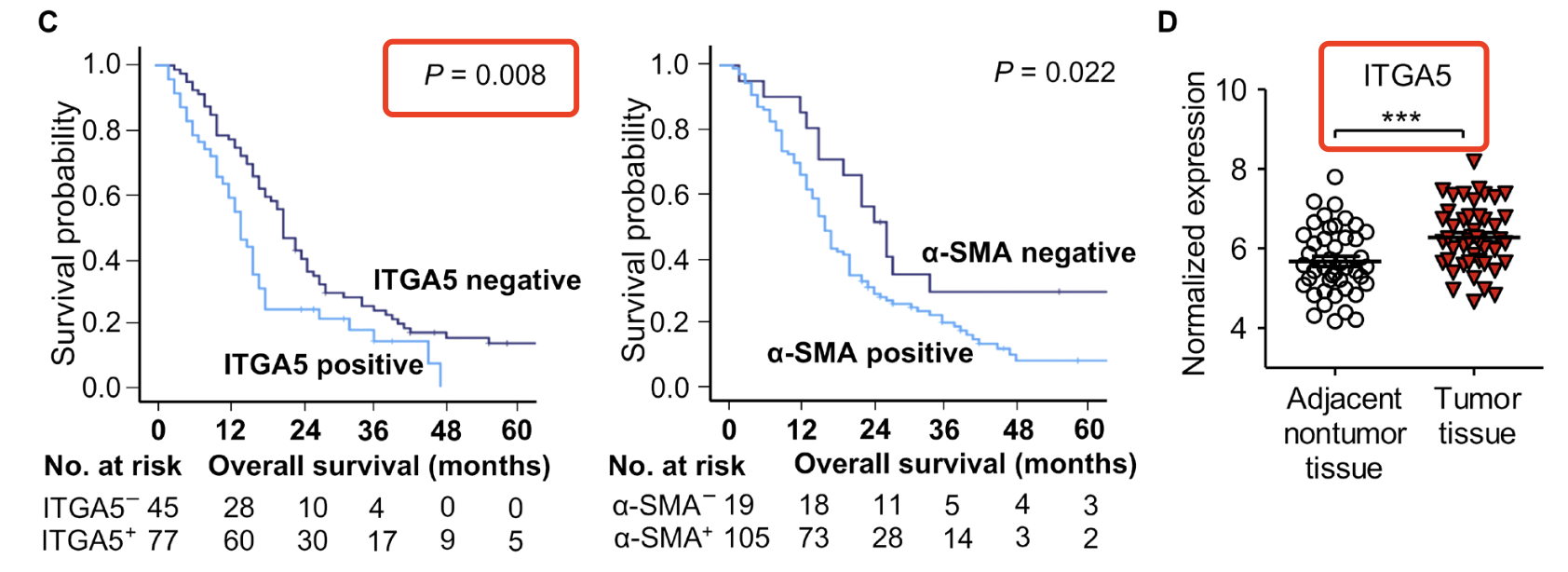
这样无可厚非,毕竟是为了发文章, 肯定是得凑阳性结果啦。
但是如果你只细看表达量,其实是专注于研究肿瘤微环境的Jai Prakash教授,带领研究团队检查了约140位胰腺癌患者的组织样本,分析预后差异和肿瘤整合素α5 (ITGA5)的蛋白表达量阳性与否分组后的生存分析。你必须首先相信他们团队收集整理的病人信息是ok的,其次你得相信他们的组织芯片是ok的,然后大多数情况下,这两个环节的可能的错误经常会发生。
其次,那个表达量箱线图是 (D) Transcriptomic analysis of ITGA5 in publicly available microarray dataset (GSE28735). 如果你去处理 GSE28735 这个表达量芯片数据集,参考我们的代码:
- 结肠腺癌细胞系过表达apoM的芯片数据分析
- 大鼠表达量芯片数据处理
- 让天下没有难处理的表达量芯片
- TNBC数据分析-GSE27447-GPL6244
- TNBC数据分析-GSE76275-GPL570
- mmu-macrophages-GSE69607-GPL1261
- HNSCC数据分析-GSE6631-HG_U95Av2-GPL8300
- HNSCC数据分析-GSE13399-GPL7540
- HNSCC数据分析-GSE33205- GPL5175
- HNSCC数据分析-GSE107591-GPL6244
- HNSCC数据分析-GSE2379-GPL830-GPL91
- ccRCC数据分析-GSE53757-GPL570
- ccRCC数据分析-GSE14672-GPL4866
- ccRCC数据分析-GSE66270-GPL570
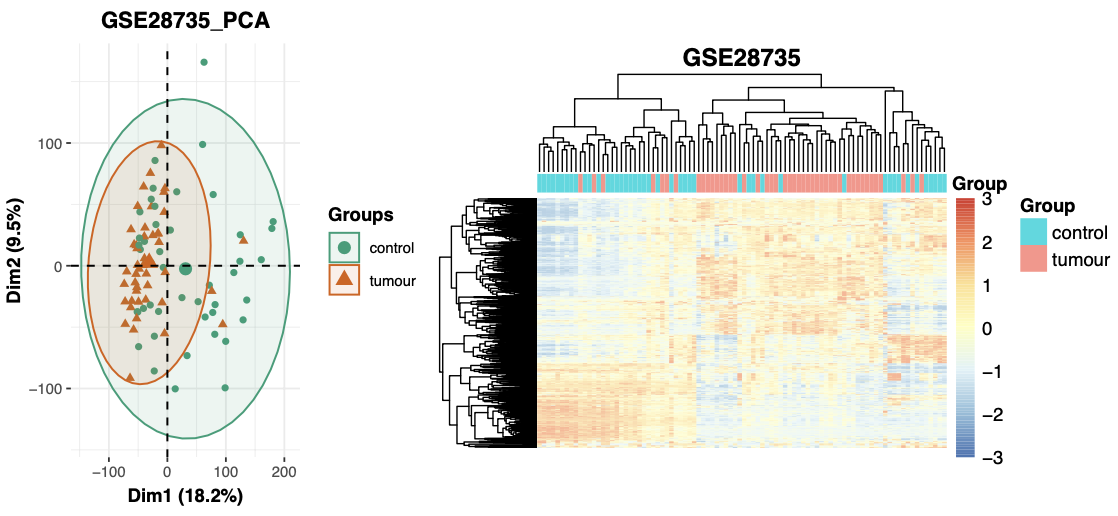
很容易看到, GSE28735 这个表达量芯片数据集质量很好,是 We compared gene expression profile of 45 pairs of pancreatic tumor and adjacent non-tumor tissues using Affymetrix GeneChip Human Gene 1.0 ST arrays.
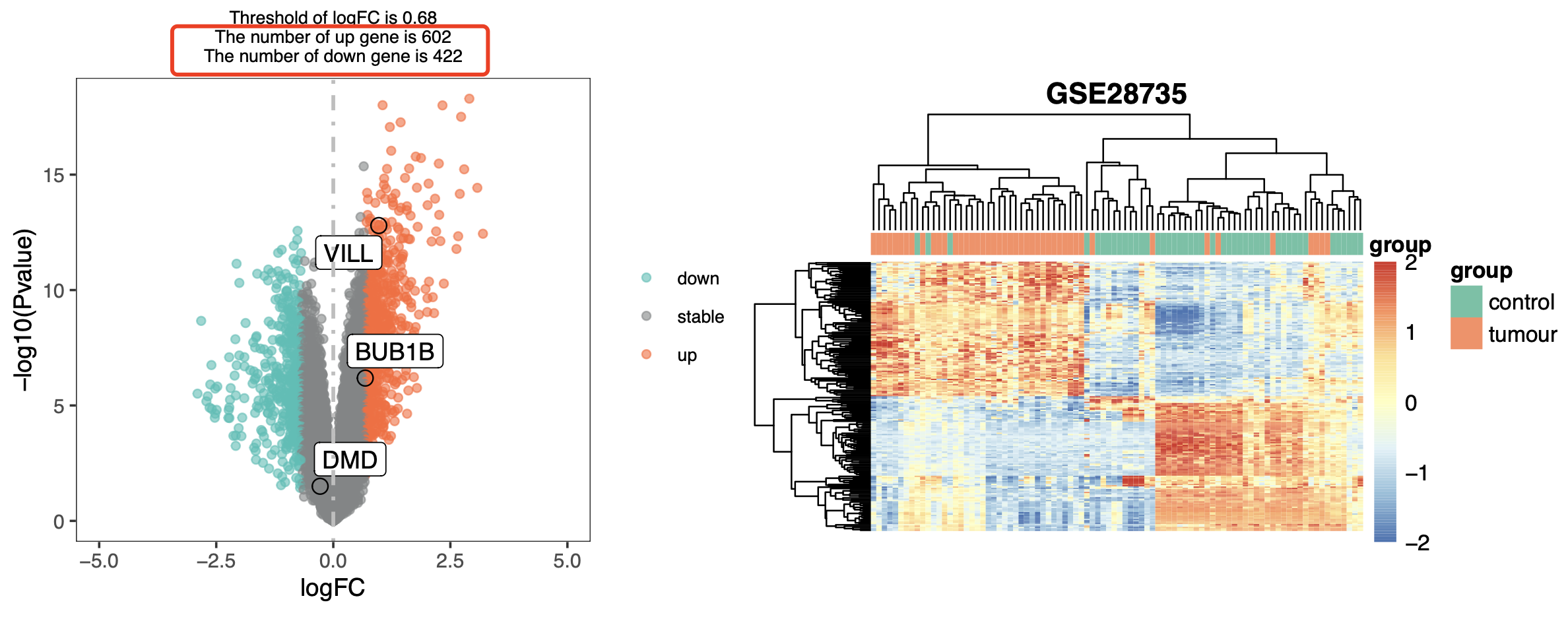
标准的差异分析后会发现你可能会迫不得已选择一个阈值来判断统计学显著的表达量差异变化的基因列表:
打开我们的差异分析结果去检索整合素,就会发现整合素α5 (ITGA5)恰好是一个临界点:
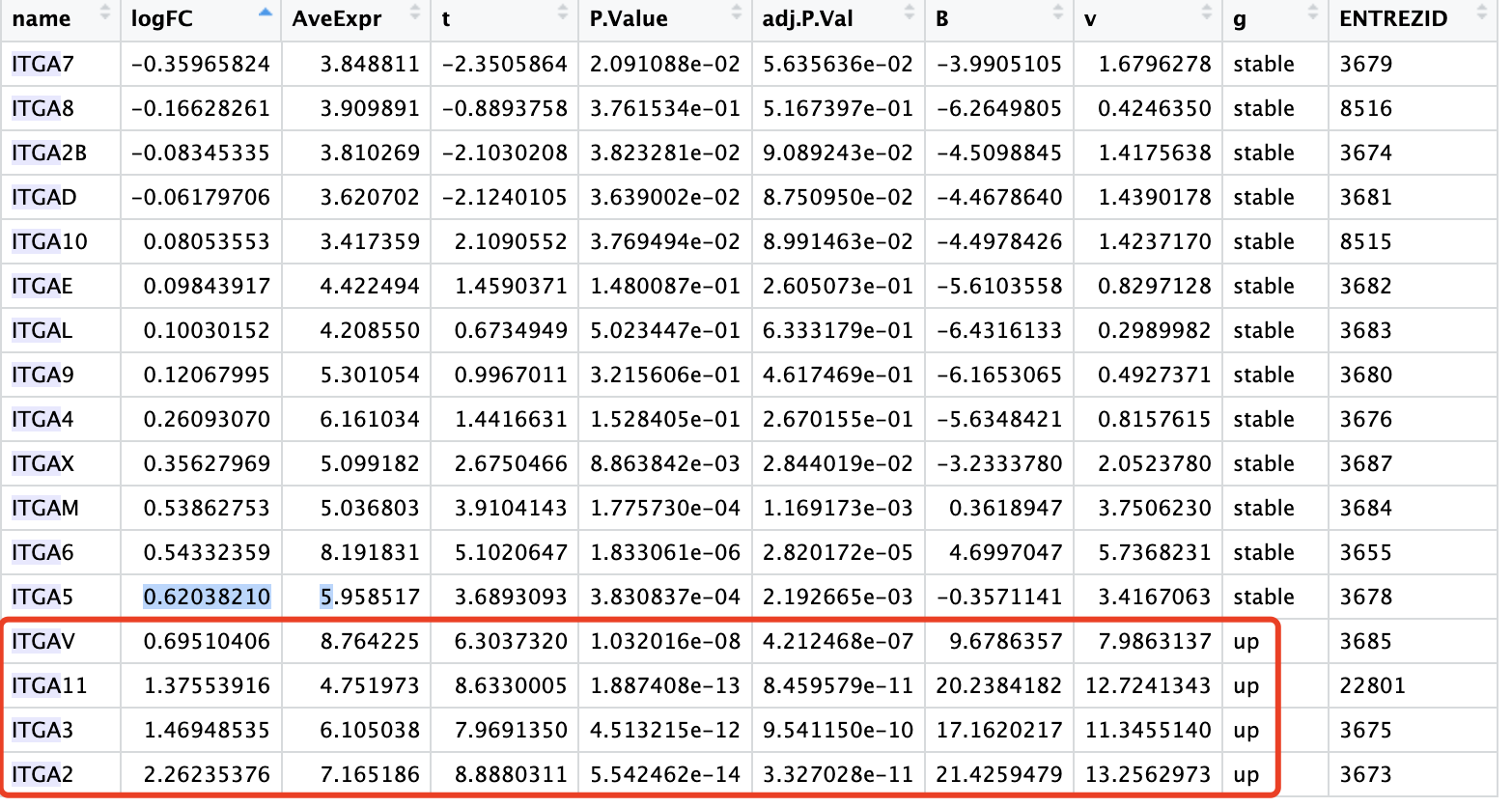
如果你执着于阈值,那么就会信仰崩塌,类似的初学者困惑还有很多,其实通过多练习,见多了世面就不会烦恼了,我们也是一直在整理这方面值得实践的案例 : - chatGPT只能给你提示没办法代替你解决单细胞报错
- 单细胞水平什么程度的表达才算是阳性呢
- 新冠病毒感染引起的急性呼吸窘迫综合征患者外周血有问题吗
- 如果只想知道细胞亚群比例变化无需单细胞转录组测序
- 基因是否具有单细胞亚群特异性居然是靠肉眼看
- 单细胞亚群细分这样弄可能不妥
学徒作业:
如果仅仅是找到类似的有癌旁和癌症的表达量信息数据集,很容易,比如2019的文章:《Identification of differentially expressed genes in pancreatic ductal adenocarcinoma and normal pancreatic tissues based on microarray datasets》:
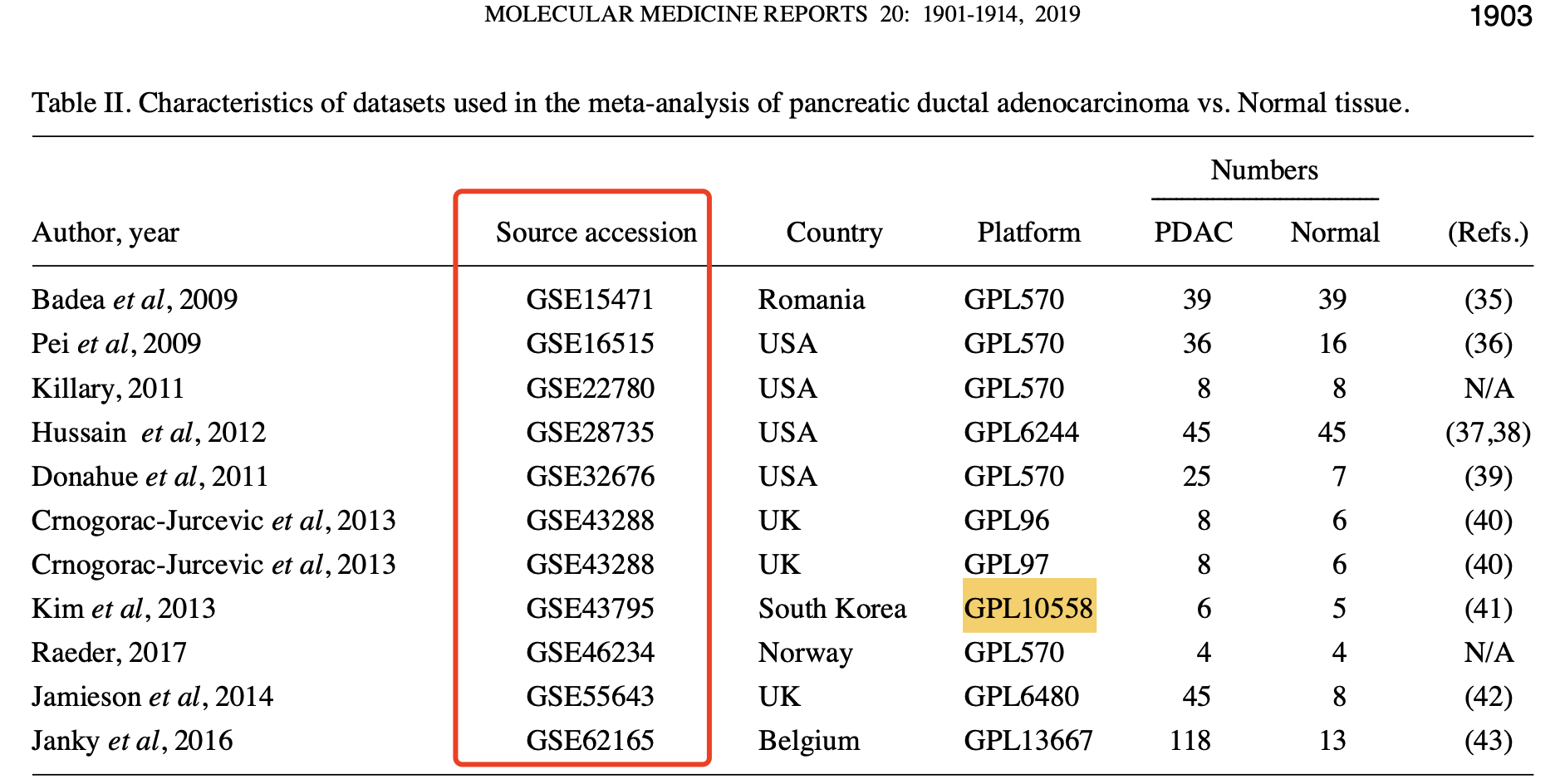
以及2022文章:《Role of Up‐Regulated Transmembrane Channel‐Like Protein 5 in Pancreatic Adenocarcinoma》
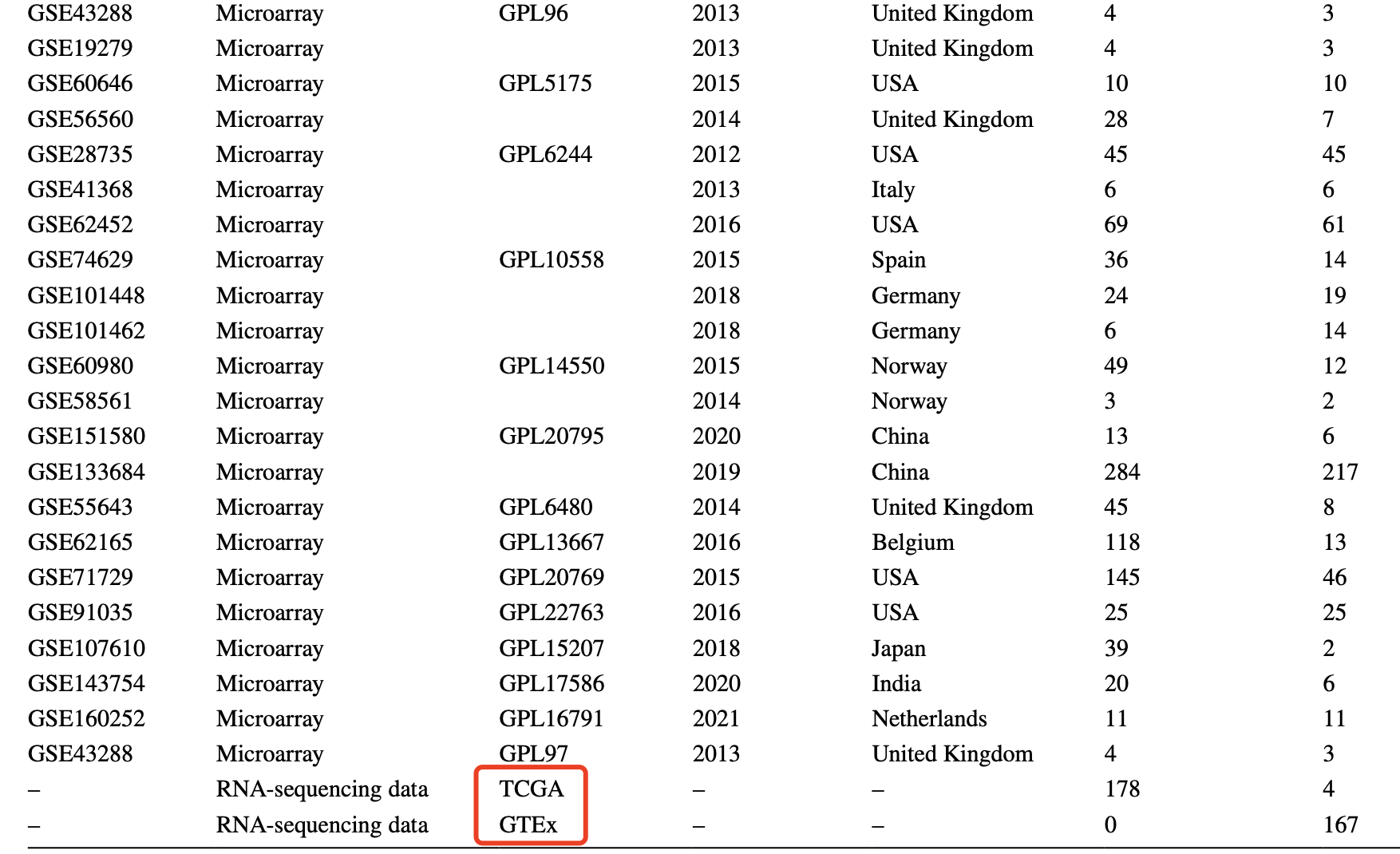
需要大家处理全部的数据集,然后汇总整合素α5 (ITGA5)的表达量差异分析情况,并且合理的可视化。