众所周知,实验室培养的癌症细胞系肯定是非常纯粹的恶性增殖的肿瘤细胞啦。
它被人诟病的一个缺点就是没办法代表真实的肿瘤病人的肿瘤部位的微环境,既然它缺失了肿瘤微环境,如果你测肿瘤细胞系培养物(药物处理前后)的单细胞转录组,就没办法follow我们分享的绝大部分的肿瘤领域的单细胞转录组的第一层次降维聚类分群以及生物学命名,详见之前的介绍 CNS图表复现08—肿瘤单细胞数据第一次分群通用规则, 是 :
- immune (CD45+,PTPRC),
- epithelial/cancer (EpCAM+,EPCAM),
- stromal (CD10+,MME,fibo or CD31+,PECAM1,endo)
比如,2020年发表的CCLE数据库的单细胞转录组数据,文章标题是:《Pan-cancer single-cell RNA-seq identifies recurring programs of cellular heterogeneity》, 这个研究数据整理的非常好,可以下载的数据真多:https://singlecell.broadinstitute.org/single_cell/study/SCP542/pan-cancer-cell-line-heterogeneity#study-download ,因为是纯粹的肿瘤细胞,所以都不需要细胞亚群命名了,仅仅是降维聚类分群即可,可以看到不同细胞系肯定是泾渭分明的,这个是细胞系之间的异质性决定的,在几乎所有的癌症病人的肿瘤单细胞数据里面都有体现:
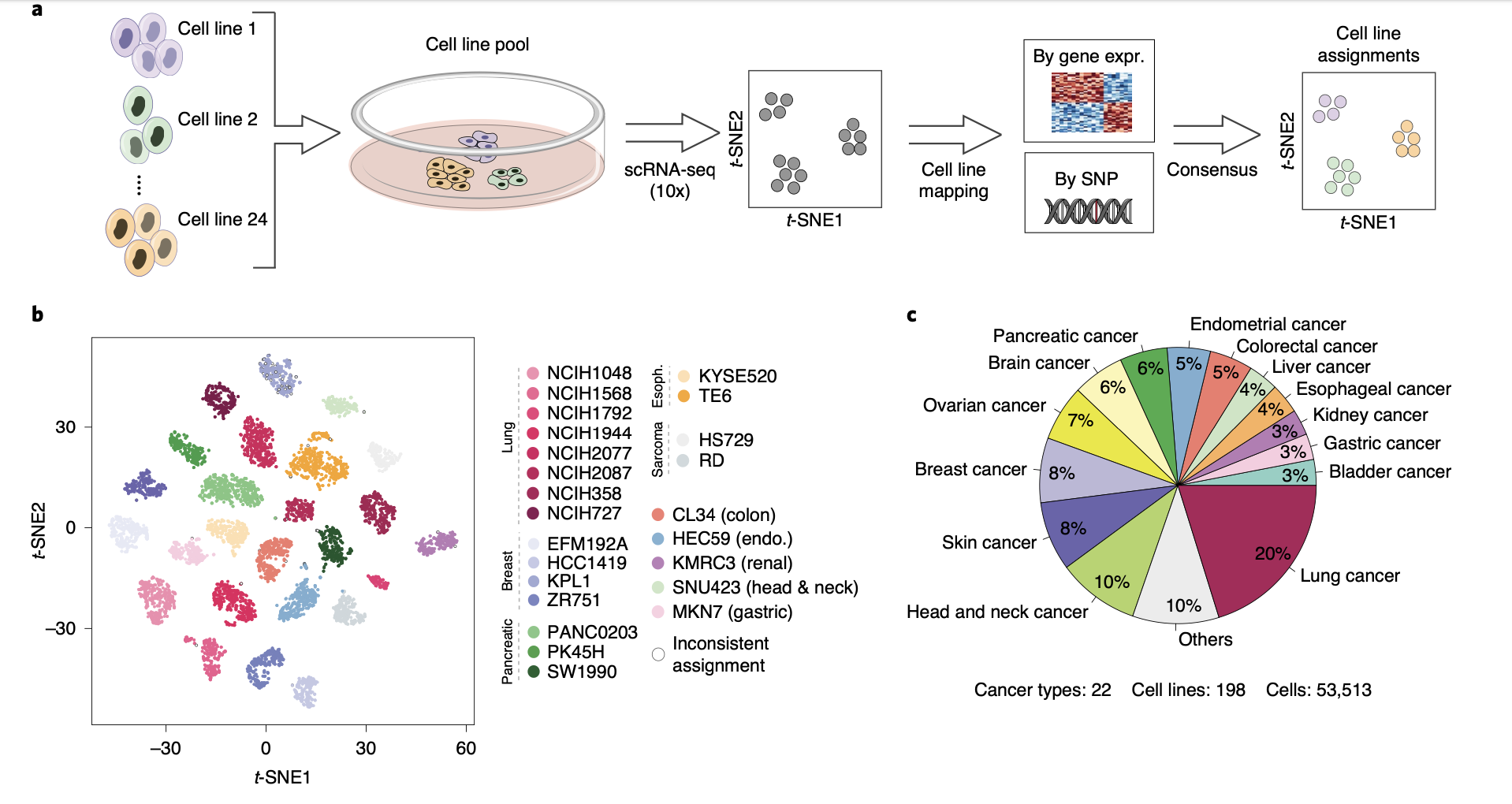
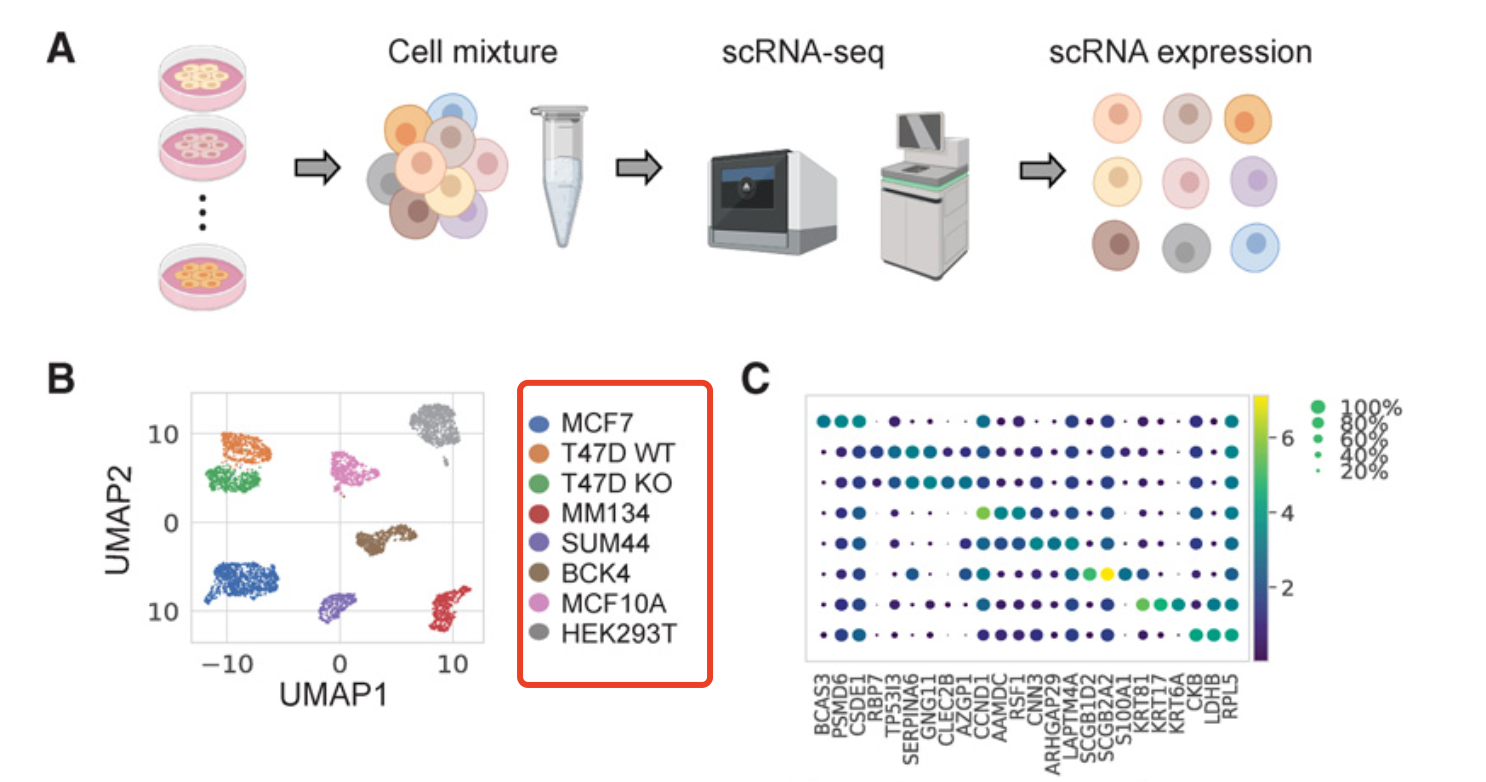
再次强调一下不需要细胞亚群命名了,仅仅是降维聚类分群即可,这样的肿瘤细胞系研究在每个癌症里面也有,比如,2021的GSE144320数据集, 就是6个乳腺癌细胞系混合的单细胞转录组,标题是:《Single-Cell Transcriptomic Heterogeneity in Invasive Ductal and Lobular Breast Cancer Cells》,也是很容易看到不同的细胞系在降维聚类分群后泾渭分明啦,这个是细胞系之间的异质性决定的,在几乎所有的癌症病人的肿瘤单细胞数据里面都有体现:
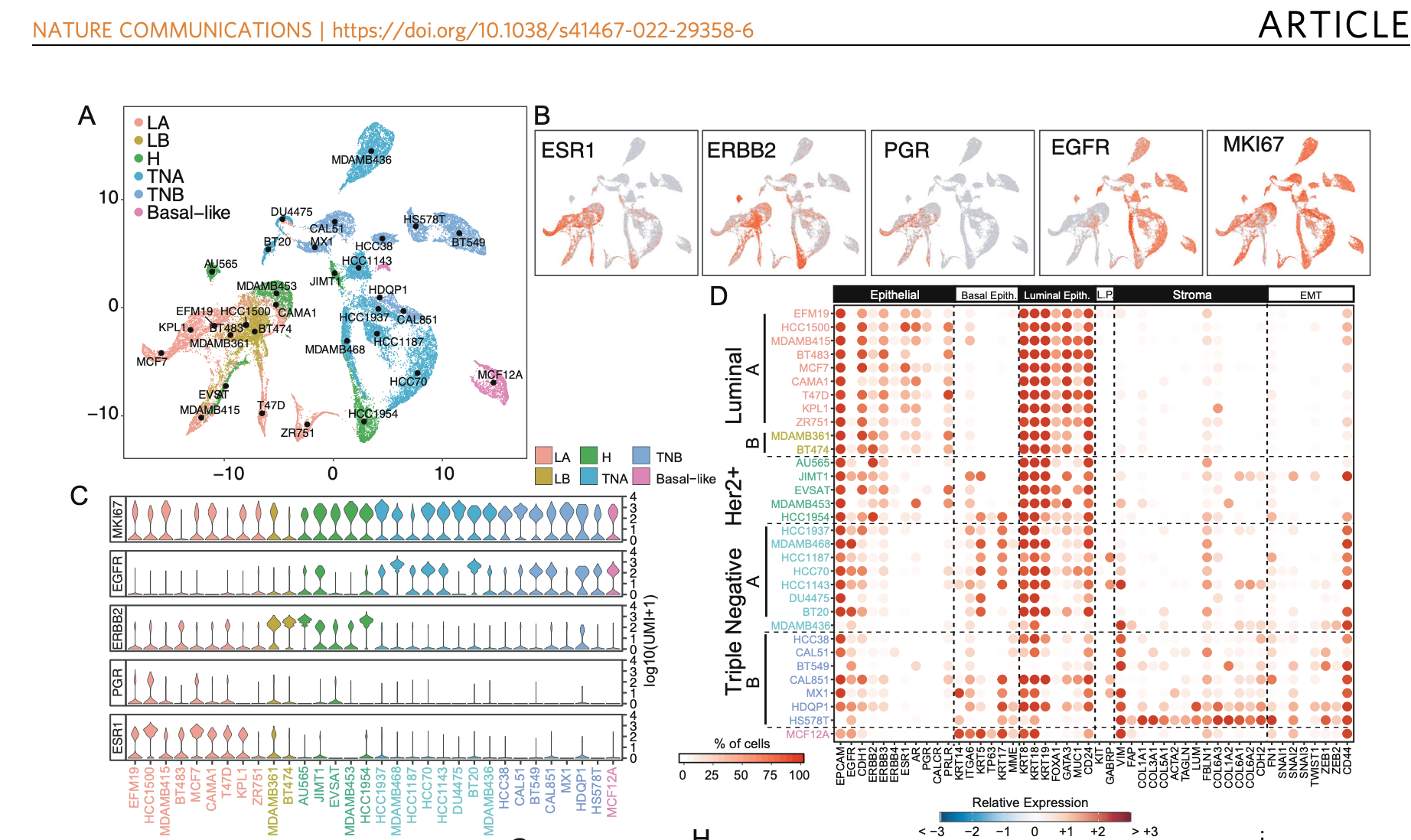
因为单细胞转录组主要是取决于经费啦,细胞系反正是基本上无限量的,2022的GSE173634数据集,就有32个乳腺癌细胞系的单细胞转录组啦,文章标题是:《A single-cell analysis of breast cancer cell lines to study tumour heterogeneity and drug response》,仍然是分群后绝大部分细胞系都是泾渭分明:Representation of single-cell expression profiles of 35,276 cells from 32 cell lines color-coded according to cancer subtype (LA luminal A, LB luminal B, H Her2-enriched, TNA triple-negative type A, TNB triple-negative type B).
我也是下载了它这个32个乳腺癌细胞系的单细胞转录组数据集,自己独立的对它32个乳腺癌细胞系的单细胞转录组降维聚类分群:
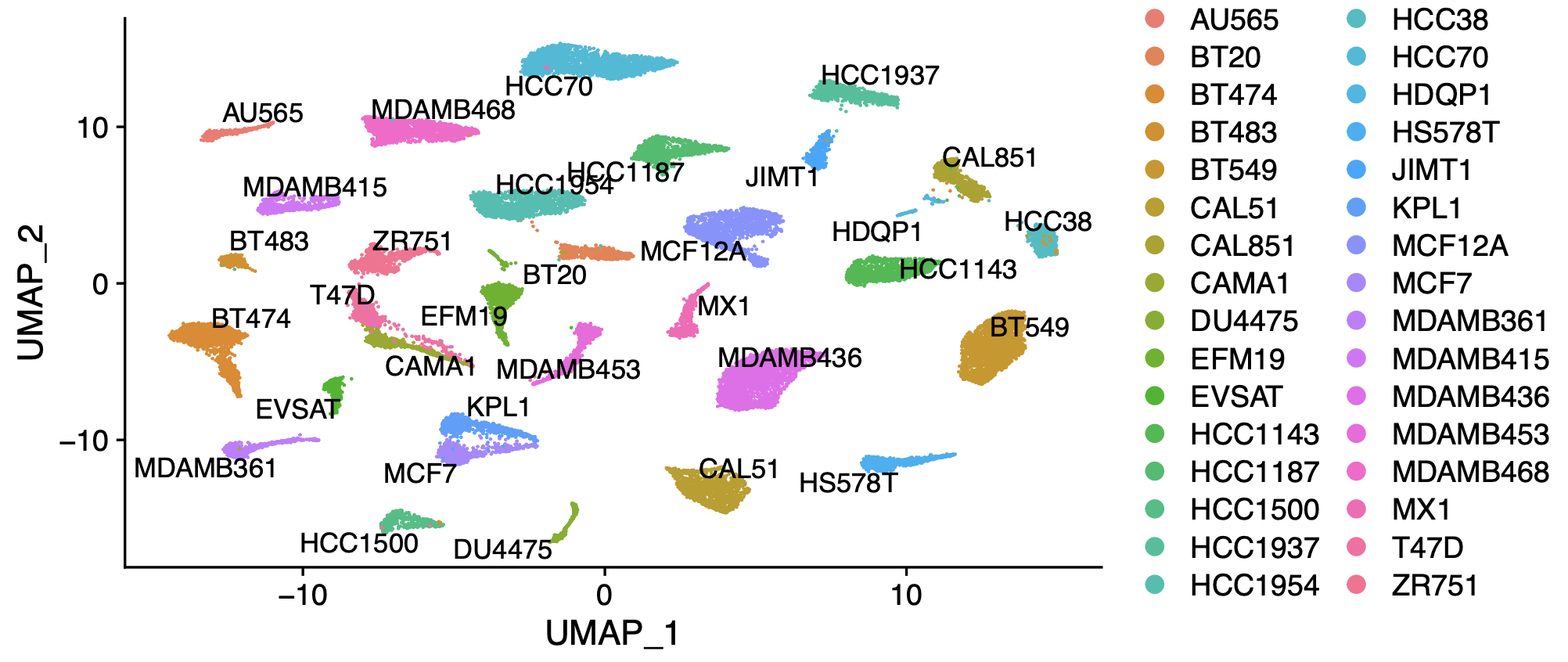
因为确定是肿瘤细胞系,理论上我们不能使用harmony这样的多个单细胞数据整合算法哦,就是需要保证各个细胞系在这样的umap图上面是泾渭分明的。
公共数据集的单细胞转录组数据分析主要难点就是读取表达量矩阵:
ct= readRDS('../GSE173634_RAW_UMI_counts/RAW.UMI.counts.BC.cell.lines.rds')
ct
if(F){
library(AnnoProbe)
ids=annoGene(rownames(ct),'ENSEMBL','human')
}
library(clusterProfiler)
library(org.Hs.eg.db)
ids = bitr(rownames(ct),'ENSEMBL','SYMBOL',OrgDb = 'org.Hs.eg.db')
head(ids)
ids=ids[!duplicated(ids$SYMBOL),]
ids=ids[!duplicated(ids$ENSEMBL),]
kp = rownames(ct) %in% ids$ENSEMBL
table(kp)
ct=ct[kp,]
rownames(ct)=ids$SYMBOL[match( rownames(ct) , ids$ENSEMBL)]
ct[1:4,1:4]
ct[1:4,(ncol(ct)-4):ncol(ct)]
cl=fread('../GSE173634_RAW_UMI_counts/barcodes.tsv.gz',header = F,data.table = F)[,1]
head(cl)
colnames(ct)=cl
sce.all=CreateSeuratObject(counts = ct)
as.data.frame(sce.all@assays$RNA@counts[1:10, 1:2])
head(sce.all@meta.data, 10)
table(sce.all@meta.data$orig.ident)
library(stringr)
length(unique(sce.all@meta.data$orig.ident))
这么多癌症细胞系模型的单细胞转录组数据都说明了里面没有肿瘤免疫微环境,但是传统的bulk转录组数据呢?
学徒作业
下载ccle矩阵的bulk转录组测序表达量矩阵以及芯片表达量矩阵,走ssGSEA流程看22个免疫相关基因集的打分情况。需要简单的免费注册后才能下载:https://portals.broadinstitute.org/ccle/users/sign_in
那么,最近三五年的后起之秀,类器官呢,它培养的肿瘤病人的原位肿瘤后形成的肿瘤类器官里面是纯粹的恶性增殖的肿瘤细胞吗,还是说里面是可以保留肿瘤微环境呢?如果保留,是倾向于保留内皮细胞和成纤维吗还是免疫细胞呢?