一大早就看到了两个chatGPT在生物信息学的应用,感觉有必要参与到其中了。如果大家看完后也有同感,可以一起加入讨论组哈, 群策群力,看看如何更好的向chatGPT提问,如何更好的使用这个AI助手,在我们的生物信息学数据处理过程中。
安装R包问题
正常情况下,如果大家系统性学习了生物信息学数据处理的计算机基础都不为过,我把它粗略的分成基于R语言的统计可视化,以及基于Linux的NGS数据处理:
- 《生信分析人员如何系统入门R(2019更新版)》
- 《生信分析人员如何系统入门Linux(2019更新版)》
不应该是有安装R包的问题,问题是这个社会大家都很着急,肯定是不会抽出三五个月去系统性梳理R知识点,匆匆忙就得干活,就必然面临各种各样的拦路虎, 排第一的就是安装R包问题,比如如果你的电脑用户名里面有中文或者说特殊字符,就会遇到如下所示的报错:
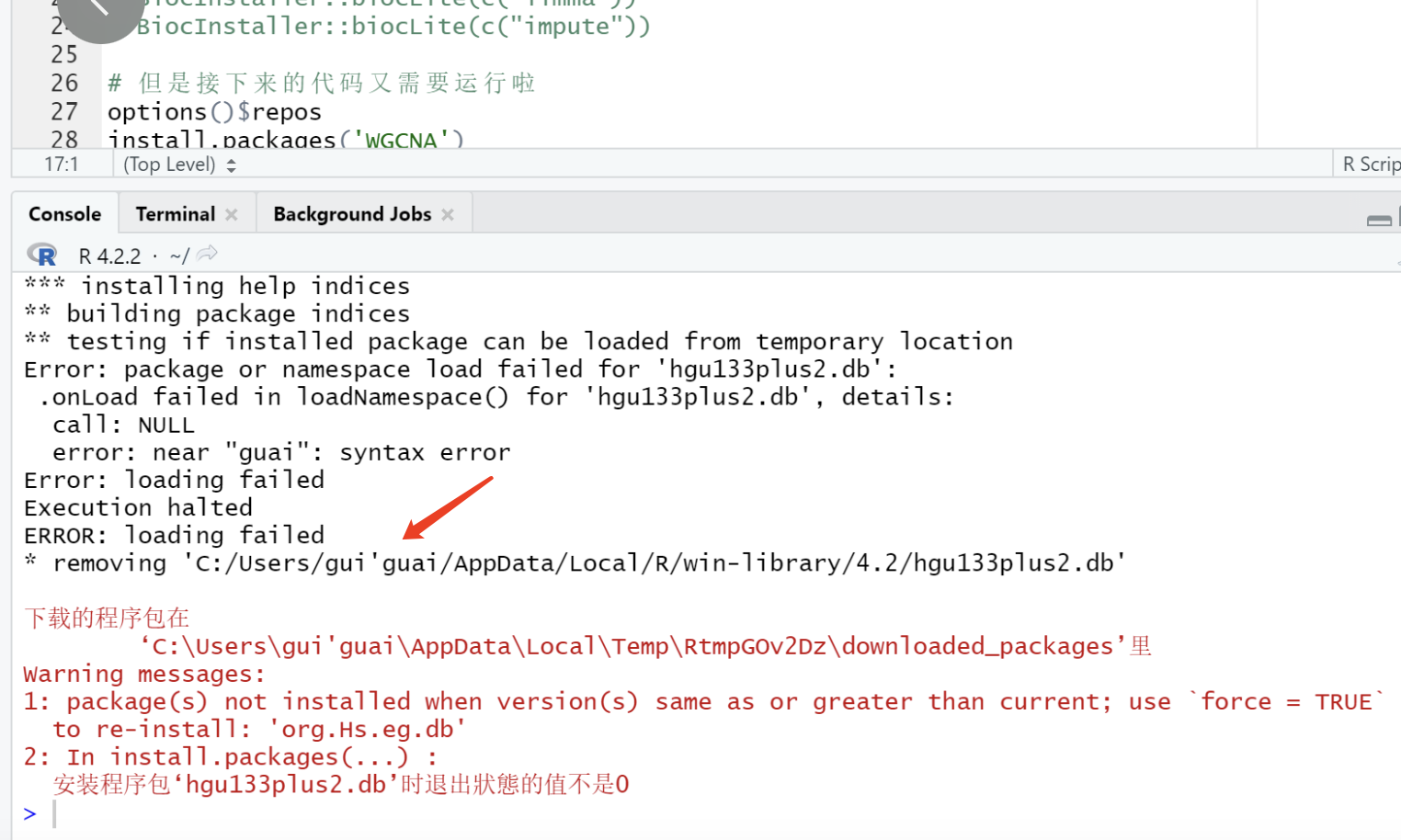
就可以去 https://chat.openai.com/chat 询问:Windows电脑使用R语言的时候因为是中文用户名导致安装R包报错,有解决方案吗?
可以得到如下所示的解决方案:
如果您在Windows电脑上使用中文用户名并且在安装R包时遇到错误,这可能是因为路径中包含中文字符,导致R无法正确加载和安装包。以下是一些可能的解决方案:
- 将R安装在路径中不包含中文字符的文件夹中,例如”C:\R”,然后尝试重新安装R包。
- 在R包的安装路径中使用短名称,避免使用包含空格和中文字符的路径名称。例如,将R包安装在”C:\Rpackages”文件夹中,而不是”C:\用户\您的用户名\文档\R包”。
- 使用管理员权限打开R Studio或R,并尝试重新安装R包。
- 如果您已经安装了R包但无法加载它,可以尝试使用以下命令加载:
vbnetCopy code library("包名", lib.loc="包的路径")其中,“包名”是您要加载的包的名称,“包的路径”是您安装包的路径。
当然了,最好的办法其实是系统性掌握R,变量命名就不应该是因为中文或者字符串,必须以字母或下划线开头。(不推荐用汉字命名) 不能包含空格或标点符号。不能使用关键字(有的书上叫保留字)。对英文字母的大小写敏感,如score和Score不是同一个变量。
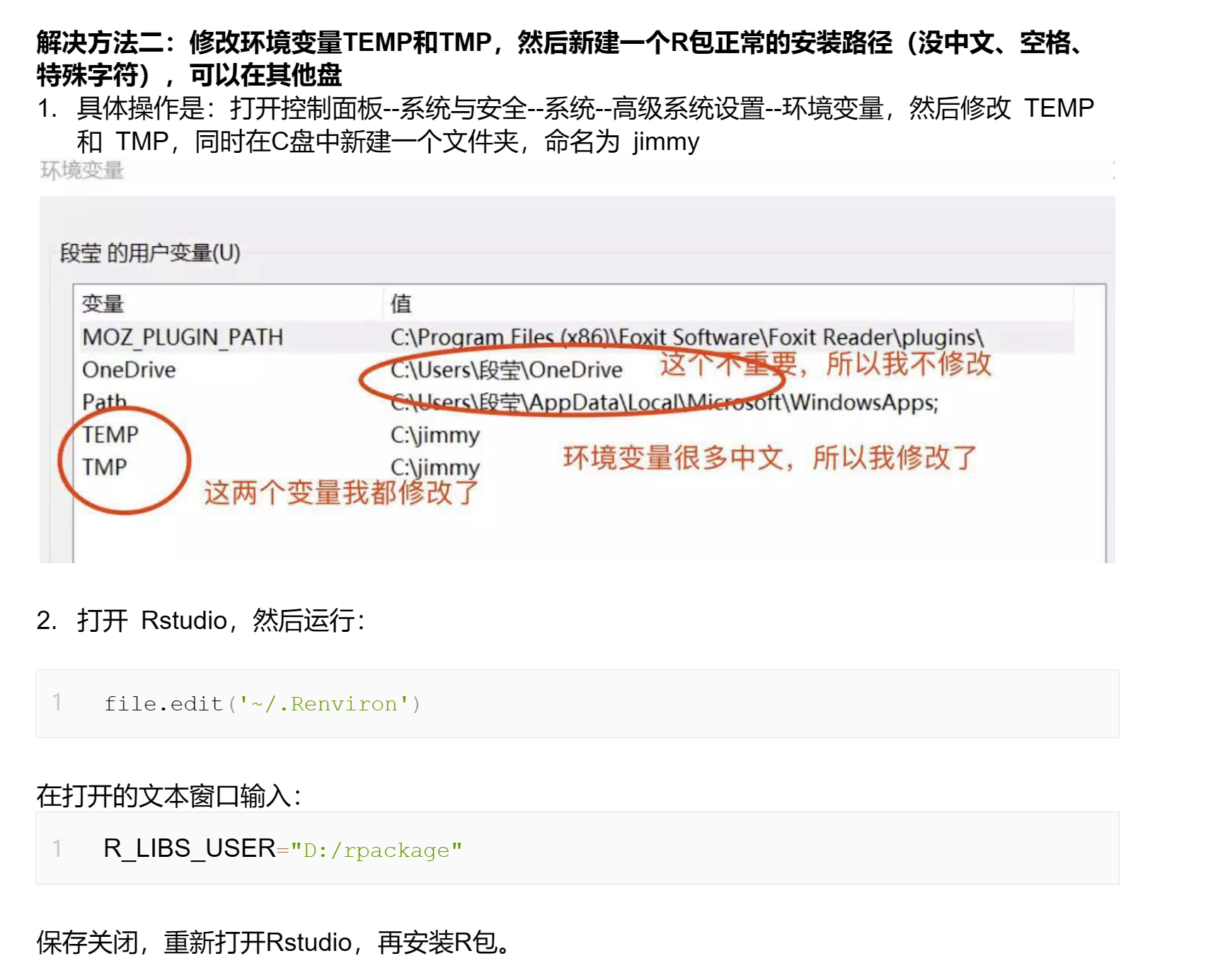
而且这个时候chatGPT也没有给出来最佳方法,我比较喜欢推荐给大家的是修改环境变量即可:
差异基因数量问题
不止一次被问到,差异分析得到的结果不满意,有时候是基因数量太少,有时候是太多,有时候是基因冲突,如下所示::
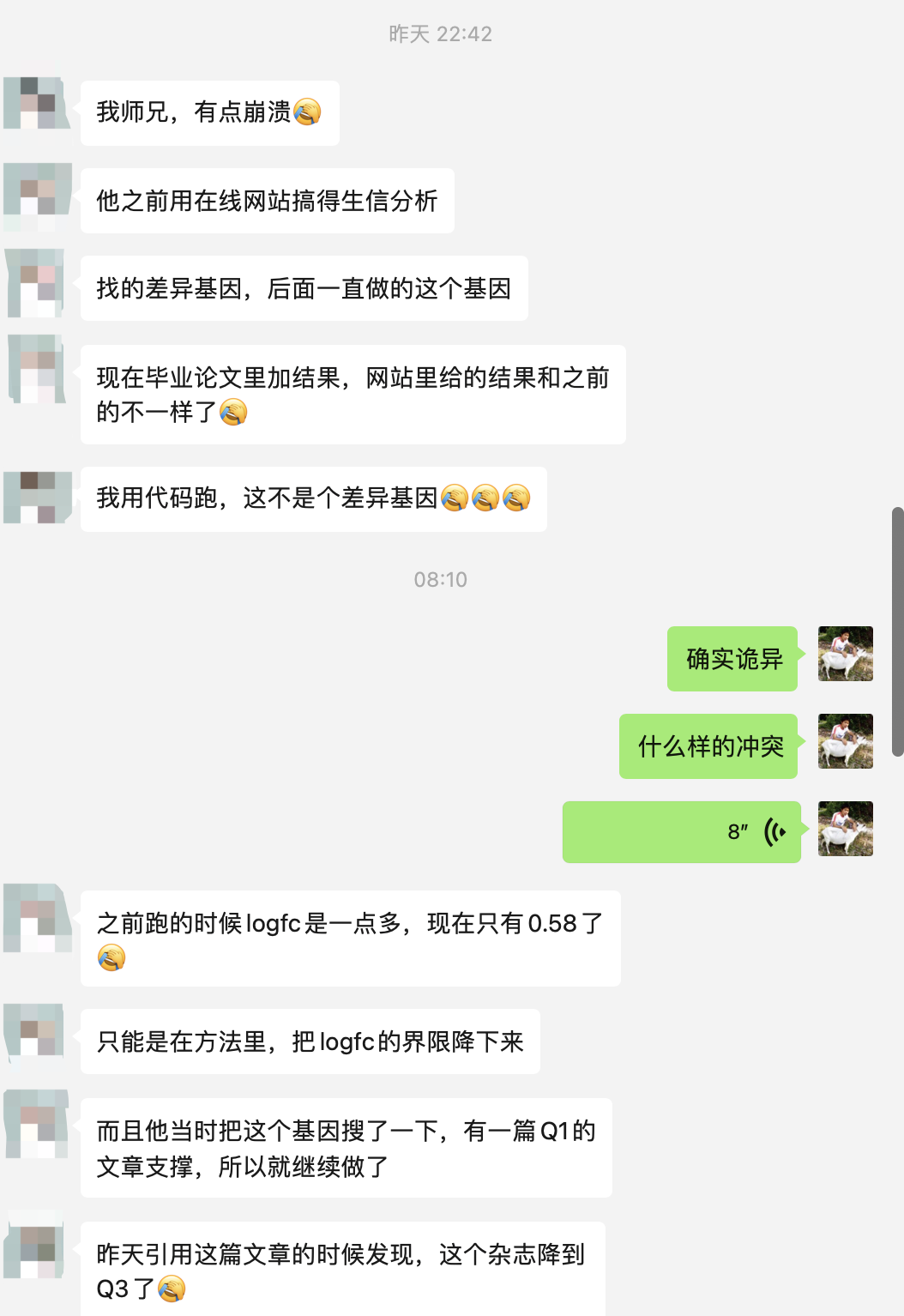
实际上,如果大家系统性学习了生物信息,看完了我的介绍:表达量矩阵差异分析决定上下调基因的阈值,是可以看到这个神奇的公式:cut_logFC <- with(df,mean(abs(df$logFC)) + 2*sd(abs(df$logFC)) )而且每个差异分析需要看质量控制的3张图,当然了,如果你询问chatGPT这个问题的解决方案,就比较官方了。当进行表达量矩阵差异分析时,如果发现差异基因的数量很小,可能是由于多种原因导致的。以下是一些可能的原因以及相应的解决方案:
- 样本数量过少:样本数量越少,检测到的差异基因数量就越少。解决方案是增加样本数量,从而增加差异基因的检测能力。
- 不均一的组内方差:如果组内方差不均匀,可能会掩盖真正的差异基因。这可能需要一些预处理步骤来调整方差,例如标准化或批次效应校正等。
- 数据质量问题:低质量的数据可能会掩盖真正的差异基因。解决方案包括重新过滤和清洗数据、使用更高质量的样品或重新进行实验等。
- 差异基因阈值过高:如果阈值设置过高,可能会导致少量的差异基因。在进行差异分析之前,应该先确定一个合适的阈值,并且应该使用多种方法来确定差异基因。
- 数据正则化问题:不同的数据正则化方法可能导致检测到不同数量的差异基因。可以尝试使用多种不同的正则化方法来检测差异基因,并比较它们的结果。
- 多重比较校正:如果进行多重比较而未进行校正,可能会导致高误报率和少量的差异基因。需要使用一些多重比较校正方法来调整差异分析的统计显著性水平。