之前我们介绍了:这近100种单细胞亚群的2348个标记基因好用吗,提到了作者这里并不是采用我们标准的单细胞可视化标记基因的方法。以前我们做了一个投票:可视化单细胞亚群的标记基因的5个方法,下面的5个基础函数相信大家都是已经烂熟于心了:
- VlnPlot(pbmc, features = c(“MS4A1”, “CD79A”))
- FeaturePlot(pbmc, features = c(“MS4A1”, “CD79A”))
- RidgePlot(pbmc, features = c(“MS4A1”, “CD79A”), ncol = 1)
- DotPlot(pbmc, features = unique(features)) + RotatedAxis()
- DoHeatmap(subset(pbmc, downsample = 100), features = features, size = 3)
是比较自动化的AUCell包的算法 ,很多小伙伴后台提问说具体该如何操作呢,代码层面是否有分享,毕竟我们仅仅是在 :这近100种单细胞亚群的2348个标记基因好用吗,截图了文章里面的method描述而已。
下面我们就以大名鼎鼎的pbmc3k为例子,说明如何使用AUCell结合单细胞亚群标记基因列表来判断亚群名字。
先介绍seurat最常用的示例数据
基本上每个人开始学习单细胞,都是从这个文档开始:https://satijalab.org/seurat/articles/pbmc3k_tutorial.html
假如你安装GitHub的包没有困难,就直接使用下面的代码:
library(Seurat)
# devtools::install_github('satijalab/seurat-data')
library(SeuratData)
library(ggplot2)
library(patchwork)
library(dplyr)
InstallData("pbmc3k")
data("pbmc3k")
pbmc3k
pbmc=pbmc3k
如果你没办法进行GitHub的访问,也可以自己下载pbmc3k_filtered_gene_bc_matrices.tar.gz文件后解压,然后使用 Read10X 函数进行 读取
# Load the PBMC dataset
# https://cf.10xgenomics.com/samples/cell/pbmc3k/pbmc3k_filtered_gene_bc_matrices.tar.gz
pbmc.data <- Read10X(data.dir = "filtered_gene_bc_matrices/hg19/")
# Initialize the Seurat object with the raw (non-normalized data).
pbmc <- CreateSeuratObject(counts = pbmc.data, project = "pbmc3k", min.cells = 3, min.features = 200)
pbmc
这个 pbmc 变量就是全部单细胞数据分析所需要的,是一个被精心定义好的对象哦:
> pbmc
An object of class Seurat
13714 features across 2700 samples within 1 assay
Active assay: RNA (13714 features, 0 variable features)
然后搞定单细胞亚群标记基因列表
我这里测试了多种单细胞亚群的标记基因列表,发现如果是比较过于类似的细胞是没办法区分的,比如 CD14+ Mono 和 FCGR3A+ Mono,以及 Memory CD4 T和 Naive CD4 T ,或者说是需要首先区分了它们后找到了它们的特异性基因然后再使用这些基因才有可能区分它们,就陷入了一个鸡生蛋蛋生鸡的死循环。。。。
但是,对b细胞来说,就没有这个问题, 因为它和t细胞是截然不同,而且也不可能去跟髓系免疫细胞混淆。。。
library(AUCell)
library(Seurat)
Bcells = c('MS4A1','SDC1','CD27','CD38','CD19', 'CD79A')
counts <- GetAssayData(object = sce, slot = "counts")
cell_rankings <- AUCell_buildRankings(counts)
cells_AUC <- AUCell_calcAUC(Bcells, cell_rankings)
cells_assignment <- AUCell_exploreThresholds(cells_AUC, plotHist = TRUE, assign=TRUE)
thr = cells_assignment$geneSet$aucThr$selected;thr
new_cells <- names(which(getAUC(cells_AUC)["geneSet",]> thr))
sce$is_list <- ifelse(colnames(sce) %in% new_cells, "list", "non_list")
colnames(sce@meta.data)
DimPlot(object = sce, group.by = "is_list", label = TRUE) +
DimPlot(object = sce, group.by = "seurat_annotations", label = TRUE)
让我们来看看b细胞的效果吧:
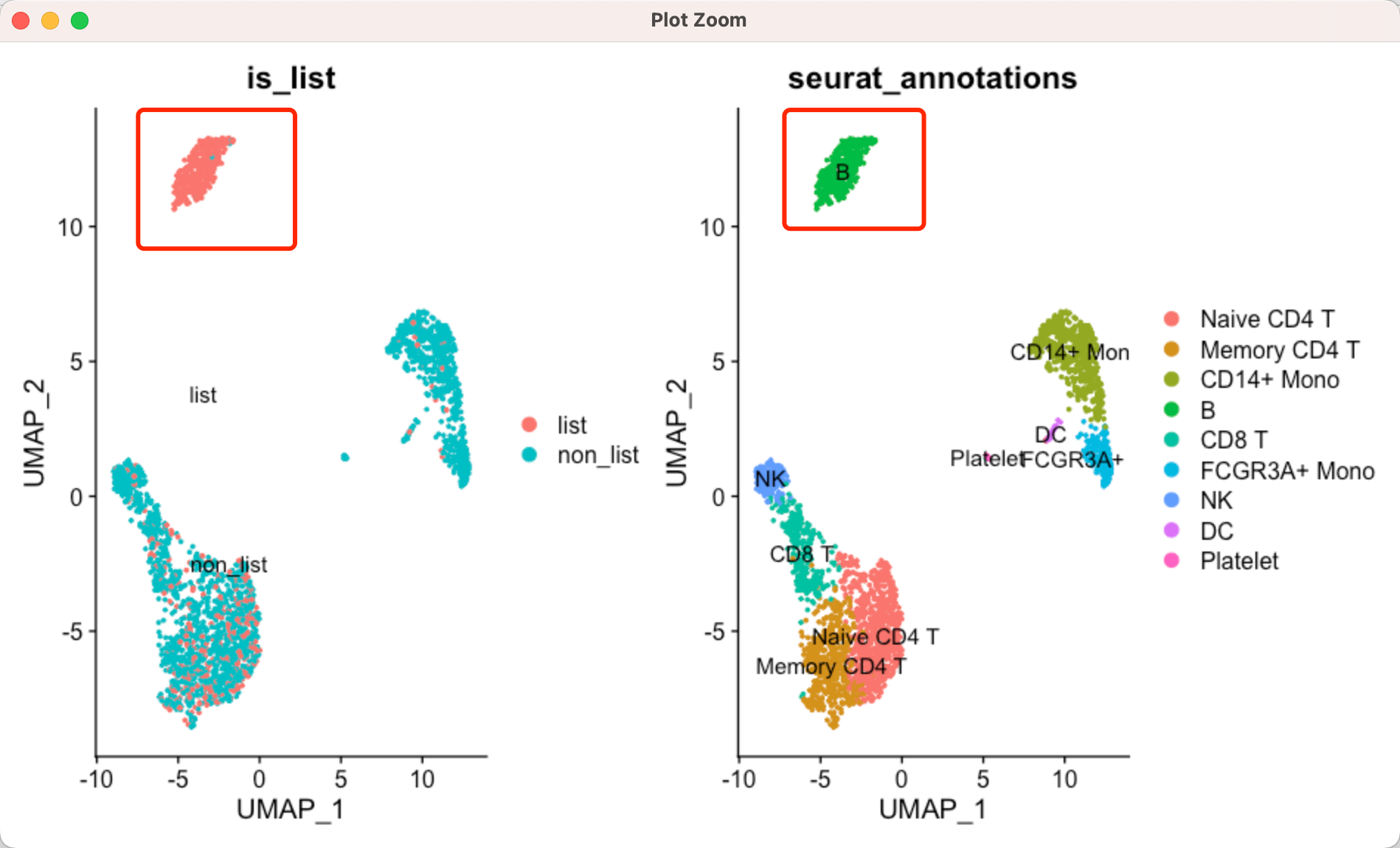
可以看到基本上是近乎完美的区分了b细胞和其它细胞,就需要实现知道所有的单细胞亚群的标记基因然后去一个个测试。所以这种方法并不是单细胞亚群命名的主流操作,仅仅是特殊情况下,需要去确定某个未知的单细胞亚群或者说想搞清楚某个状态的单细胞亚群或者某个通路激活的单细胞才会这样的操作。。。
也可以是其它来源的基因列表,比如:PanglaoDB_markers_27_Mar_2020.tsv,大家可以去官网下载它,我这里给一个github链接(可能会失效):
markers <- read.csv("PanglaoDB_markers_27_Mar_2020.tsv", sep = "\t")
markers <- markers[markers$cell.type == "Endothelial cells" & markers$species != "Hs",]
markers
genes <- markers$official.gene.symbol
mousify <- function(a){
return(paste0(substr(a,1,1), tolower(substr(a,2,nchar(a)))))
}
genes <- sapply(genes, mousify)
genes
或者 download all cell-type gene signatures from panglaoDB官方网站 ,https://panglaodb.se/markers/PanglaoDB_markers_27_Mar_2020.tsv.gz
更多的用处可以是针对某个亚群显示多个基因的打分
上面的AUCell的3个函数,就是针对b细胞的多个基因,在pbmc的3000多个细胞里面打分了,然后展现打分后的值:
Bcells = c('MS4A1','SDC1','CD27','CD38','CD19', 'CD79A')
这样的打分算法非常多,而且在多种场合多有应用,比如2020的文章:《Human and mouse single-nucleus transcriptomics reveal TREM2-dependent and TREM2-independent cellular responses in Alzheimer’s disease》,是阿兹海默症的单细胞数据集,可以看到里面的小胶质巨噬细胞的打分非常特异:

学徒作业
完成上面的2020-GSE140511-阿兹海默症单细胞数据集的降维聚类分群,然后使用前面的AUCell的3个函数,针对文章里面的小胶质巨噬细胞的6个基因进行打分哈。