最近单细胞数据挖掘文章如雨后春笋般冒出来了,总体来说就两个方向:
- 找到一个数据集,降维聚类分群后,拿到基因列表后,去TCGA建模
- 首先TCGA建模然后拿模型里面的基因或者基因集合去单细胞转录组数据集看是否有特殊的表现
这两个方向都需要掌握基础的单细胞转录组数据集的降维聚类分群即可,这不过两个方向其实都是只需要一个数据集即可,而且因为单细胞数据处理对计算机资源要求比较高,绝大部分小伙伴也更倾向于处理单个数据集。第一个策略是从单细胞拿到基因列表
- 2023-单细胞-LUAD-NK细胞相关预后基因集-GSE131907.pdf (doi: 10.3389/fimmu.2022.850745)
- 2023-单细胞-肝癌-NK细胞相关预后基因集-GSE140228.pdf(https://doi.org/10.3390/ijms24119587)
- 2023-单细胞-肝癌-NK细胞相关预后基因集-GSE162616.pdf(https://doi.org/10.3390/ijms24119587)
第二个策略是建模的基因去单细胞验证
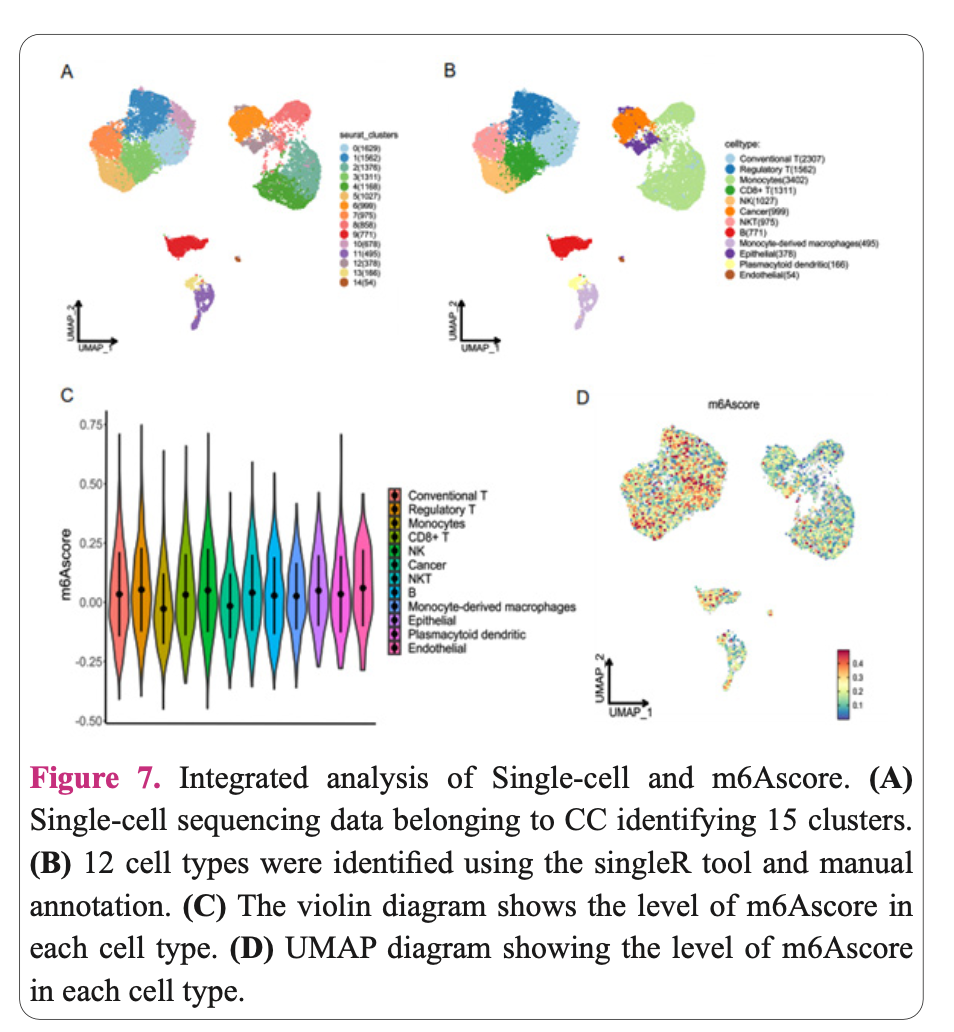
比如国庆节前一天刚刚在线的:《The prognostic, immunological and single-cell features of m6A molecules in cervical cancer》,就是前面的常规的差异分析+wgcna后拿到了m6a相关的预后模型(m6Ascore),然后去单细胞数据集里面看模型(m6Ascore)的表现:
目前来说,处理单个数据集对绝大部分小伙伴来说难度很小,所以就陷入了第一个内卷的后果, 就是同一个数据集被多个文章反反复复挖掘,就非常尴尬,如下所示: - 2023-单细胞挖掘-GSE131907-LUAD-Scissor.pdf (DOI 10.3389/fcell.2023.1163314)
- 2023-单细胞挖掘-GSE131907-LUAD-脑转移-拷各亚群细分.pdf (https://doi.org/10.1007/s12672-023-00784-2 )
- 2023-单细胞挖掘-GSE131907-LUAD-脑转移-拷贝数看肿瘤起源.pdf( https://doi.org/10.1093/neuonc/noad017 )
- 2023-单细胞挖掘-GSE131907-LUAD-调控网络.pdf(https://doi.org/10.3390/biom13040671)
内卷2:一次性挖掘多个单细胞数据集
但是最近有一些小伙伴开始找我买服务器来处理单细胞数据集了,而他们不太可能是能花成百上千万的经费自己产出单细胞数据集的课题组,但是要处理的单细胞数量却已经是成百上千万了。。。。
简单的交流后,发现原来是单细胞数据挖掘的内卷时代到来了,简单的处理一个数据集没办法从工作量角度去打动杂志期刊。 - 2023-脑瘤-单细胞数据挖掘-4个数据集.pdf (doi: 10.7150/thno.81407)
- 2023-骨肉瘤-单细胞数据挖掘-3个数据集.pdf(https://doi.org/10.1016/j.jbo.2023.100493)
- 2023-单细胞数据挖掘-心衰-3个数据集.pdf(doi: 10.6515/ACS.202307_39(4).20221219B)
- 2023-五种自免疫疾病-单细胞差异.pdf(doi: 10.15212/bioi-2023-0012 )
当然了,这样的内卷,仍然是所以低水平的重复啦。
如果真的要学习,建议看张泽民课题组的泛癌层面的单细胞数据挖掘文章, 我简单看了看,类似的策略的数据挖掘居然一直可以发CNS级别杂志,让人膜拜: - 2021年2月发表在CELL的《A pan-cancer single-cell transcriptional atlas of tumor infiltrating myeloid cells》
- 2021年12月发表在science的《Pan-cancer single cell landscape of tumor-infiltrating T cells》
- 2023年9月发表在CELL的《A pan-cancer single-cell panorama of human natural killer cells》