看到了一个单细胞表观资源文章,是 2.3 million individual brain cells from 117 anatomical dissections
对应的文章是Single-cell analysis of chromatin accessibility in the adult mouse brain. Nature 2023 Dec ,PMID: 38092917
分析得到的结果非常惊人,是 1 million cCREs and their chromatin accessibility across 1482 distinct brain cell populations
真的是名副其实的 a comprehensive atlas of candidate cis-regulatory DNA elements (cCREs)
数据确实是公开的:
- Demultiplexed FASTQ files are available at https://assets.nemoarchive.org/dat-bej4ymm
- https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE246791
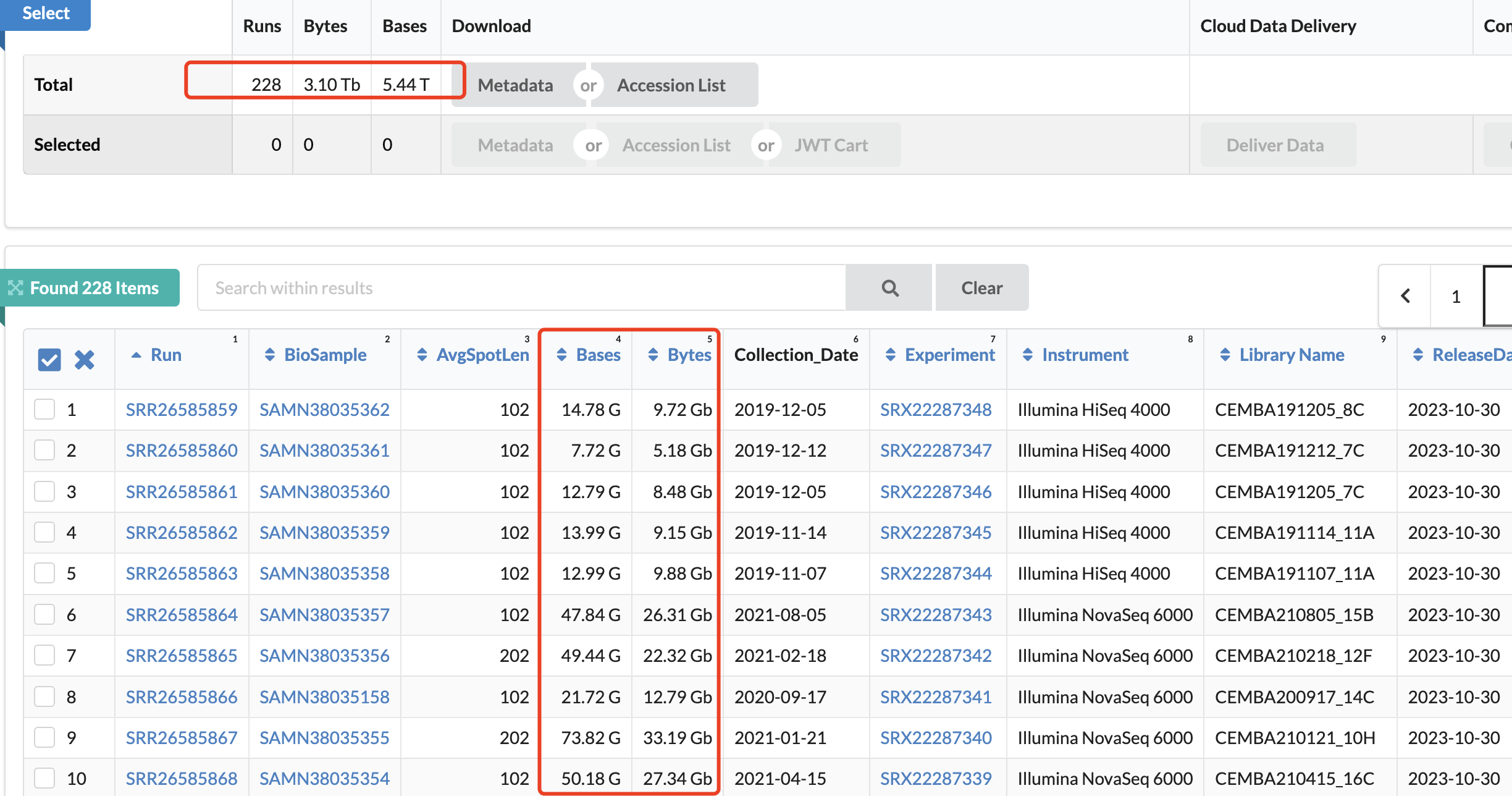
首先,仅仅是GEO数据库的二级数据就 150G 的压缩包文件:
Supplementary file Size Download File type/resource
GSE246791_RAW.tar 149.8 Gb TAR (of H5AD)
GSE246791_README.txt 874 b TXT
GSE246791_sa2.subclass.names.map.csv.gz 8.5 Kb CSV
GSE246791_wmb_SnapATAC2_anndata.tar.gz 117.7 Gb TAR
GSE246791_wmb_bigwigs.tar.gz 233.1 Gb TAR
GSE246791_wmb_enhancer.tar.gz 16.8 Mb TAR
228个样品确实是让人望而生畏啦:
Samples (228)
GSM7876880 9L,rep1
GSM7876881 6F,rep1
GSM7876882 9L,rep2
GSM7876883 7F,rep1
GSM7876884 8D,rep2
GSM7876885 10B,rep1
GSM7876886 10B,rep2
GSM7876887 8F,rep2
GSM7876888 8F,rep1
一般人真的是没办法利用这个 single-nucleus assys for transposase-accessible chromatin using sequencing (snATAC-seq) libraries from 117 anatomic dissections of mouse brain and two replicates for each dissection.
但是作者( University of California San Diego)给出来了他们的课题组网页,里面公布了最近他们全部的单细胞 图谱研究:
- Web link http://catlas.org/catlas_hub/
- 是 https://biccn.org/ 的一部分 ( NIH’s Brain Research through Advancing Innovative Neurotechnologies (BRAIN) Initiative - Cell Census Network (BICCN) )
最近三五年,都是类似的 单细胞 图谱研究 :
如果是从fastq文件开始:
因为基本上99%的生物信息学数据分析人员是没有自己独立的服务器,只能说是做一点点二级数据的统计可视化级别的数据挖掘而已,所以仅仅是raw data就这么大, 肯定是没戏了。