食管鳞状细胞癌(Esophageal Squamous Cell Carcinoma,ESCC)和食管腺癌(Esophageal Adenocarcinoma,EAC)是两种常见的食管癌症类型,它们在病理学、发病机制和临床特征上存在显著差异。
- ESCC是一种起源于食管鳞状上皮的癌症。鳞状细胞是食管内最外层的上皮细胞。主要与饮食、烟草使用、酒精摄入和慢性食管炎等因素有关。遗传因素也可能对发病有一定影响。东亚、中东和部分亚非地区是高发地区。
- EAC是一种起源于食管黏液腺细胞的癌症。通常,它是从食管下半段的腺体组织中发展而来。与胃食管反流病(Gastroesophageal Reflux Disease,GERD)和Barrett食管(Barrett’s Esophagus)的发展密切相关。Barrett食管是一种黏液腺上皮替代食管正常鳞状上皮的情况。
昨天我们介绍了2023年5月某院士团队发表在顶刊的文章:《Epithelial cells activate fibroblasts to promote esophageal cancer development》。详见:肿瘤相关成纤维单细胞亚群的iCAF和myCAF迷思,该研究的取样涵盖了 NOR(正常食管上皮)、LGIN(低级上皮内病变)和HGIN(高级上皮内病变) ,而且很清晰的看到了这个成纤维细胞和巨噬细胞(属于髓系免疫细胞)都是比例下降的:
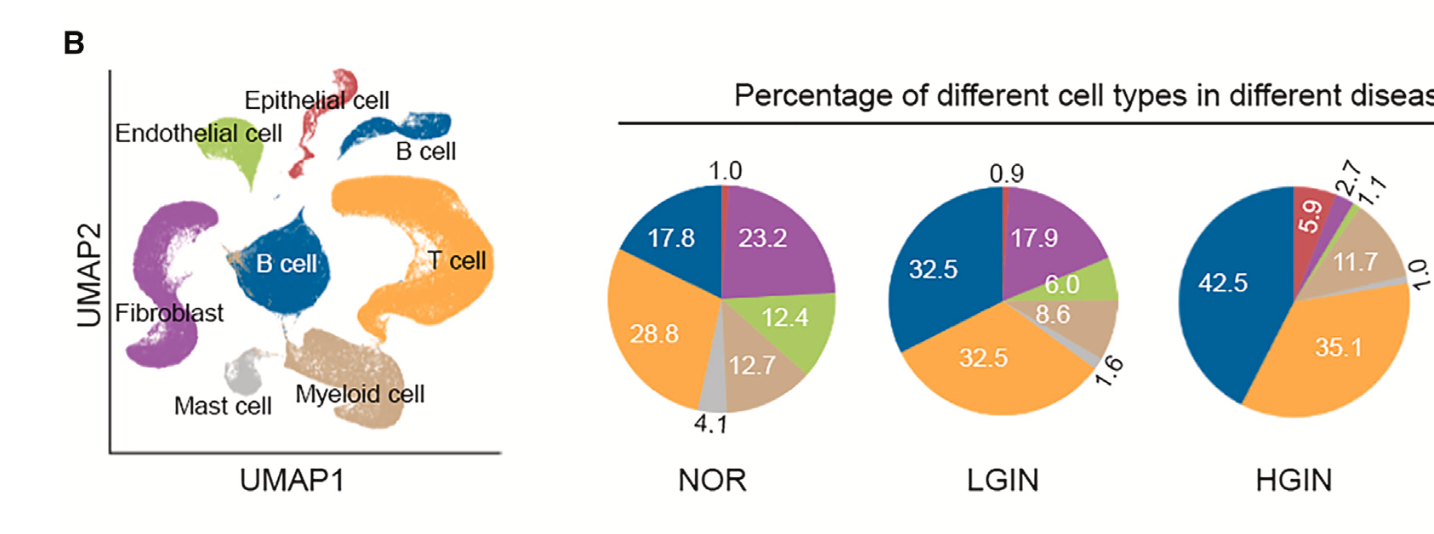
但是, 另外一个2023年6月也是发表在顶刊的文章:《Spatial transcriptomics analysis of esophageal squamous precancerous lesions and their progression to esophageal cancer》,做的说食管鳞状上皮癌前病变(Esophageal Squamous Precancerous Lesions,ESPL)的疾病进展,也是包括了正常上皮(NE,Normal Epithelia)、低级上皮内病变(LGIN,Low-Grade Intraepithelial Neoplasia)、高级上皮内病变(HGIN,High-Grade Intraepithelial Neoplasia) 。但是他们得到的细胞比例变化规律就居然是完全相反的,成纤维细胞和巨噬细胞(属于髓系免疫细胞)都是比例上升的,如下所示:
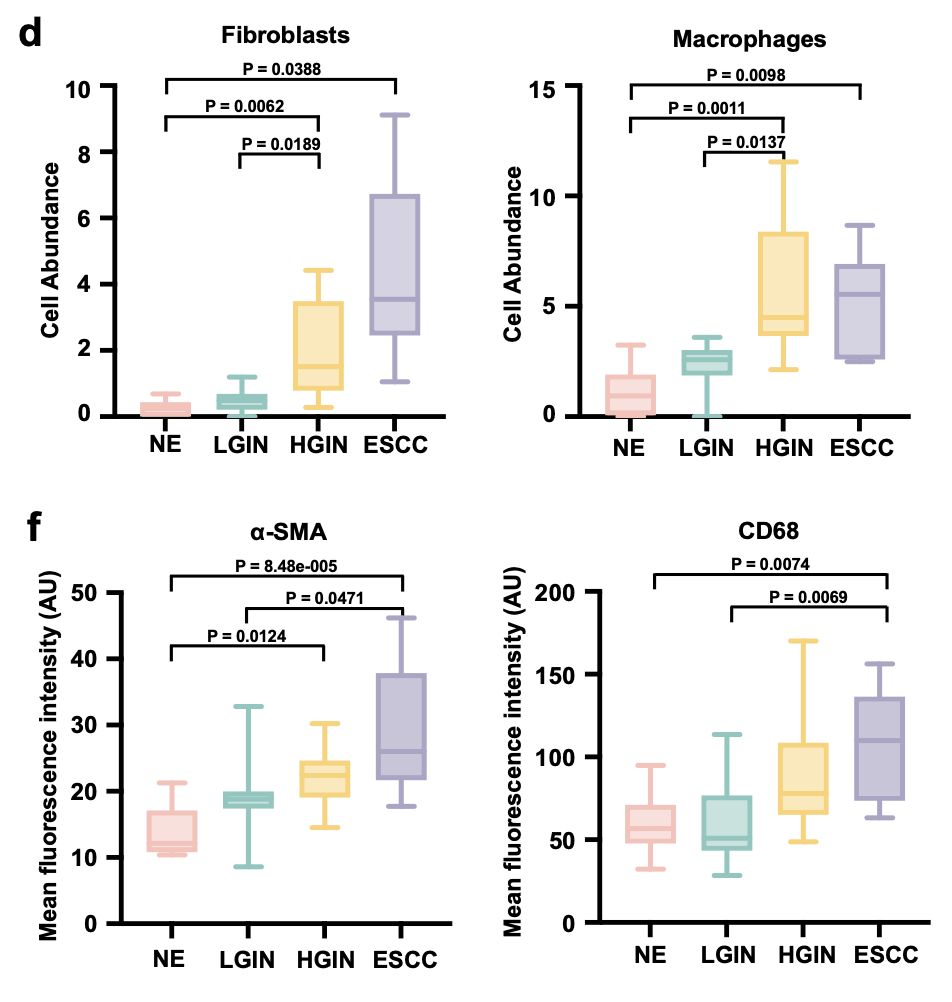
这个就很尴尬了,几百年过去了,癌症研究领域这么多专家,居然没办法在这么简单的一个问题上面达成共识吗?简简单单的疾病进展过程的细胞比例变化都搞不清楚吗?生命科学领域如此有意思的啊!
是因为成纤维和巨噬细胞都有各自的细分亚群,所以它们的各自的标记基因表达量变化趋势以及其所代表的细分亚群的比例,可能会与其所属于的成纤维和巨噬细胞总体变化不一致呢?那疾病进展状态的其它组学
前面的表达量和细胞比例变化都是转录组层面的,早期的表达量芯片到后来的转录组测序到现在的单细胞转录组,理论上应该是同样的实验设计的转录组学文章非常多,或者说起码应该是有一个综述来汇总这方面研究然后提炼出来一个后面的研究者可以拿来就用的规律啊。
那疾病进展状态的其它组学信号,是否在疾病进展期间的变化是稳定的呢?
众所周知,肿瘤外显子是TCGA计划的6大数据之一,而TCGA数据库是目前最综合最全面的癌症病人相关组学数据库,包括: - DNA Sequencing(主要是肿瘤外显子)
- miRNA Sequencing(很小众)
- Protein Expression array (基本上不用)
- mRNA Sequencing(数据挖掘玩烂了的)
- Array-based Expression (基本上淘汰了)
- DNA Methylation (也是有点小众)
- Copy Number array(会跟肿瘤外显子重叠的生物学意义)
知名的肿瘤研究机构都有着自己的TCGA数据库探索工具,比如: - Broad Institute FireBrowse portal, The Broad Institute
- cBioPortal for Cancer Genomics, Memorial Sloan-Kettering Cancer Center
比如发表于2023年9月的文章:《Absence of Lugol staining indicates initiation of esophageal squamous cell carcinoma: A combined genomic and epidemiologic study》,纳入了Lugol-unstained lesions (LULs) 的分组,Lugol溶液通常含有碘,对于正常的食管黏膜,碘溶液能够使其变为深褐色,而对于癌变区域则失去了这种反应,呈现未被染色的状态。在食管癌的早期诊断中,Lugol染色法被用来检测这些LULs,因为这些区域可能是食管鳞状细胞癌(ESCC)的潜在早期病变。然后发现SNV和CNV的趋势很明显是随着疾病进展而增加的,如下所示:
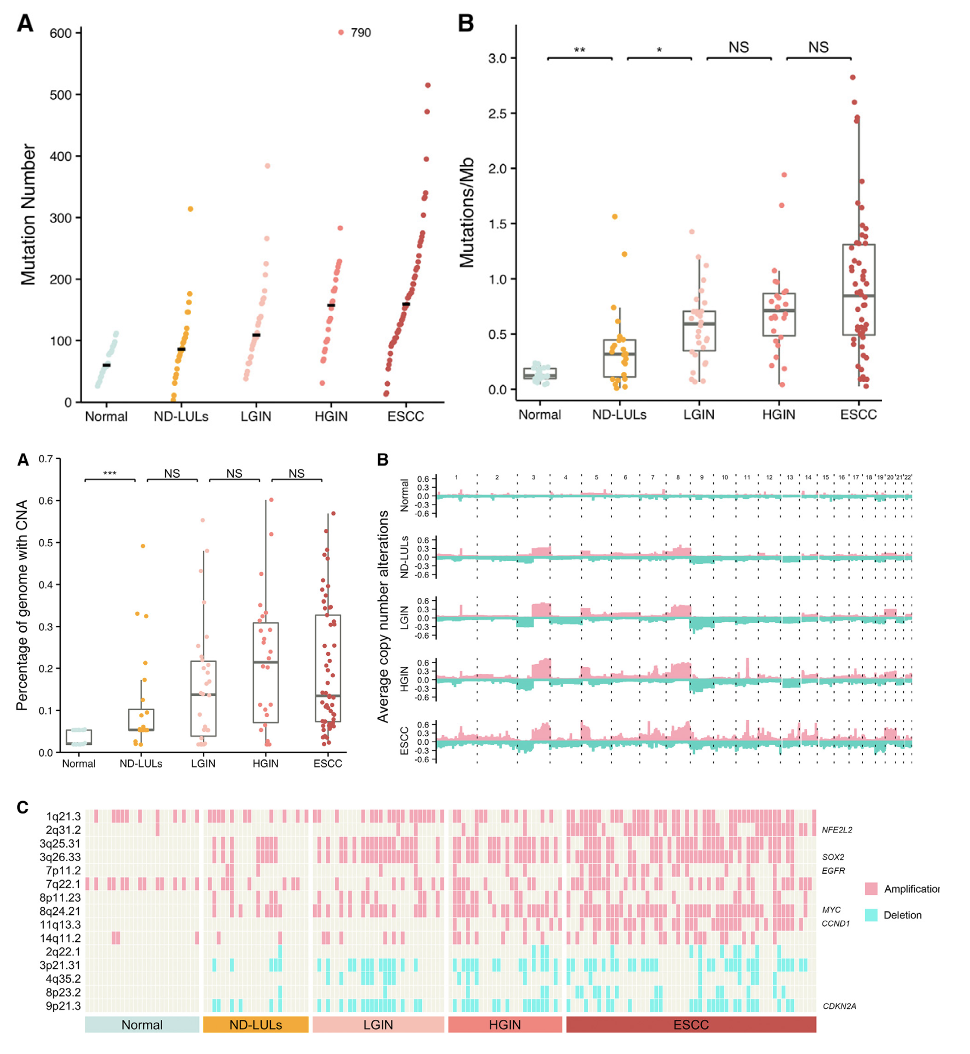
- The mutation burden per cell showed a median of 0.12, 0.32, 0.59, 0.71, and 0.87 mutations/MB per cell for normal samples, ND-LULs, LGIN, HGIN, and ESCC samples, respectively
肿瘤外显子的数据分析已经是非常标准的,基本上收集好样品后公司会完成全部的测序和数据分析流程,因为没有什么个性化的内容了。任意癌症,很容易在TCGA数据库下载到其肿瘤外显子队列的somatic突变信息,包括SNV和CNV,这样的话,除非大家感兴趣的疾病并不在TCGA数据库里面,或者说具有其它特殊性(比如人种),否则都不建议继续设计这样的简单的课题了。而且肿瘤外显子价格也不便宜,数据分析也对绝大部分初学者来说不友好。在全新服务器配置肿瘤外显子数据处理环境。流程步骤: - 获取fastq文件(aspera从ebi的ena数据库下载,prefetch从ncbi的sra数据库下载,自己测序)
- fastqc+trim-galore 质量控制
- baw的比对
- GATK工具套件一站式处理
- mutect2看配对样品的somatic突变
- CNVkit2看配对样品的拷贝数变异
- 其它
如果仅仅是为了得到队列的somatic突变信息,包括SNV和CNV,如果是样品数量太少了, 很难得到可以讲故事的点,课题就搁浅了。。。。
