有一个烂梗,说的就是癌症相关基因,详见: 『重磅!最新发现癌症相关基因』真相:基本找不着跟癌症无关的基因 ,也就是说大家看到了各式各样的文献都“恬不知耻”的落脚点是自己研究的基因在癌症领域多么多么在重要,要么是抑癌基因要么是原癌基因,跟癌症的发生发展,转移复发,耐药或多或少有相关性!
虽然是烂梗, 但是确实绝大部分研究确实是到癌症相关基因就可以发表了,都是为了自己的研究成果的发表“不惜一切代价”。
比如发表于《HEPATOLOGY》杂志的2011的文章:《Genome-Wide Copy Number Analyses Identified Novel Cancer Genes in Hepatocellular Carcinoma》,就是使用了affymetrix的SNP6.0芯片,针对58 paired HCC and nontumor tissues,进行肿瘤拷贝数变化的探索:
affymetrix的SNP6.0芯片数据分析也是拿到了bed格式的segment文件,然后使用GISTIC这样的软件进行somatic的CNV分析并且映射到基因组区域(主要是cytoband),如下所示:
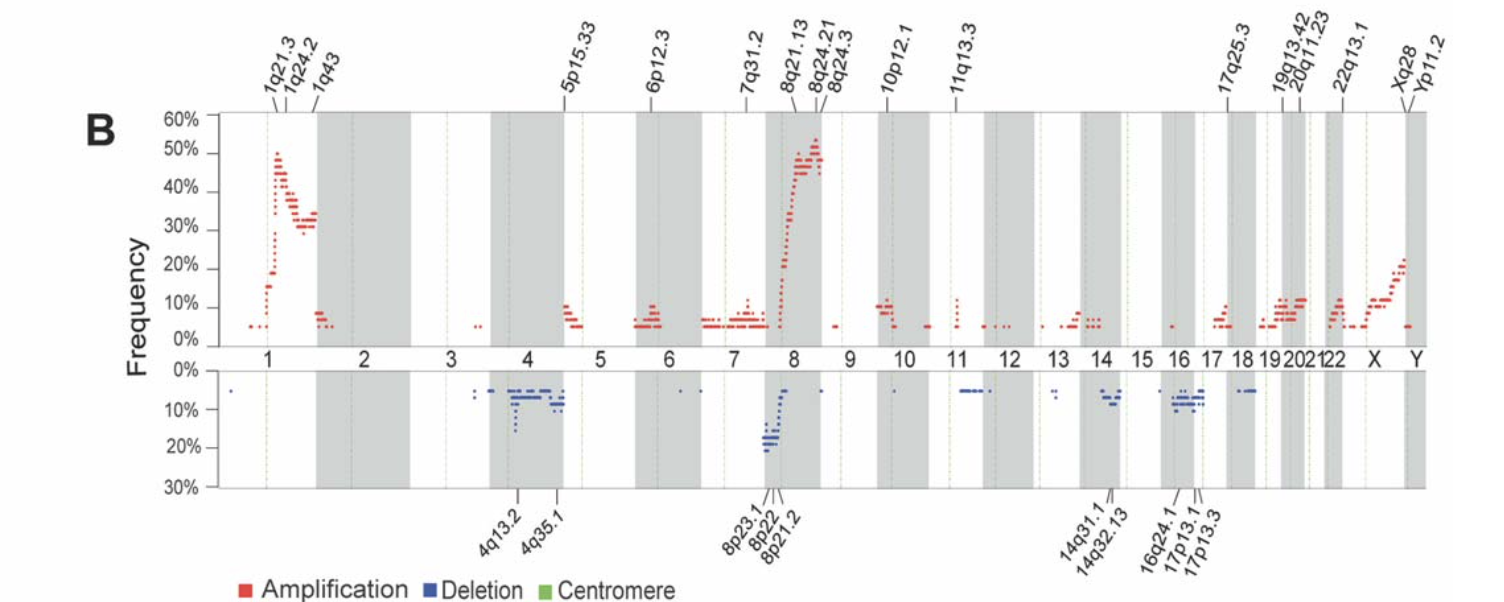
Accordingly, a total of 1,241 significant CNAs were obtained, including 963 amplifica- tions and 278 deletions (Fig. 1B).
上面的图很清晰的展示了,但是没有具体到基因,所以可以进行如下所示的整理:
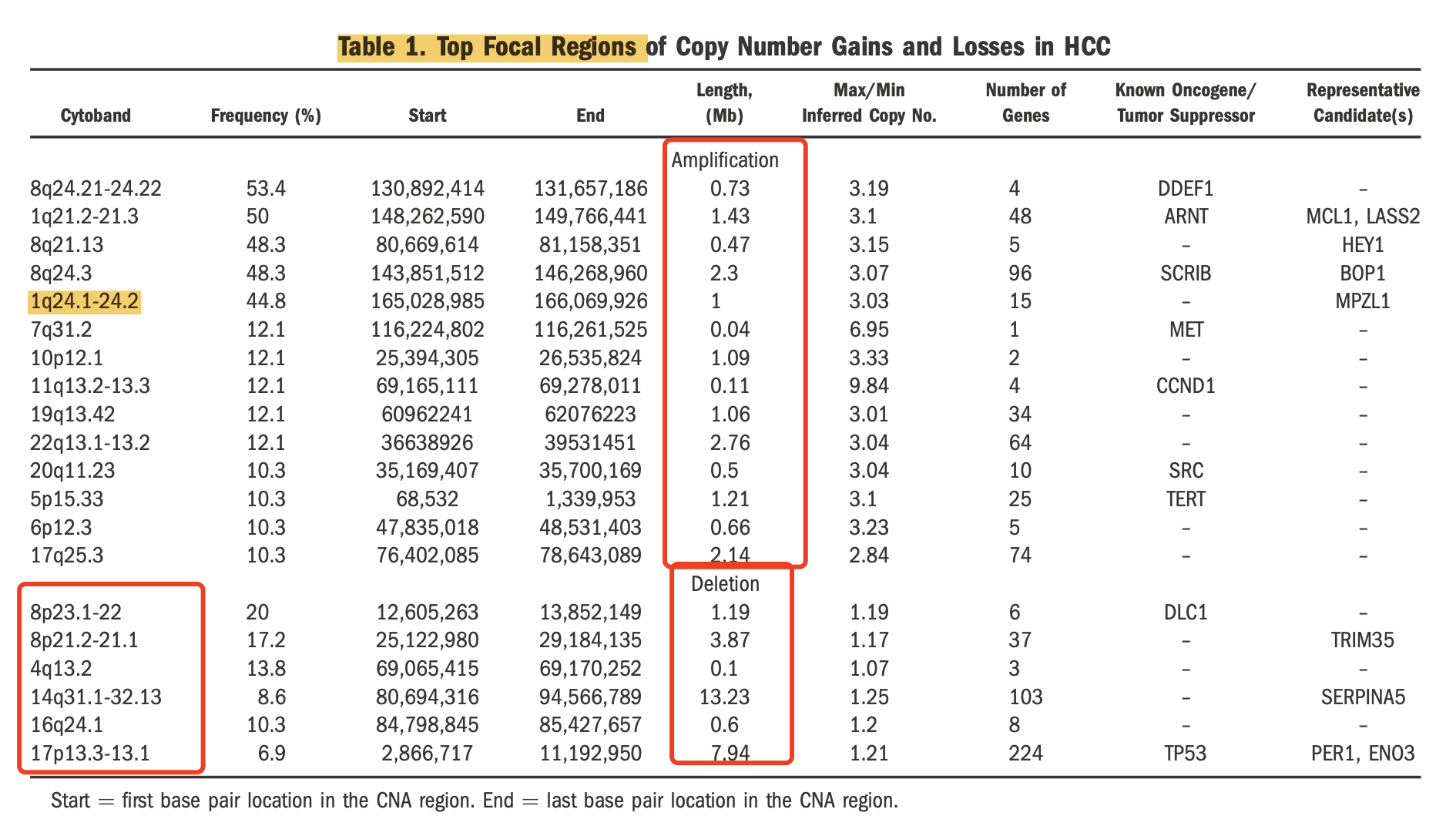
其实每个拷贝数片段什么远不止一两个基因,但是一般来说会优先展示癌症相关基因!区域是按照样品突变数量来排序的,The most frequently amplified region observed was 8q24.21-24.22, which occurred in 53.4% of samples and targets the known oncogenes MYC, DDEF1, and MLZE.
因为文章发表于2011,属于比较早期的组学文章,所以绝大部分数据分析结果都是当时的新发现,比如 The most commonly deleted loci included DLC1 at 8p23.1-8p22 and a previously unreported tripartite motif-containing 35 (TRIM35) deletion at 8p21.2-8p21.1.
拷贝数变异的分析到此为止了,但是《HEPATOLOGY》杂志毕竟是该领域的权威,还是需要再做一点,这个时候研究者们选择了 转录水平的表达量研究:49 paired HCC and nontu- mor tissues 表达量矩阵
- a total of 1,409 differentially expressed genes (DEGs) were obtained.
- the list of genes located in the 1,241 aberrant regions was matched with the DEG list.
两个组学技术交集如下所示: - 228 exhibiting increased expres- sion in the amplified regions
- 134 showing decreased expression in the deleted regions
这些上下调的差异基因进行热图可视化:
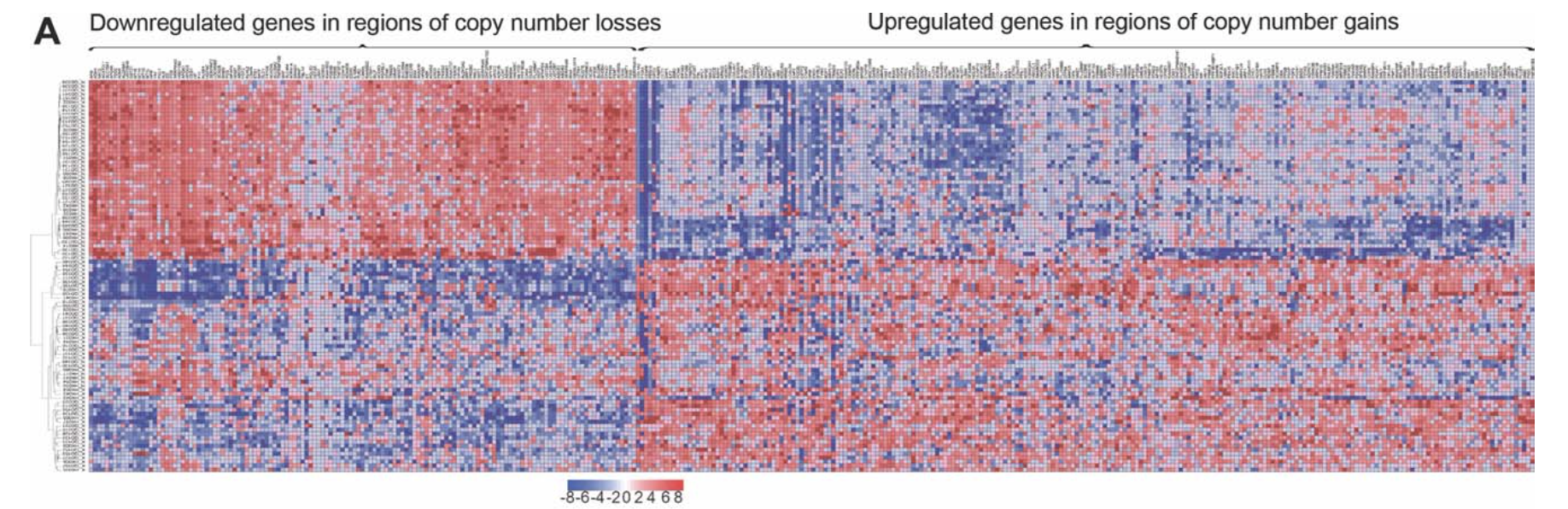
最后作者还有大量工作是验证 tripartite motif-containing 35 (TRIM35) 这个基因的功能,我不喜欢看这样的图表,就不解读啦!
可惜的是,这个研究发表的比较早,已经是十年前了,那个时候并不强调上传数据,理论上需要SNP6.0的CEL文件,方便大家对该数据进行重新分析!学徒作业
大家可以去TCGA数据库拿到HCC的拷贝数变异数据分析文件,看看是不是同样的拷贝数扩增和缺失。
另外, 推荐阅读:《美国医学遗传学会对基因芯片拷贝数变异结果解读指南》,以及中华临床医师杂志(电子版)2013 年 7 月第 7 卷第 14 期 的 《DNA 拷贝数变异及其研究进展》,可以帮助你获得一些拷贝数变异芯片的背景知识。