前些日子我们《生信技能树》的工程师做了一个ATAC-seq的项目,给客户汇报结果的时候,照例提供了全套代码。不过这次是从fq文件开始,所以大量代码都是在Linux平台的命令行而已,虽然给了客户全部的代码,但是客户直接说不想学,问有没有基于R的实现方式。
虽然有点难度,但其实确实是可以的,对生信工程师来说,就是整理流程(把Linux命令替换成为R语言代码)工作量比较大。如果大家感兴趣而且确实有需求,不妨看看这个文档:《RNASeqR : RNA-Seq analysis based on one independent variable》
链接是:https://bioconductor.org/packages/devel/bioc/vignettes/RNASeqR/inst/doc/RNASeqR.html
首先是基于Linux的ngs组学
Linux本身门槛还是有的,一般来说,每个阶段都需要至少一天以上的学习:
- 第1阶段:把linux系统玩得跟Windows或者MacOS那样的桌面操作系统一样顺畅,主要目的就是去可视化,熟悉黑白命令行界面,可以仅仅以键盘交互模式完成常规文件夹及文件管理工作。
- 第2阶段:做到文本文件的表格化处理,类似于以键盘交互模式完成Excel表格的排序、计数、筛选、去冗余,查找,切割,替换,合并,补齐,熟练掌握awk,sed,grep这文本处理的三驾马车。
- 第3阶段:元字符,通配符及shell中的各种扩展,从此linux操作不再神秘!
- 第4阶段:高级目录管理:软硬链接,绝对路径和相对路径,环境变量。
- 第5阶段:任务提交及批处理,脚本编写解放你的双手。
- 第6阶段:软件安装及conda管理,让linux系统实用性放飞自我。
如果你并不想学Linux,这个《RNASeqR : RNA-Seq analysis based on one independent variable》里面的R语言替代方案值得拥有!
第一步是测序数据的质量控制
这个流程里面是选择systemPipeR包替换了我们的fastqc软件,如果是在Linux环境,理论上我们的命令应该是:
ls -lh |cut -d" " -f 5-
197K 2月 20 10:36 SRR10574381_1.fastq.gz
208K 2月 20 10:36 SRR10574381_2.fastq.gz
198K 2月 20 10:36 SRR10574382_1.fastq.gz
201K 2月 20 10:36 SRR10574382_2.fastq.gz
206K 2月 20 10:36 SRR10574383_1.fastq.gz
-------
因为是双端测序,所以trim_galore命令需要加上—paired 的参数哦!如果样本量比较少,就直接 nohup到后台 一次性运行全部的样品:
cd $HOME/rna/test/small_raw
mkdir ../cleanData
ls *gz|cut -d"_" -f 1|sort -u |while read id;do
nohup trim_galore -q 25 --phred33 --length 36 --stringency 3 --paired -o ../cleanData ${id}*.gz &
done
如果样本量比较多,就控制一下每次并行的时候项目提交的数量:
ls *gz|cut -d"_" -f 1|sort -u |while read id;do
echo trim_galore -q 25 --phred33 --length 36 --stringency 3 --paired -o ../cleanData ${id}_*.gz
done > trim_galore.sh
for i in {0..29};do ( nohup bash ../submit.sh trim_galore.sh 30 $i 1>log.trim_galore.$i.txt 2>&1 & ) ;done
# 如果是单端 :
ls *gz| sort -u |while read id;do
echo trim_galore -q 25 --phred33 --length 36 --stringency 3 -o ../cleanData ${id}
done > trim_galore.sh
这样就是每次并行10个样品,相当于批处理啦,其中 submit.sh 脚本内容如下所示;
cat $1 |while read id
do
if((i%$2==$3))
then
$id
fi # check the number1 number2
i=$((i+1))
done
# for i in {0..9};do ( nohup bash ../submit.sh trim_galore.sh 10 $i 1>log.trim_galore.$i.txt 2>&1 &) ;done
全部的 trim_galore 命令运行完毕后,得到的clean的fq文件如下:
$ ls -lh *gz |cut -d" " -f 5-
173K 2月 20 10:40 SRR10574381_1_val_1.fq.gz
188K 2月 20 10:40 SRR10574381_2_val_2.fq.gz
177K 2月 20 10:40 SRR10574382_1_val_1.fq.gz
183K 2月 20 10:40 SRR10574382_2_val_2.fq.gz
152K 2月 20 10:40 SRR10574383_1_val_1.fq.gz
160K 2月 20 10:40 SRR10574383_2_val_2.fq.gz
71K 2月 20 10:40 SRR10574384_1_val_1.fq.gz
----
这个时候,还可以使用 fastqc软件看看raw和clean的fastq软件的测序质量情况。
第二步是比对,仍然选择hisat2或者star
仍然是需要参考基因组,需要构建索引,需要一个个样品的比对。对应的Linux命令是:
cd ~/rna/test/cleanData
indexPrefix=/home/data/server/reference/index/hisat/hg38/genome
ls *gz|cut -d"_" -f 1|sort -u |while read id;do
nohup hisat2 -p 1 -x $indexPrefix -1 ${id}*_1_val_1.fq.gz -2 ${id}*_2_val_2.fq.gz -S ${id}.hisat.sam &
done
# sam文件转bam
ls *.sam|while read id ;do (nohup samtools sort -O bam -@ 2 -o $(basename ${id} ".sam").bam ${id} & );done
# 这个过程会输出大量中间文件
rm *.sam
# 为bam文件建立索引
ls *.bam |xargs -i samtools index {}
sam文件转换为bam文件
因为sam文件太耗费磁盘空间,转为二进制的bam文件,这个时候是Rsamtools包替代了samtools软件的命令行操作。
StringTie的组装
这个其实是可选了,比如发表于2021的综述《Best practices on the differential expression analysis of multi-species RNA-seq》就并没有提到它:
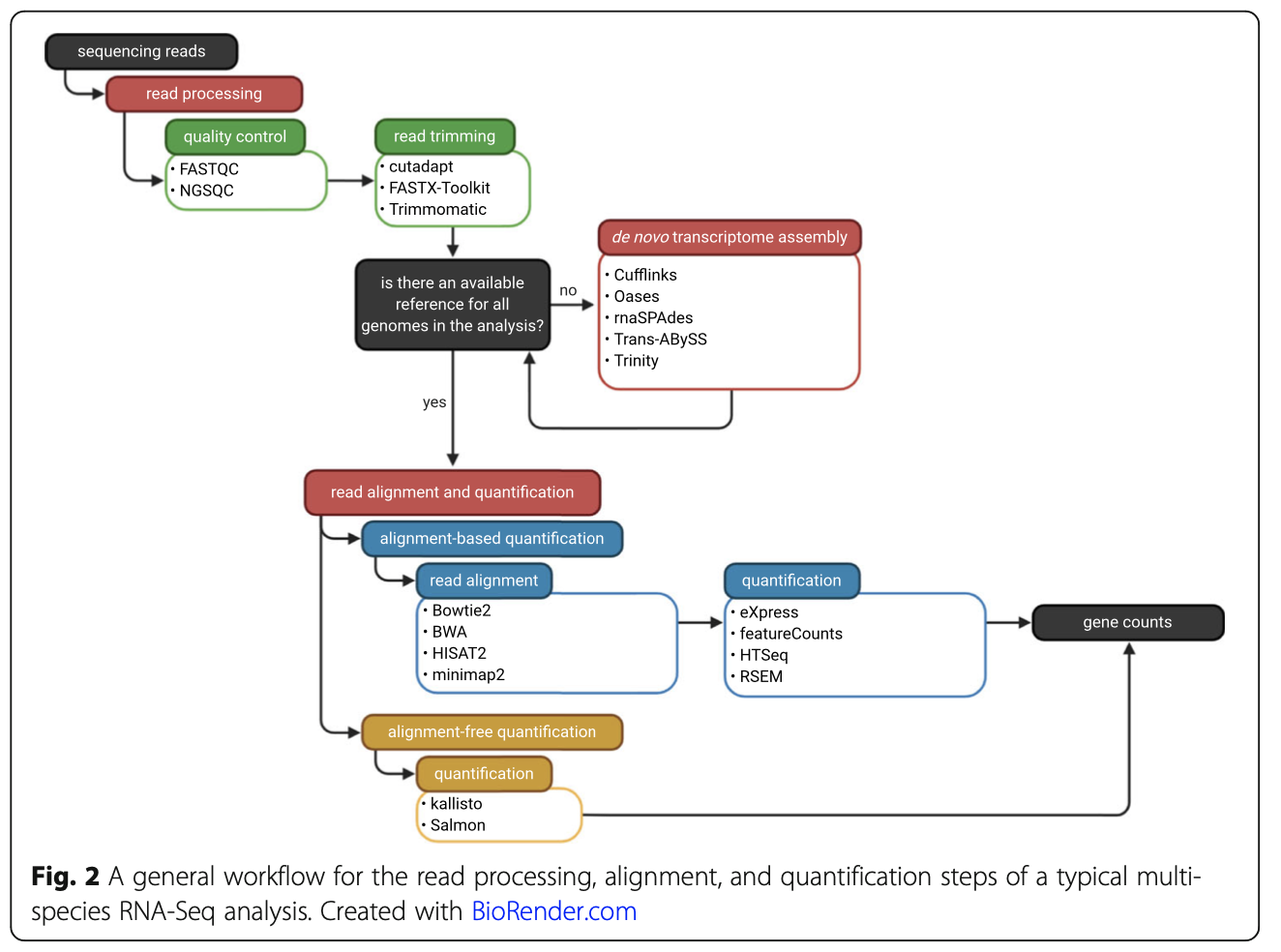
接下来是基于R语言的统计可视化
再怎么强调生物信息学数据分析学习过程的计算机基础知识的打磨都不为过,我把它粗略的分成基于R语言的统计可视化,以及基于Linux的NGS数据处理:
把R的知识点路线图搞定,如下:
- 了解常量和变量概念
- 加减乘除等运算(计算器)
- 多种数据类型(数值,字符,逻辑,因子)
- 多种数据结构(向量,矩阵,数组,数据框,列表)
- 文件读取和写出
- 简单统计可视化
- 无限量函数学习
表达量矩阵的一系列可视化
密度图,箱线图,小提琴图,PCA图,相关性图,这些本来就是R语言,属于下游分析,所以并不需要Linux环境。
“ballgown” Analysis Based on FPKM Value
同样的,属于可选!
“DESeq2” Analysis Based on Read Count
核心代码超级简单!
一步法运行全部流程
如果你觉得前面的步骤有点繁琐,在你把准备工作做好了的前提下是可以一句话完成整个流程,代码如下所示:

但真实情况下,你很难完全准备好全部的输入数据文件和软件安装,除非你本来就是熟练工。但是熟练工通常是并不会选择这个包,自己Linux命令肯定是玩的很溜!
作者提供4个成功案例
都在:https://github.com/Kuanhao-Chao/RNASeqR_analysis_result
如下所示:
Homo sapiens
GEO : GSE100075
BioProject : PRJNA390636
SRA : SRP109286
Homo sapiens
GEO : GSE50760
BioProject : PRJNA218851
SRA : SRP029880
Sample : metastasized_cancer vs normal_colon
Homo sapiens
GEO : GSE50760
BioProject : PRJNA218851
SRA : SRP029880
Sample : primary_colorectal_cancer vs normal_colon
Saccharomyces cerevisiae
GEO : GSE80357
BioProject : PRJNA318684
SRA : SRP073391
Sample : amphotericin_B (0.03 µg/mL) vs control
值得注意的是,即使是基于R的流程搭建,其实也是对操作系统有要求,不能运行在Windows平台哦。所以仍然是不太可能使用自己的笔记本电脑就跑ngs的上游流程哈。