最近有粉丝在我b站的数据挖掘视频课程发弹幕吐槽我授课时候作为例子的火山图不怎么好看,希望我提高一下自己的神秘,课程是:三年前的数据挖掘课程(TNBC表达矩阵探索)
- https://m.bilibili.com/video/BV1dy4y1C7jz 当时有人编辑修饰视频,加工,耗费了很多精力
- 配套代码在GitHub哈:https://github.com/jmzeng1314/GSE76275-TNBC
- TCGA数据库挖掘,代码在:https://github.com/jmzeng1314/TCGA_BRCA
- GTEx数据库挖掘,代码在:https://github.com/jmzeng1314/gtex_BRCA
- METABRIC数据库挖掘,代码在:https://github.com/jmzeng1314/METABRIC
- 学徒配套笔记:https://mp.weixin.qq.com/s/OivyB28Gs9aZ6f7VX11SFw
确实,颜值即正义,所以我干脆去搜索了一下解决方案,果然发现了一个成熟的轮子:
而且这个包的文档呢,超级详细:https://www.bioconductor.org/packages/release/bioc/vignettes/EnhancedVolcano/inst/doc/EnhancedVolcano.html

首先是安装和加载它
if (!requireNamespace('BiocManager', quietly = TRUE))
install.packages('BiocManager')
BiocManager::install('EnhancedVolcano')
library(EnhancedVolcano)
输入数据
其实只需要是 a single data-frame, data-matrix, or tibble of test results 即可,关键是需要有 point labels, log2FC, and adjusted or unadjusted P values. 这些列,这些信息是绘制火山图的必备!也就是说不管是基因芯片的表达量矩阵的差异分析,还是RNA-seq测序数据的差异分析,拿到的结果,都是可以无缝连接到火山图函数。
不过这个火山图的默认值有点夸张:The default cut-off for log2FC is >|2|; the default cut-off for P value is 10e-6,不过使用它的示例数据是绰绰有余啦!
示例数据来源于下面的代码 :
library(airway)
library(magrittr)
data('airway')
airway$dex %<>% relevel('untrt')
## Annotate the Ensembl gene IDs to gene symbols:
ens <- rownames(airway)
library(org.Hs.eg.db)
symbols <- mapIds(org.Hs.eg.db, keys = ens,
column = c('SYMBOL'), keytype = 'ENSEMBL')
symbols <- symbols[!is.na(symbols)]
symbols <- symbols[match(rownames(airway), names(symbols))]
rownames(airway) <- symbols
keep <- !is.na(rownames(airway))
airway <- airway[keep,]
# Conduct differential expression using DESeq2 in order to create 2 sets of results:
library('DESeq2')
dds <- DESeqDataSet(airway, design = ~ cell + dex)
dds <- DESeq(dds, betaPrior=FALSE)
res <- results(dds,
contrast = c('dex','trt','untrt'))
res <- lfcShrink(dds,
contrast = c('dex','trt','untrt'), res=res, type = 'normal')
复制粘贴即可运行,对一个RNA-seq的表达量矩阵,进行DESeq2包的差异分析,后续直接火山图可视化。
核心函数就是EnhancedVolcano
代码超级简单:
EnhancedVolcano(res,
lab = rownames(res),
x = 'log2FoldChange',
y = 'pvalue')
出图如下:
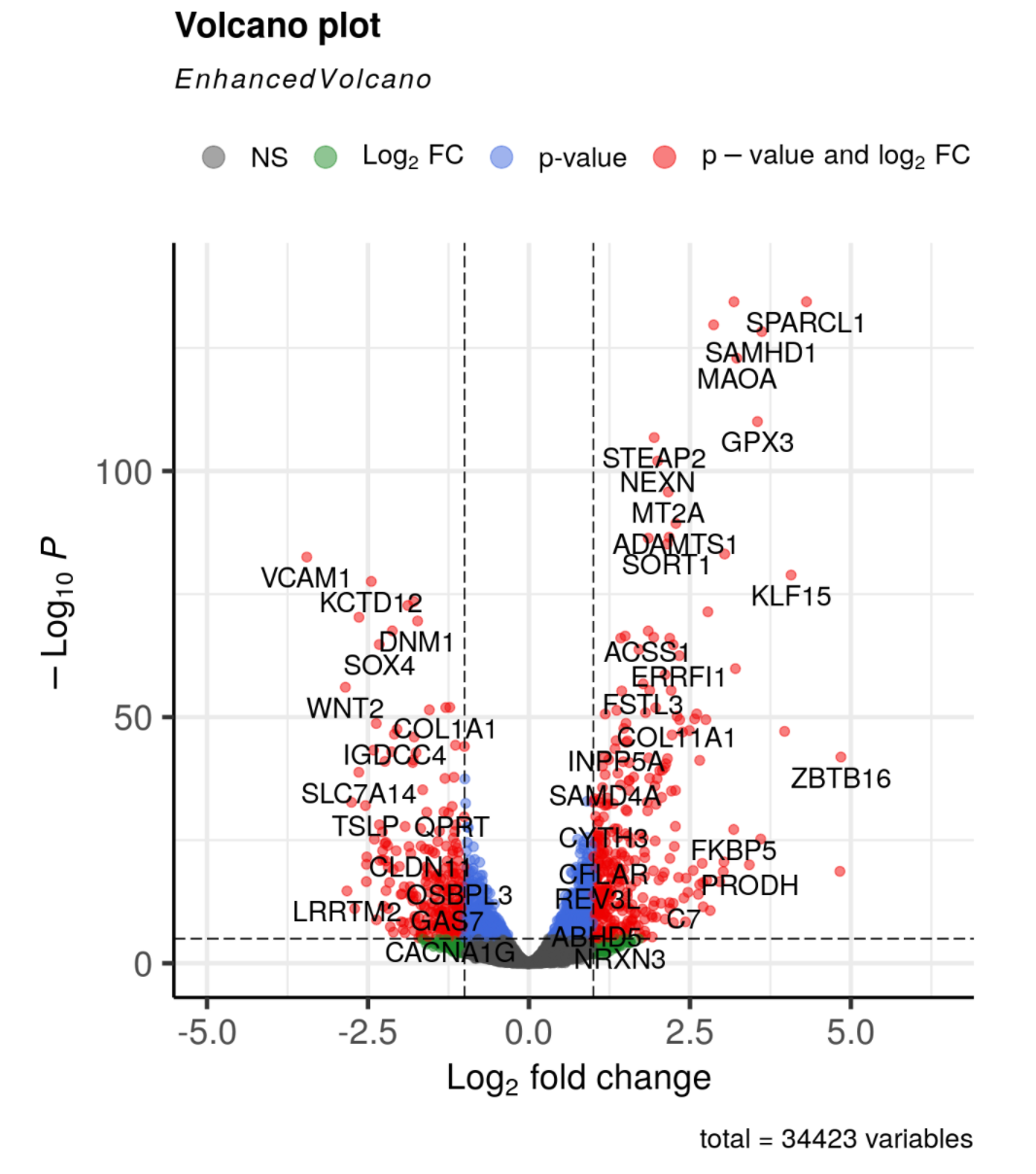
不得不说,有颜值,不过再好看的图也需要人去解释,给它一定的生物学意义。
而且,这个包函数的用法我仅仅是起了个头,后面还有超级多丰富的用法,感兴趣的朋友完全是可以自己看文档哦,不要做伸手党!