最近在有粉丝求助,他的scATAC-seq数据的分析,使用 scATAC pro 这个软件得到 cluster 里面的细胞类型好少。
其实scATAC-pro 软件呢于2020年4月发表,在一个综合性的流程软件,数据分析的上下游打通了。包括上游拿到peaks的信号矩阵,以及下游的降维聚类分群和注释。如果你的scATAC-seq数据本身就是10X仪器的,那么就直接使用官方提供的cellranger-atac软件即可完成上游分析拿到peaks的信号矩阵。不过10X仪器scATAC-seq数据出现的比较晚,于2019年8月发表在《Nature Biotechnology》杂志的文章《Massively parallel single-cell chromatin landscapes of human immune cell development and intratumoral T cell exhaustion》,链接是:https://www.nature.com/articles/s41587-019-0206-z 应该是 10X的scATAC产品首秀,我还记得当初也是刷爆了我的朋友圈。
虽然我们现在有了成熟的商业产品,以及成熟的分析工具,但是我们仍然不能忘记没有这些前提的时候,我们是如何拿到scATAC-seq数据以及分析它的。比如于2018年8月发表在《CELL》杂志的文章,标题是:《A Single-Cell Atlas of In Vivo Mammalian Chromatin Accessibility》,链接是:https://doi.org/10.1016/j.cell.2018.06.052 就是草莽阶段的单细胞ATAC研究。
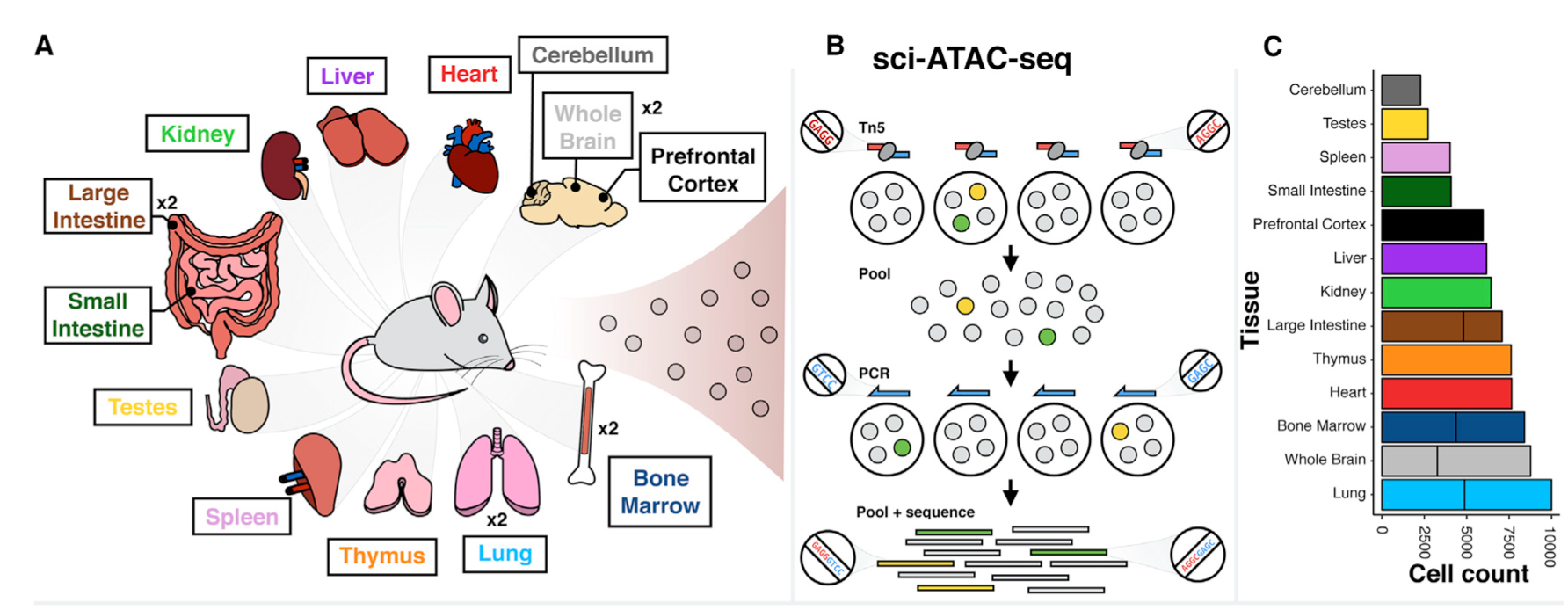
数据公开可以获取
在:https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE111586
GSM3034622 Bone Marrow 62016
GSM3034623 Bone Marrow 62216
GSM3034624 Cerebellum 62216
GSM3034625 Large Intestine A 62816
GSM3034626 Large Intestine B 62816
GSM3034627 Heart 62816
GSM3034628 Small Intestine 62816
GSM3034629 Kidney 62016
GSM3034630 Liver 62016
GSM3034631 Lung A 62216
GSM3034632 Lung B 62216
GSM3034633 Prefrontal Cortex 62216
GSM3034634 Spleen 62016
GSM3034635 Testes 62016
GSM3034636 Thymus 62016
GSM3034637 Whole Brain A 62216
GSM3034638 Whole Brain A 62816
上游分析拿到peaks的信号矩阵
如果感兴趣上游数据处理流程,也可以从fastq文件开始,链接在:https://www.ncbi.nlm.nih.gov/bioproject/PRJNA437471,因为并不是 10X仪器的数据,所以没办法使用10X官方提供的cellranger-atac软件来完成上游分析拿到peaks的信号矩阵。文章给出来的流程是:
- Trimmomatic
- bowtie2 with ‘-X 2000 −3 1’
- filtered out read pairs that did not map uniquely to autosomes or sex chromosomes with a mapping quality of at least 10 using Samtools
- removed PCR duplicates
- macs2 callpeak command with the following parameters: ‘–nomodel–keep-dup all–extsize 200–shift −100’.
下游分析是:降维聚类分群
有了peaks的信号矩阵,下游的降维聚类分群就比较容易了,只不过是考验你的计算机资源。因为scATAC pro 这个软件在2018年还没有发表,所以研究者肯定也是自己的流程啦,步骤如下;
- Finally, we generated a lower dimensional representation of the data by including the first 50 dimensions of the singular value decomposition of this TF-IDF-transformed matrix. This representation was then used as input for the Rtsne package in R
- To identify clusters of cells in this two dimensional representation of the data, we used the Louvain clustering algorithm implemented in Seurat
- Through this round of “iterative” t-SNE, we identified a total of 85 distinct clusters.
得到的细胞亚群如下:
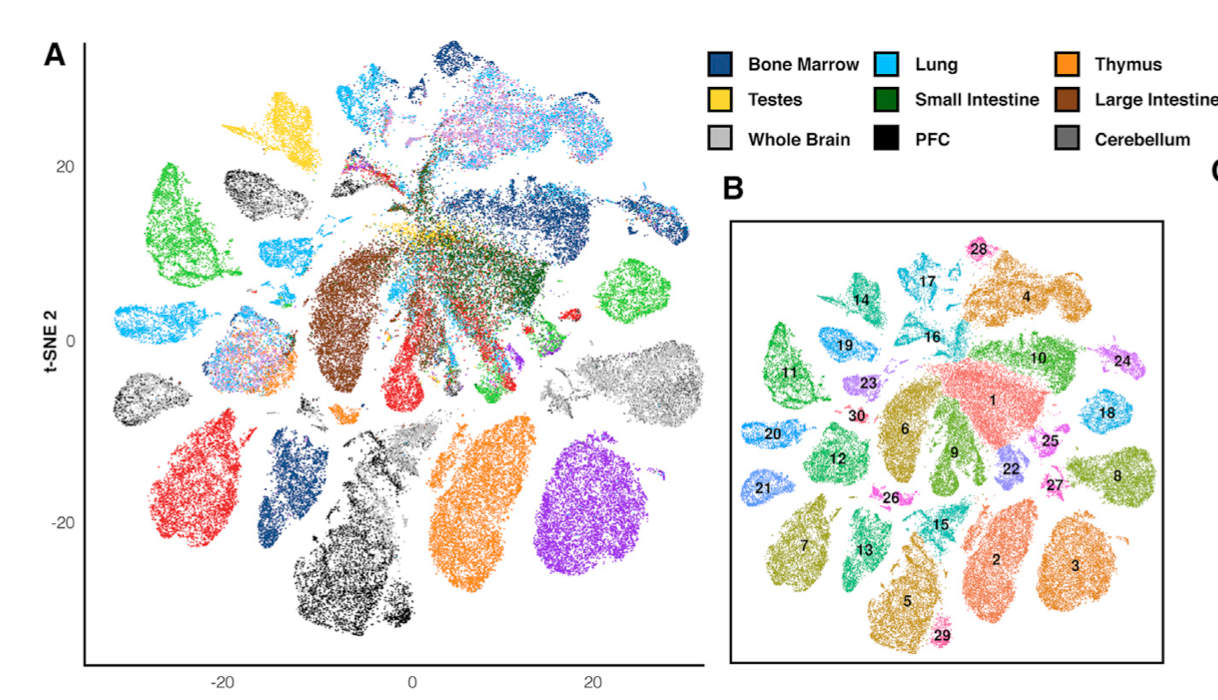
细胞亚群的生物学功能注释非常困难
研究者把小鼠的13个不同组织器官的超10万个细胞分成了85个亚群,但是想把这些亚群映射到生物学功能亚群就遇到了麻烦!
Cell-type identification from scATAC-seq is more challenging than from scRNA-seq, largely because we have fewer guideposts in the literature.
- After curating a set of marker genes from the literature corresponding to expected cell types (Table S1), we estimated their activity scores in each of the 85 clusters (see Additional Resources). This enabled the assignment of 51 clusters to a specific cell type.
- We therefore developed a classifier trained on the accessibility profiles of marker-associated cells that allowed us to assign cell types to 12 additional clusters
注释结果如下:

思考题
现在是2021的3月底了,你已经知道了 scATAC pro 这个软件,那么你会如何处理这个文章的peaks的信号矩阵呢?