目前单细胞转录组以10X公司为主流,我们也是在单细胞天地公众号详细介绍了cellranger全部使用细节及流程,大家可以自行前往学习,如下:
- 单细胞实战(一)数据下载
- 单细胞实战(二) cell ranger使用前注意事项
- 单细胞实战(三) Cell Ranger使用初探
- 单细胞实战(四) Cell Ranger流程概览
- 单细胞实战(五) 理解cellranger count的结果
但是这个两年前的系列笔记是基于V2,V3版本的cellranger,在2020的7月我看到了其更新到了V4,也里面写了一个总结,见:cellranger更新到4啦(全新使用教程)
没想到这2020才结束,它马上有升级了,目前是Cell Ranger - 5.0.1 (December 16, 2020),好像没有改变,其实并没有必要再总结一个笔记,但是反正也没有啥事情,就水一个吧!反正再怎么水,也比不上周运来的高产,仅仅是一个seurat的V4更新,就可以写十几篇笔记:
- Seurat Weekly 专栏总结(送圣诞礼物)
- Seurat Weekly NO.0 || 开刊词
- Seurat Weekly NO.1 || 到底分多少个群是合适的?!
- Seurat Weekly NO.2 || 我该如何取子集?
- Seurat Weekly NO.3 || 定制可视化
- Seurat Weekly NO.4 || 高效数据管理
- Seurat Weekly NO.05 || 大数据集处理之Pseudocell
- Seurat Weekly NO.06 || Scanpy2Seurat
我们的这个cellranger更新,我就一篇就写完了!
软件下载及安装
同样的,需要自己简单注册后就可以获取wget下载地址,因为版权的问题,我这里就不复制粘贴出来地址啦,反正简单填写邮箱即可注册拿到地址。
注册网页是: https://support.10xgenomics.com/single-cell-gene-expression/software/downloads/latest
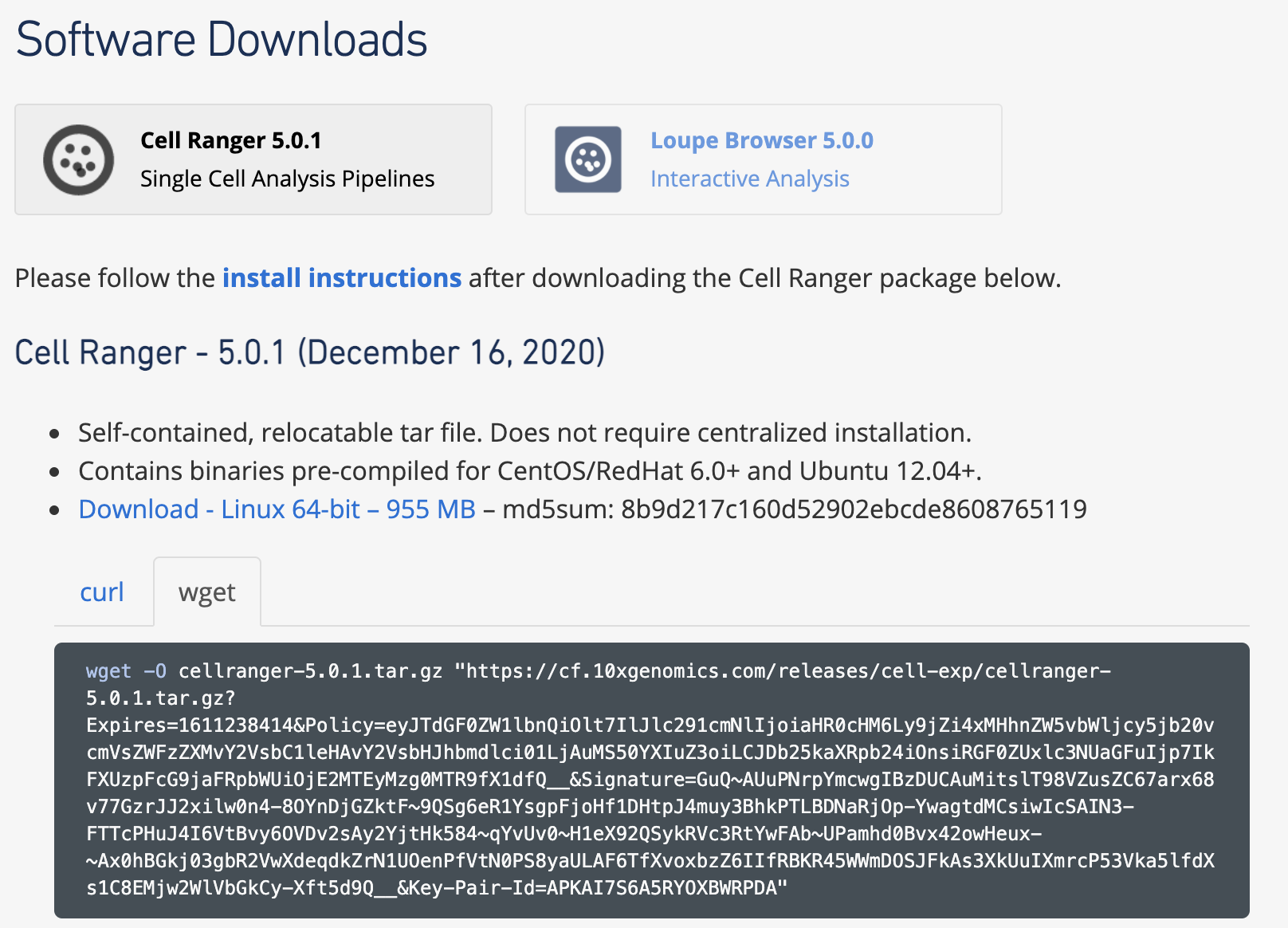
一般来说,软件以及配套的数据库都需要下载,下载速度呢,就取决于你自己的网路情况啦,反正在中国大陆地区下载肯定会很慢的,建议nohup到后台,等一个晚上即可。
我看了看,在中国大陆广东省,可以跑满我的联通200M宽带,不存在速度问题。
再次认识10x的fastq数据文件
官网给指出来了文件名规则:https://support.10xgenomics.com/single-cell-gene-expression/software/pipelines/2.0/using/fastq-input#wrongname ,如果你的fastq数据不是这样命名,就需要自行更改过来了,我上面截图的就是需要修改的,因为里面混入了AK这样的编号。
如果要理解这3个文件的区别,同理,也是需要自己去学习了解10x的原理,我这里就不再赘述:
-
首先,1-26个cycle就是测序得到了26个碱基,先是16个Barcode碱基,然后是10个UMI碱基;
-
然后,27-34这8个cycle得到了8个碱基,就是i7的sample index;
-
最后35-132个cycle得到了98个碱基,就是转录本reads
使用Cell Ranger
Cell Ranger主要的流程有:拆分数据 mkfastq、细胞定量 count、定量组合 aggr、调参reanalyze,还有一些小工具比如mkref、mkgtf、upload、sitecheck、mat2csv、vdj、mkvdjref、testrun。
但是,大概率上,我们只需要使用它的定量流程,就是 cellranger count 命令,教程在consult Running 10x Pipelines on FASTQ Files,主要就是需要把软件和配套的数据库文件,以及10x的fastq文件准备好(主要就是命名符合规则)。
我写了一个脚本,文件名是 run-cellranger.shn,内容如下所示 :
bin=../pipeline/cellranger-5.0.1/bin/cellranger
db=../pipeline/refdata-gex-GRCh38-2020-A
ls $bin; ls $db
fq_dir=/home/data/project/10x/raw
$bin count --id=$1 \
--localcores=4 \
--transcriptome=$db \
--fastqs=$fq_dir \
--sample=$1 \
--expect-cells=5000
是不是超级简单,值得注意的是我把样本名字进行了修改,全部的技术含量都体现在这个文件名修改哦,才成功运行这个 cellranger count 命令。
然后每个样本都只需要提交上面的这个脚本即可:
nohup bash run-cellranger.sh YX-Endo-Decidu 1>log-YX-Endo-Decidu.txt 2>&1 &
nohup bash run-cellranger.sh YX-PBMC-Decidu 1>log-YX-PBMC-Decidu.txt 2>&1 &
nohup bash run-cellranger.sh HSY-PBMC 1>log-HSY-PBMC.txt 2>&1 &
nohup bash run-cellranger.sh HSY-fushui 1>log-HSY-fushui.txt 2>&1 &
nohup bash run-cellranger.sh HSY-yi 1>log-HSY-yi.txt 2>&1 &
服务器配置不一样,这个cellranger count流程运行时间不一样,我 一个样本是60G的fq文件数据走这个流程是5小时。
如果你真想学会Cell Ranger - 5.0.1
首先你需要有单细胞转录组数据,其次你需要有强大的服务器。如果是普通的R语言教程,当然是可以提供测试数据,但是我们的这个单细胞转录组数据,实在是太大了,如果一定要分享,只能说是跟服务器搭配分享,如下所示:
paper-rawData-30g/SRR7722937_S1_L001_I1_001.fastq.gz
paper-rawData-30g/SRR7722937_S1_L001_R1_001.fastq.gz
paper-rawData-30g/SRR7722937_S1_L001_R2_001.fastq.gz
paper-rawData-30g/SRR7722938_S1_L001_I1_001.fastq.gz
paper-rawData-30g/SRR7722938_S1_L001_R1_001.fastq.gz
paper-rawData-30g/SRR7722938_S1_L001_R2_001.fastq.gz
paper-rawData-30g/SRR7722939_S1_L001_I1_001.fastq.gz
paper-rawData-30g/SRR7722939_S1_L001_R1_001.fastq.gz
paper-rawData-30g/SRR7722939_S1_L001_R2_001.fastq.gz
paper-rawData-30g/SRR7722940_S1_L001_I1_001.fastq.gz
paper-rawData-30g/SRR7722940_S1_L001_R1_001.fastq.gz
paper-rawData-30g/SRR7722940_S1_L001_R2_001.fastq.gz
paper-rawData-30g/SRR7722941_S1_L001_I1_001.fastq.gz
paper-rawData-30g/SRR7722941_S1_L001_R1_001.fastq.gz
paper-rawData-30g/SRR7722941_S1_L001_R2_001.fastq.gz
paper-rawData-30g/SRR7722942_S1_L001_I1_001.fastq.gz
paper-rawData-30g/SRR7722942_S1_L001_R1_001.fastq.gz
paper-rawData-30g/SRR7722942_S1_L001_R2_001.fastq.gz
你可以考虑我们的共享云服务器,来一起体验这个Cell Ranger - 5.0.1的单细胞转录组上游分析流程。