全外显子测序(WES)和RNA测序(RNA-Seq)是二代测序(NGS)的两个主要平台,其中WES主要用于发现DNA变异,而RNA-Seq的使用集中在基因表达量的测量,我在生信技能树B站都分享过这两方面数据的处理视频教程:
- 免费视频课程《RNA-seq数据分析》
- 免费视频课程《WES数据分析》
其实两者均可用于检测遗传变异,特别是在单核苷酸变异方面(SNVs)。如果大家对RNA-seq数据如何找变异位点的流程不是很清楚,可以看我们生信技能树以前的教程:- 2017年6月:RNA-seq 检测变异之 GATK 最佳实践流程
- 2019年11月:最新版针对RNA-seq数据的GATK找变异流程
然而如何从WES和RNA-Seq中检测出突变的一致性尚未得到系统的评估。2015的一个文章在肿瘤病人里面做了这样的比较:Inconsistency and features of single nucleotide variants detected in whole exome sequencing versus transcriptome sequencing: 有意思的是引用情况非常糟糕,截止到目前(2020-09-05)都不到20个引用!
在这项研究中,Vanderbilt University医学部的研究人员使用27对肿瘤样本及其匹配的正常样本的WES和RNA-Seq数据,研究了SNV检测中技术和生物学上的不一致性。他们分析了三类SNVs:- (1)仅在WES中检测到的
- (2)仅在RNA-Seq中检测到的
- (3)在两者中均检测到的。
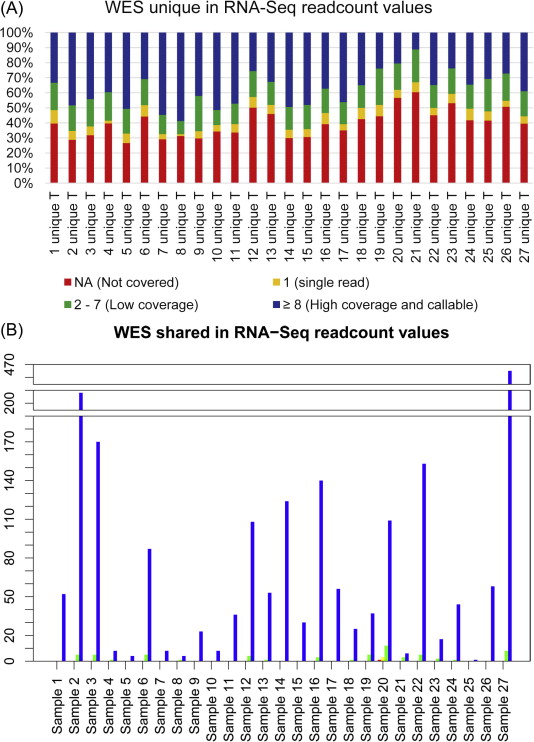
他们发现在WES和RNA-Seq中检测到的SNVs有很小的重叠(约14%),仅在WES中检测到的SNVs主要由于其低覆盖度、低表达或它们位于RNA-Seq数据中的非转录链,而只在RNA-Seq中检测到的SNVs主要因其位置超出了WES检测边界(这一部分约占71%),以及区域覆盖度低、突变等位基因覆盖率低或RNA编辑。两种技术共享的SNVs在WES和RNA-Seq中都具有较高的基因座特异性覆盖度,并且具有较高的基因表达水平。
而WES和RNA-Seq各自特有的SNVs显示出不同的核苷酸替代模式,例如55%的RNA-Seq特有的突变是从A:T → G:C(RNA编辑的一个标志)。这项研究对WES和RNA-Seq数据中获得的somatic SNVs的不一致性提供了重要的评估。
VarScan2读取 count值确定在仅在WES中检测中的 SNVs的原因。(A)堆叠柱状图展示了仅在 WES检测出的 SNVs的 RNA-Seq的 counts结果。(B)条形图展示了 RNA-Seq和 WES共同检测到的 SNVs的 counts结果。红色代表 counts值为 NA(没有覆盖),黄色代表 counts = 1,绿色代表 counts = 2-7,蓝色代表counts ≥ 8。大多数仅在WES中检测出的 SNVs不在 RNA-Seq中。
O’Brien T D, Jia P, Xia J, et al. Inconsistency and features of single nucleotide variants detected in whole exome sequencing versus transcriptome sequencing: A case study in lung cancer[J]. Methods, 2015, 83: 118-127.
来源:https://rna-seqblog.com/inconsistency-of-somatic-snvs-called-in-wes-and-rna-seq-data/
备注:
SNVs(单核苷酸变异)和SNPs(单核苷酸多态性)有所不同,SNPs既存在于肿瘤DNA中,也存在于对照DNA中,而 somatic SNV仅存在于肿瘤样本中。当然,更主流的描述其实是 germline和somatic的变异位点的描述。学徒作业
现在提供WES和RNA-seq数据的队列研究非常多,如果大家有服务器,完全可以重复一下这篇文章的分析过程,做一下同样的比较!
很久以前,生信技能树分享过台湾OSCC癌症多组学,文章是 2016年10月年发表的:APOBEC3A is an oral cancer prognostic biomarker in Taiwanese carriers of an APOBEC deletion polymorphism 就是提供WES和RNA-seq数据对外显子数据的分析
首先统计了50个OSCC病人的肿瘤癌旁配对全外显子数据的测序总览,走bwa+gatk流程,并且走mutect得到somatic mutations列表,经由 Oncotator 注释成maf文件。最终对着50个病人找到了24,051 somatic mutations 。
然后找拷贝数变异,使用的是 GATK DepthOfCOverage 计算测序深度,再用exome CNV 判断拷贝数情况。
测序策略是PE100bp, (75-Mbp target region, mean depth = 244 ± 54×),使用的测序仪和试剂盒是 HiSeq 2000 with the TruSeq PE Cluster kit v3 and TruSeq SBS kit v3对转录组数据的分析
该实验共得到39对OSCC病人的肿瘤癌旁配对转录组数据,首先检查的测序总览。
然后走标准的转录组数据分析流程: Trimmomatic+hg19+STAR+RSEM+GENCODE
然后,定义了 3548个显著的差异表达基因,阈值是 p-value < 0.05 and fold change > 2 ,只保留 transcripts per million (TPM) larger than 0.5。
大家可以下载全部的数据,走Inconsistency and features of single nucleotide variants detected in whole exome sequencing versus transcriptome sequencing:提到的图表。文末友情推荐
要想真正入门生物信息学建议务必购买全套书籍,一点一滴攻克计算机基础知识,书单在:什么,生信入门全套书籍仅需160 。
如果大家没有时间自行慢慢摸索着学习,可以考虑我们生信技能树官方举办的学习班:- 数据挖掘学习班第7期(线上直播3周,马拉松式陪伴,带你入门),原价4800的数据挖掘全套课程, 疫情期间半价即可抢购。
- 生信爆款入门-第9期(线上直播4周,马拉松式陪伴,带你入门),原价9600的生信入门全套课程,疫情期间3.3折即可抢购。
如果你课题涉及到转录组,欢迎添加一对一客服:详见:你还在花三五万做一个单细胞转录组吗?
号外:生信技能树知识整理实习生招募,长期招募,也可以简单参与软件测评笔记撰写,开启你的分享人生!另外,:绝大部分生信技能树粉丝都没有机会加我微信,已经多次满了5000好友,所以我开通了一个微信好友,前100名添加我,仅需150元即可,3折优惠期机会不容错过哈。我的微信小号二维码在:0元,10小时教学视频直播《跟着百度李彦宏学习肿瘤基因组测序数据分析》