前面我们一起学习到了单细胞转录组数据的降维聚类分群,而且拿到的亚群,也可以解释其生物学意义,见:单细胞转录组数据处理之细胞亚群注释
其实理论上细胞亚群是可以无限划分的,因为世界上没有两个一模一样的细胞,关键是要把握一个度,什么样的差异可以判定为不同细胞亚群,什么样的差异是可以容忍的细胞类群内部异质性。
有一个策略就是找出主要因素和次要因素。主要因素划分为主要亚群,比如外周血里面的T,B细胞当然是不同亚群,但是T细胞里面还可以继续划分:CD4或者CD8的T细胞,甚至继续划分, 如下图所示:
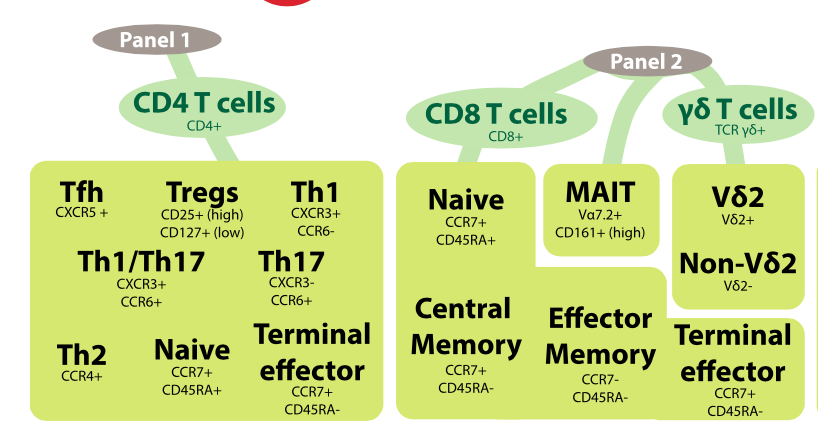
主要和次要细胞亚群同一个tSNE图展现
现在10x单细胞转录组技术一个样本出来3~8K细胞的数据,很多课题都是十几个以上样本,所以妥妥的十万多个单细胞数量级,它们走聚类分群对技术资源的消耗当然是很可观的,而且,拿到的细胞亚群数量也是惊人。比如下面这个,就分成了近100个细胞亚群。
实际上,细胞二维散点图,是没办法写全部细胞亚群的生物学功能定义的, 我们通常也是把主要细胞亚群标记上去。然后每个细胞亚群再进行继续分群。只不过是在同一个散点图上面展示。
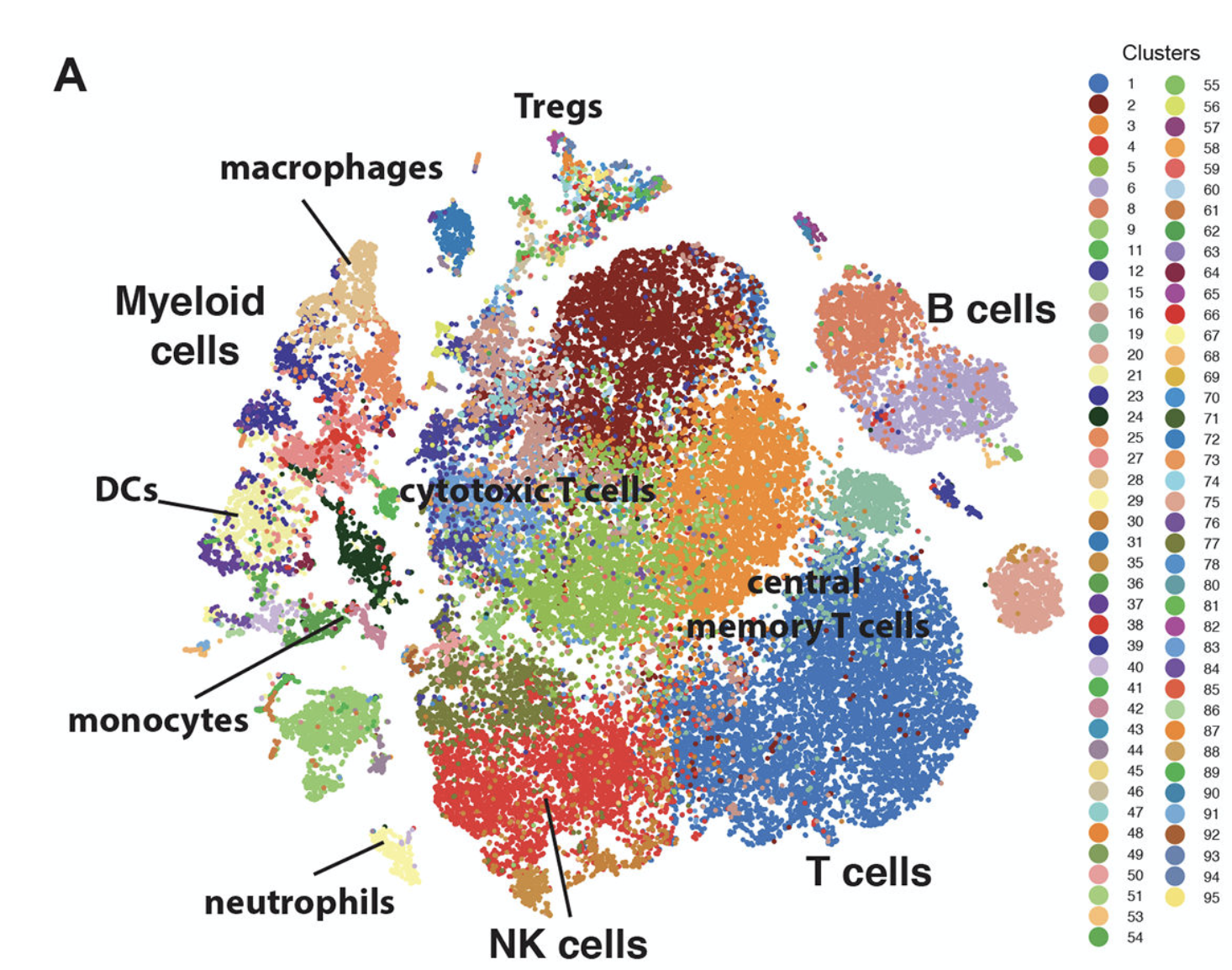
主要和次要细胞亚群以标记基因来展现
上面的散点图毕竟展示的细胞数量太多, 大多数情况下以炫酷为主,一般人很难看出来不同细胞亚群内部具体如何划分子亚群,以及不同亚群到底以什么标记基因来进行区分。
有一个策略是,把标记基因的表达量在所有亚群及子亚群里面展现,以热图形式:
e, Clustergrammer heatmap showing protein marker expression (top) in each MC (left) and the canonical annotation of these communities (right).
The dendrogram bars (light gray) indicate the clustering of MCs based on the cosine distance method in Clustergrammer.
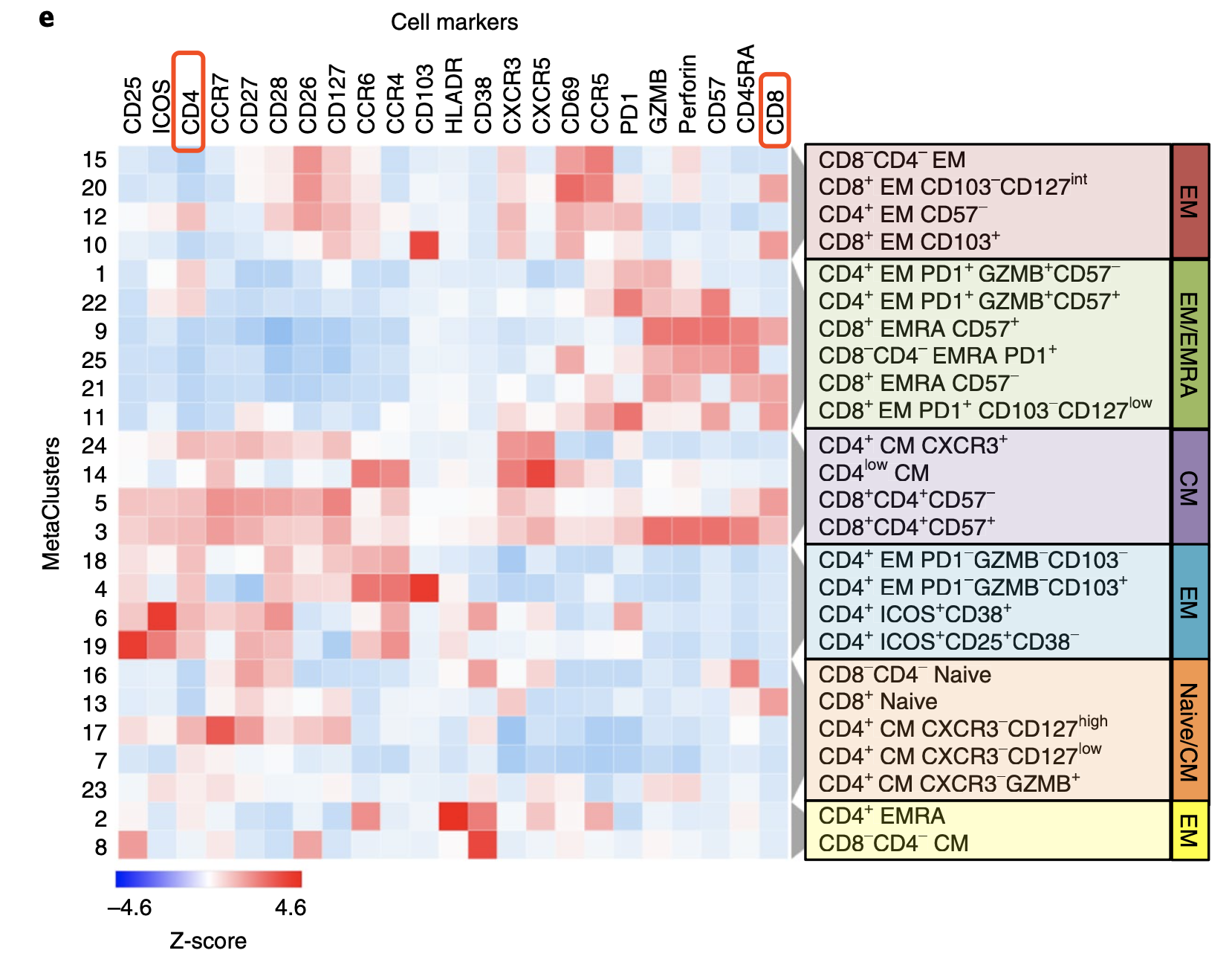
这个其实有点像我们前些天在生信技能树提到的学徒练习题:
- 在CCLE数据库里面根据指定基因在指定细胞系里面提取表达矩阵 ,
- 学徒带你一步步从CCLE数据库里面根据指定基因在指定细胞系里面提取表达矩阵进行热图可视化
后来我升级为了bodymap和Gtex数据库的,指定基因在指定组织里面的表达量热图,就需要使用代码,把同一个亚群的全部细胞表达量综合一下:mat=do.call(rbind, lapply(unique(id$SMTS), function(t){ rowMeans(dat[,id$SMTS==t]) }) )其中 id$SMTS 里面是每个细胞的亚群属性,而dat是我们的表达矩阵,所以对表达矩阵来说,依次取出每个亚群的表达矩阵子集,然后取 rowMeans,就拿到了每行的基因在该亚群的表达量平均值。
这个综合后的表达量值就可以去绘制上面的标记基因的表达量在所有亚群及子亚群里面热图。多个样本可以分开走聚类分群流程
比如中山大学的最新研究《一个人的15个器官单细胞测序数据 》,链接是:https://www.biorxiv.org/content/10.1101/2020.03.18.996975v1.full.pdf 。这里面首先有不同器官,可以分开独立走单细胞流程。然后全部的八万多细胞,分成了主要细胞群之后,仍然是可以进行每个亚群细致研究:
- we performed single-cell transcriptomes of 88,622 cells derived from 15 tissue organs of one adult donor and generated an adult human cell atlas (AHCA).
- 首先是 a total of 20,494 T cells,可以区分成为 CD4+ (7,122) and CD8+ (13,372) T cells 两个大类,然后继续每个大类细分为11 and 22细胞亚群。
- 然后是 10,655 B and plasma cells
- 还有 5,605 myeloid cells ,可以细分为7个monocyte亚群,9个macrophage亚群,一个small dendritic cell (DC) 亚群。
- 继而是 18,090 epithelial cells ,进而细分为33个亚群,值得注意的是这些上皮细胞来源于9种器官,189 genes with tissue-specific expression (FC 5, pct.1 0.2) 。
- 然后是7,137个 Endothelial cells (ECs) 细胞, including 6,863 blood endothelial cells (BECs, marked with VWF) and 274 lymphatic endothelial cells (LECs, marked with LYVE1).
- 最后是 a total of 17,835 fibroblasts and smooth muscle cells , 分成
- 14 fibroblast clusters (11,767 cells, MMP2),
- four smooth muscle cell clusters (3,201 cells, ACTA2),
- another five clusters assigned as FibSmo (2,867 cells; marked with MMP2 and ACTA2; )
单细胞测序的肺癌肿瘤免疫微环境中基质细胞
文章是:Phenotype molding of stromal cells in the lung tumor microenvironment。
基本上也是细胞分群后继续分群,文献里面公布的主要亚群标记基因是:
- endothelial cells:CLDN18, FOLR1, AQP4 and PEBP4
- endothilial:CLDN5, FLT1, CDH5 ,RAMP2
- epithelial cells: CAPS, TMEM190, PIFO,SNTN
- fibroblasts,COL1A1, Decorin (DCN) Collagen type I alpha 2 (COL1A2) and C1R 9 ; B-cells,:genes encoding the B-cell antigen receptor complex-associated protein alpha (CD79A),Immunoglobulin Kappa Constant (IGKC), Immunoglobulin Lambda Constant 3 (IGLC3) and Immunoglobulin Heavy Constant Gamma 3 (IGHG3)
- myeloid cells, genes encoding Lysozyme (LYZ), the Macrophage Receptor With Collagenous Structure (MARCO), CD68 and CD16a, the Fc Fragment Of IgG Receptor IIIa (FCGR3A) 10
- T-cells:(TRAC, TRBC1 and TRBC2).
然后对每个亚群细胞继续划分,比如 1592 个内皮细胞进行重新聚类分为 6 个亚群: - Cluster 1:正常组织来源,MT2A +
- Cluster2 :未发现标记基因,在进一步分析时丢弃。
- Cluster3 :血液内皮细胞,IGFBP3 +,主要来自肿瘤样本
- Cluster4:血液内皮细胞,SPRY1 +,肿瘤样本
- Cluster5:血液内皮细胞, EDNRB +
- Cluster6:淋巴管内皮细胞,PDPN 和 PROX1
可以看到这个时候的亚群,已经太深入,所以命名的时候就不一定都是有很明确生物学功能的。文末友情宣传
强烈建议你推荐给身边的博士后以及年轻生物学PI,多一点数据认知,让他们的科研上一个台阶:
- 全国巡讲全球听(买一得五)第3期(4月6日开始) ,你的生物信息学入门课。
- 数据挖掘线上班来袭(两天变三周,实力加量),医学生/医生首选技能提高课。
- 生信技能树的2019年终总结 ,你的生物信息学成长宝藏
- 2020学习主旋律,B站74小时免费教学视频为你领路