昨天我们生信技能树分享了 TCGA数据库免疫相关文件下载大全,里面给出来了2018年4月Immunity杂志上发表的文章The Immune Landscape of Cancer 整理好的49G的TCGA数据库文件,其中一个是甲基化信号值矩阵,单个文件就39G。
大量粉丝留言表示基本上在中国大陆是无法下载的,希望我们生信技能树好人做到底,帮忙下载整理好。
通常呢,这样的要求我是拒绝的, 比较绝大部分粉丝都是白嫖,一分钱不花,从来没有看到他打赏,但是提起要求来绝不含糊。就跟疫情期间那些耀武扬威的海外党,平时不交社保医保,这个时候想回国白嫖我们纳税人撑起来的医疗资源!我们不答应!
不过,生信技能树有一个宝藏男孩,唐医生,人如其名,偏向虎山行,用他的128G内存服务器,加上特殊手段,下载完了49G的TCGA数据库文件。而且还帮你把单个文件就39G甲基化信号值矩阵按照癌症拆分了,处理过程非常精彩。
下面是唐医生投稿的教程:
读取39G甲基化信号值矩阵按照癌症拆分
处理数据步骤 安装包
rm(list = ls())
options(stringsAsFactors = F)
###安装必须的包
if(!require('tidyverse')) install.packages('tidyverse')
if(!require('data.table')) install.packages('data.table')
读取甲基化矩阵数据
#######读取甲基化数据############# 另外也可以尝试使用vroom包中vroom函数读取
methy1<-data.table::fread('~/Desktop/jhu-usc.edu_PANCAN_HumanMethylation450.betaValue_whitelisted.tsv')
methy1[1:5,1:5]
这个时候不同读取代码速度不一样,但是39G甲基化信号值矩阵仍然是服务器级别的计算机才能hold住的哦。
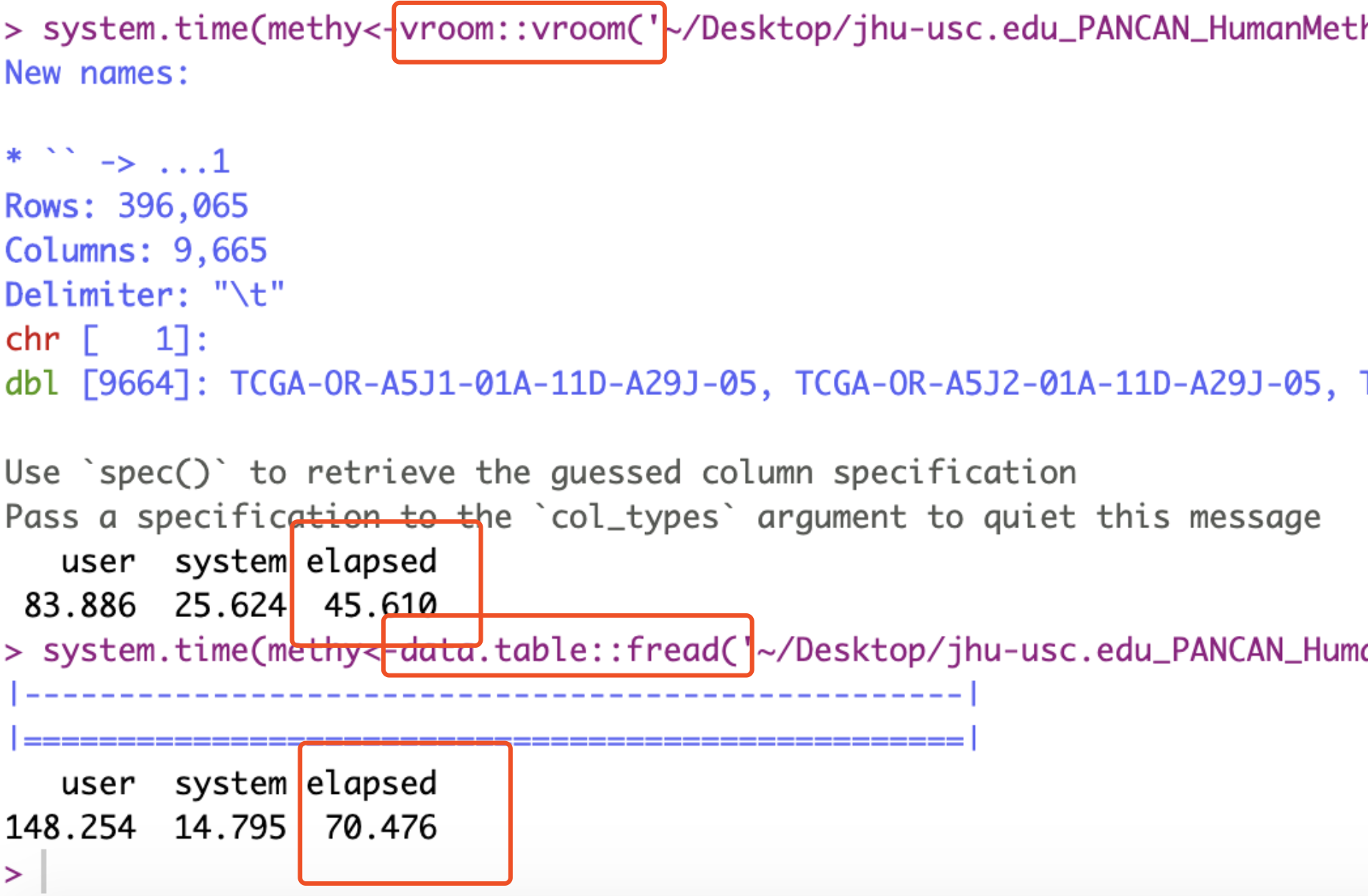
千万不要使用最基础的文件读取函数哦!
TCGA 官网下载全部甲基化样本sample-sheet信息
TCGA 官网摸索教程,我们这里不细说了,下载如下信息:
把从gdc 下载官网全部甲基化的sample sheet信息读入R进行处理。
sam<-read_tsv('/home/ftp/gdc_sample_sheet.2020-03-17.tsv')%>%
mutate(barcode=substr(`Sample ID`,1,12))%>%
dplyr::select(project=`Project ID`,barcode)
head(sam)
sample sheet信息文件如下:
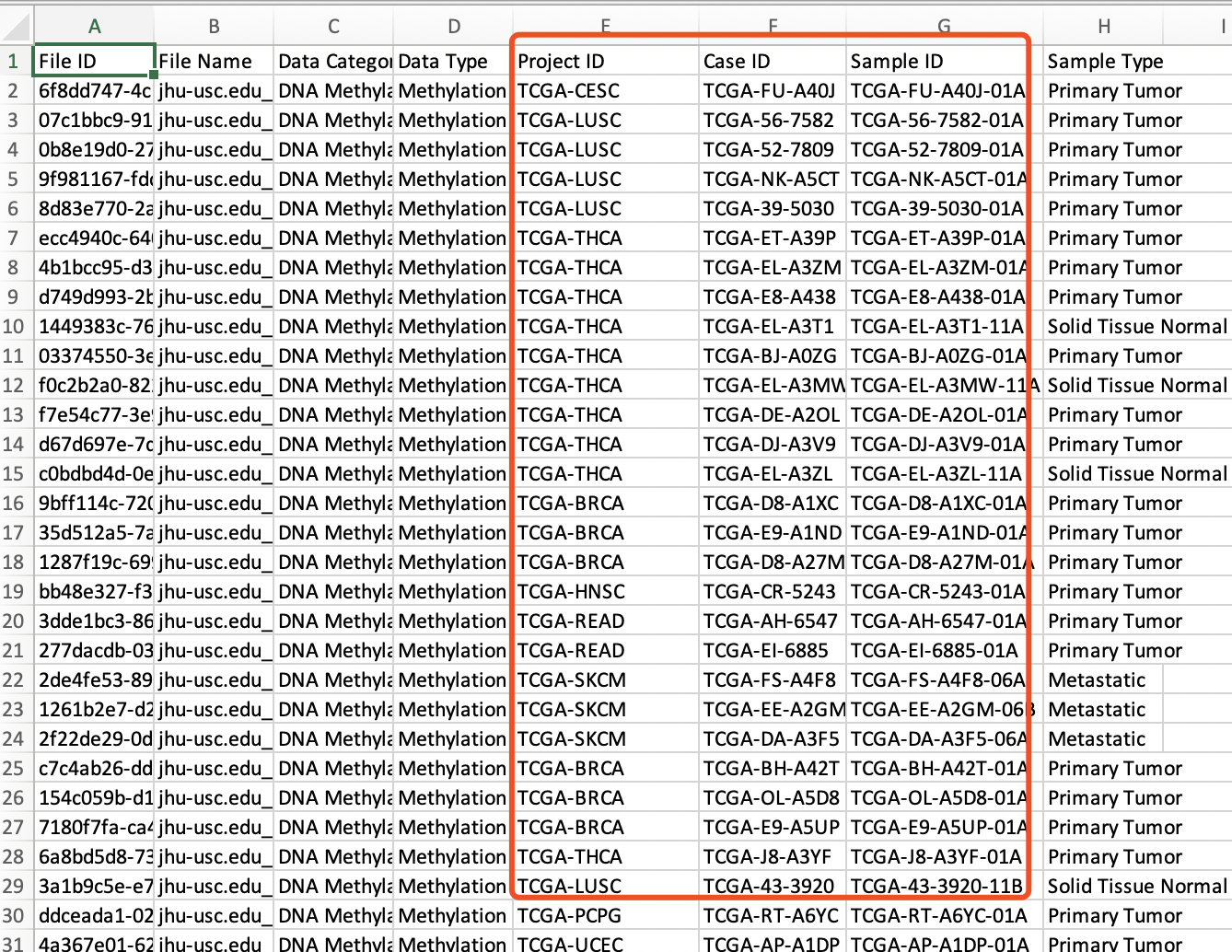
构建甲基化样本注释信息
samp<-data.frame(sample=colnames(methy1),barcode=substr(colnames(methy1),1,12))%>%
dplyr::filter(row_number()!=1)%>%
left_join(.,sam,by='barcode')
head(samp)
批量分割提取数据
splitdata<-function(i=1){
tcga_type<-unique(samp$project)
barcode<-dplyr::filter(samp,project==tcga_type[i])###提取单个肿瘤的barcode
methy450<-dplyr::select(methy1,V1,barcode$sample) ##按sample id 提取单个肿瘤
save(methy450,file = paste0(tcga_type[i],'_methy450.Rdata')) ###按肿瘤类型分别保存
}
lapply(1:33,splitdata) # 精彩
拿到的文件如下:
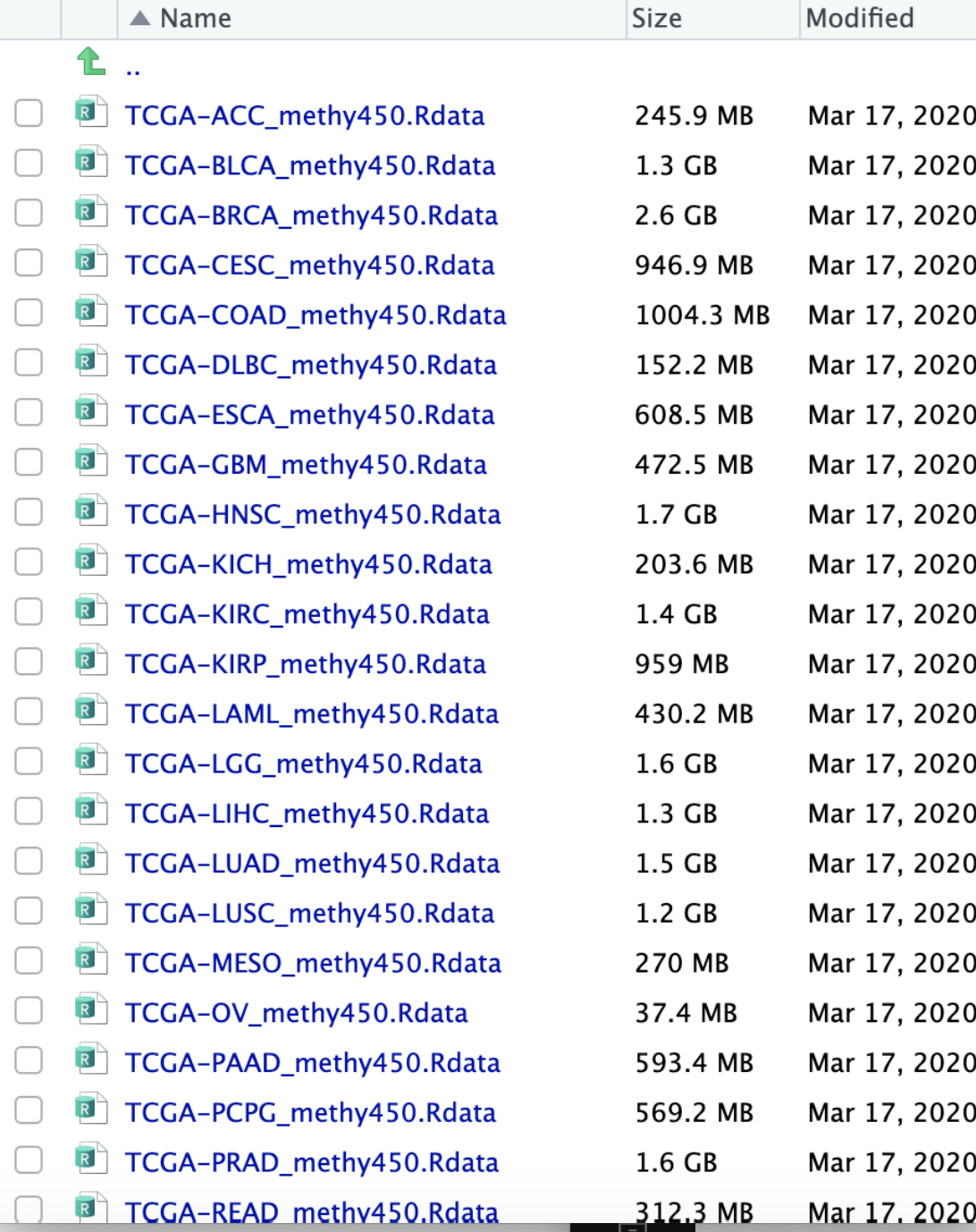
用同样的方法,也可以分割提取的pan cancer RNA-seq数据集!
这些文件都存储在百度云啦
如果你确实有需要,而唐医生也耗费了好几个小时做了这些工作,有必要稍微设置一下门槛!链接是:https://pan.baidu.com/s/1eI95uJC8tzDSbxdCkvrDog
但是密码需要付费6元才能查看哦!(文末付费)
文末友情宣传
强烈建议你推荐给身边的博士后以及年轻生物学PI,多一点数据认知,让他们的科研上一个台阶:
- 全国巡讲全球听(买一得五) ,你的生物信息学入门课
- 生信技能树的2019年终总结 ,你的生物信息学成长宝藏
- 2020学习主旋律,B站74小时免费教学视频为你领路
付费内容分割线
为有效杜绝黑粉跟我扯皮,设置一个付费分割线,这样它们就没办法复制粘贴我的代码,也不可能给我留言骂街了!
世界顿时清净很多!(下面就是百度云链接的提取码!付费查看,所获得的收益,完全归唐医生所有,慰藉他辛苦整理好几个小时的付出。)
密码烦请去微信公众号里面付费查看哦,链接是: