最近给学徒布置了一个作业,是一篇文章的数据图表重现,如下:
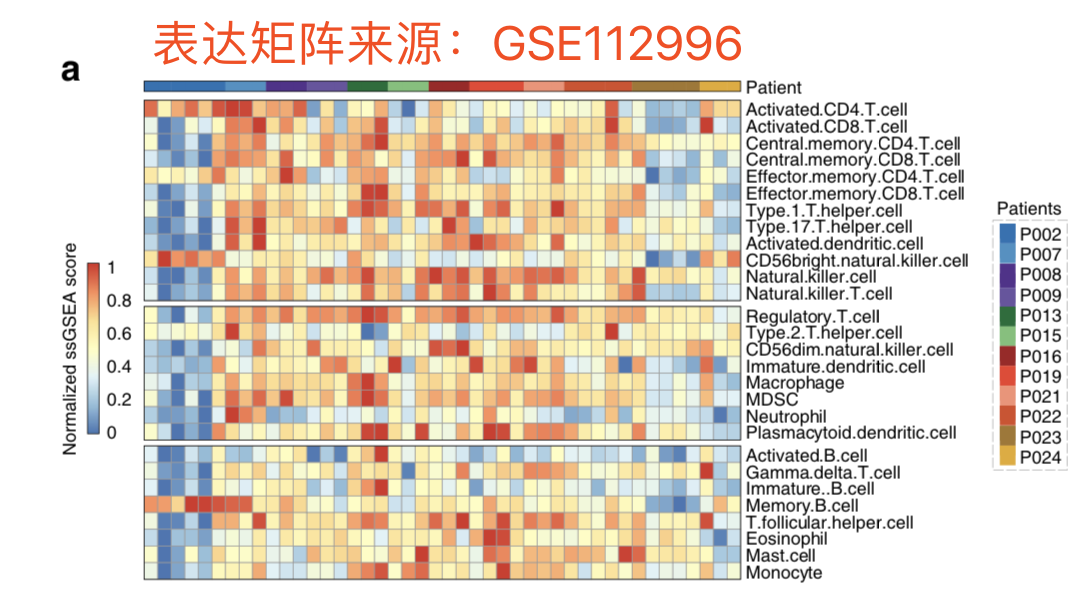
很简单,就是参考文献的28个免疫基因集拿出来,对从GEO下载的表达矩阵进行ssGSEA分析的结果热图呈现即可,比较难的应该是理解那28个免疫基因集,并且拿到每个基因集对应的基因列表,我本来以为是
- 2013的CELL文章:Spatiotemporal Dynamics of Intratumoral Immune Cells Reveal the Immune Landscape in Human Cancer
- 结果是2016的CELL reports文章:Pan-cancer Immunogenomic Analyses Reveal Genotype-Immunophenotype Relationships and Predictors of Response to Checkpoint Blockade
表现优异的学徒
但是拿到学徒提交的代码才眼前一亮,她居然是从上面文章的PDF附件里面,使用R语言的pdftools包进行自动化读取,并且格式化成为基因集列表进行后续ssGSEA分析,虽然代码很丑,但是实现了目的,PDF如下所示:

可以看到第 20到36页,是记录着基因集信息。
读取PDF并且提取信息的代码如下:
rm(list=ls())
library(pdftools)
options(stringsAsFactors = F)
b <- pdf_text('SupplementaryTables.pdf')
geneset_substract<- function(tmp){split_to_line<- gsub('\r','',strsplit(tmp,split = '\n')[[1]])
gene_name<- apply(data.frame(split_to_line),1,function(x){ line<- strsplit(x,split=' ')[[1]]
pos<- grep('[A-Za-z\\d+]|\\d+',line)
res <- line[pos[1]]})
cell_type<- apply(data.frame(split_to_line),1,function(x){ line<- strsplit(x,split=' ')[[1]]
pos<- grep('[A-Za-z\\d+]|\\d+',line)
res <- line[pos]
res <- res[c(-1,-length(res))]
s <- ''
for (i in 1:length(res)){
s<- paste(s,res[i])}
return(s)})
result<- data.frame(gene_name,cell_type)
return(result)
}
gene_set <- data.frame()
for(i in 20:36){
gene_set<- rbind(gene_set,geneset_substract(b[i]))
}
gene_set <- gene_set[c(-1,-2),]
list <- list()
for(i in 1:length(unique(gene_set$cell_type))){
list[[i]] <- gene_set$gene_name[gene_set$cell_type== (unique(gene_set$cell_type)[i])]
}
names(list)<- unique(gene_set$cell_type)
得到的基因集列表如下:

后续ssGSEA分析以及热图可视化,见生信菜鸟团的周一数据挖掘专场吧,这里留个悬念哈!
apply家族函数要活学活用
不过, 我还是觉得学徒代码太丑,修改了一下:
rm(list=ls())
library(pdftools)
options(stringsAsFactors = F)
b <- pdf_text('SupplementaryTables.pdf')
tmp = unlist(lapply(20:36, function(i){
trimws(strsplit(b[[i]],split = '\n')[[1]])
}))
tmp=tmp[-c(1,2)]
library(stringr)
tmp=do.call(rbind,lapply(str_split(tmp,' '), function(x){
x=x[-length(x)]
gene_name<- x[1]
cell_type<- trimws(paste(x[-1],collapse = ' '))
return(c(gene_name,cell_type))
}))
immune_list <- split(tmp[,1],tmp[,2])
后记
我相信这个技巧在很多场合都蛮有用的,不仅仅是生物信息学,如果你根据我们的教程学了后用到了可以发邮件跟我们交流哦。