最近一直在推送转录本差异相关的教程,见:每月一生信流程之rnaseqDTU(差异转录本) 扩充了大家对RNA-seq数据的理解,而且也指出来了,严格意义上的转录本定量其实是不容易的,对于二代测序来说:转录本定量本来就不是一件容易的事情 看留言,大家都深有同感!
不过好在有三代测序,ISO-seq领域的研究也不少,但是很多朋友对这个差异表达转录本的生物学认识不够,不清楚它的应用领域,恰好看到了一个组织特异性转录本的研究,分析给大家哈;
发表在 BMC Genomics的文章 October 2014, IUTA: a tool for effectively detecting differential isoform usage from RNA-Seq data 有一个示例图,非常的说明了转录本表达水平的差异:
When applied to actual mouse RNA-Seq datasets from six tissues, IUTA identified 2,073 significant genes with clear patterns of differential isoform usage between a pair of tissues.
IUTA is implemented as an R package and is available at http://www.niehs.nih.gov/research/resources/software/biostatistics/iuta/index.cfm.
在 IGV 可视化如下:
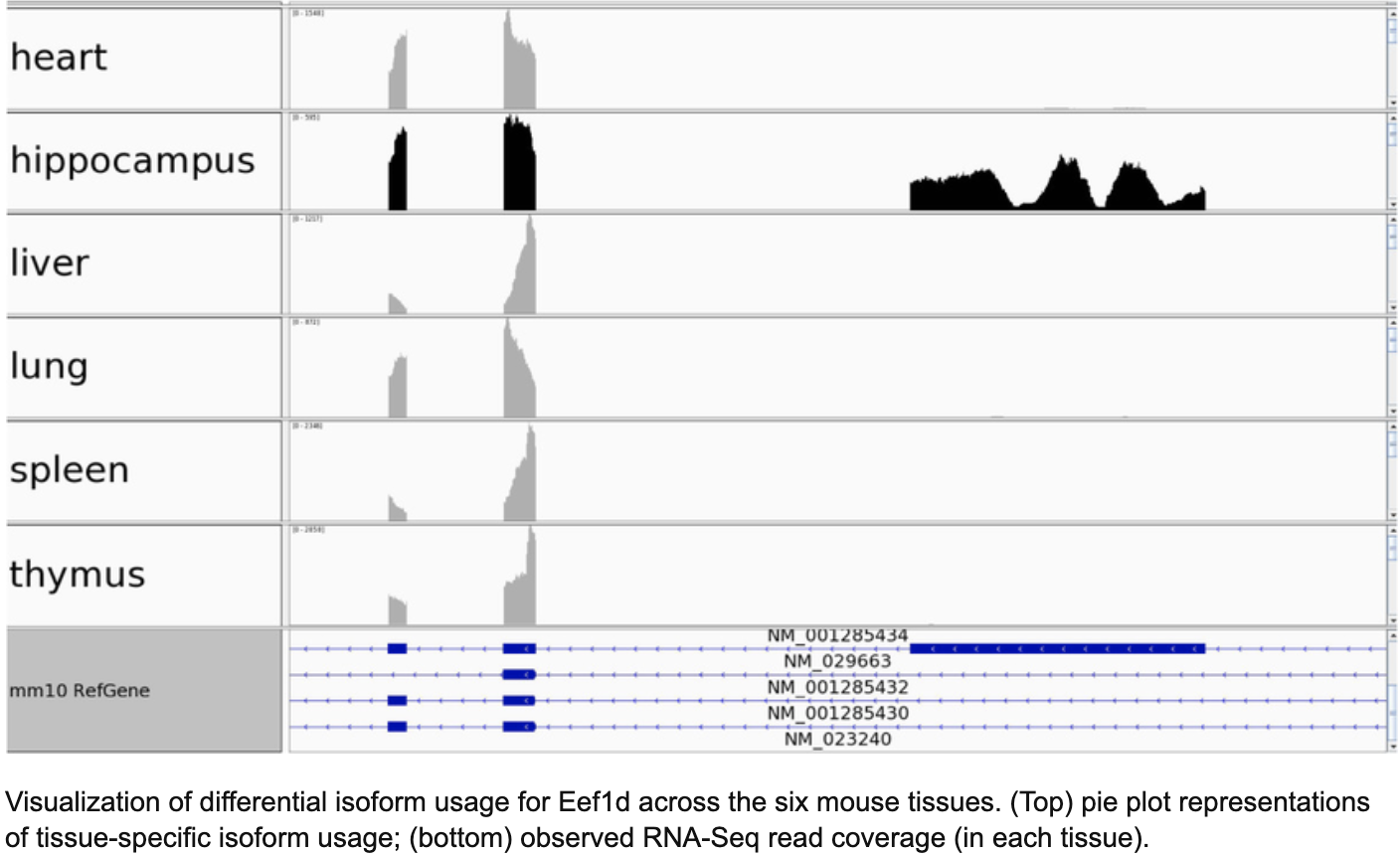
值得注意的是,该文章采用的是模拟的测试数据,来检验其软件对差异转录本的效果。这个示意图非常的理想化!
生信技能树可变剪切相关教程节选
因为做目录确实很浪费时间,差不多就下面这些,大家先学习吧: