前些天我在介绍GEO数据挖掘技术应用到RNA-seq数据分析的推文:GEO数据挖掘技术可以应用到表达芯片也可以是转录组测序 布置了一个作业:下载到GSE106292 数据集的 Excel表格如何读入R里面,做出作者文章的那样的图,可以参考关键问题答疑:
WGCNA的输入矩阵到底是什么格式,详细教程见:一文看懂WGCNA 分析(2019更新版)
本来以为是很简单,但是十万粉丝里面,我只收到了13份作业,可怜的13份答卷里面,还有5个是错的!其中大家错的最离谱的就是,搞不清楚文中的WGCNA针对的5个分组到底是什么!
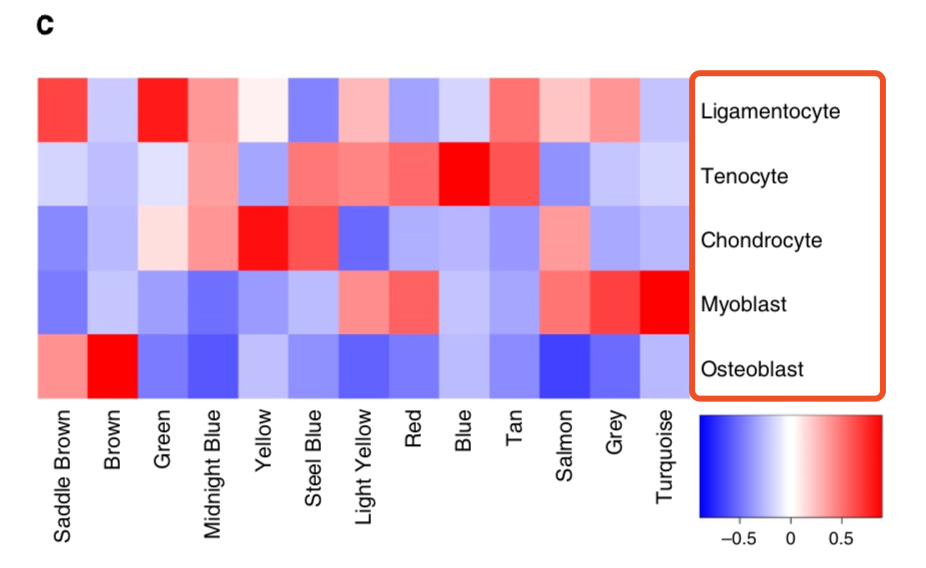
首先关注原文WGCNA图
下面的5个分组,都是英文专有名词,大家不理解其实是很正常的,没有人什么生物学背景都精通。
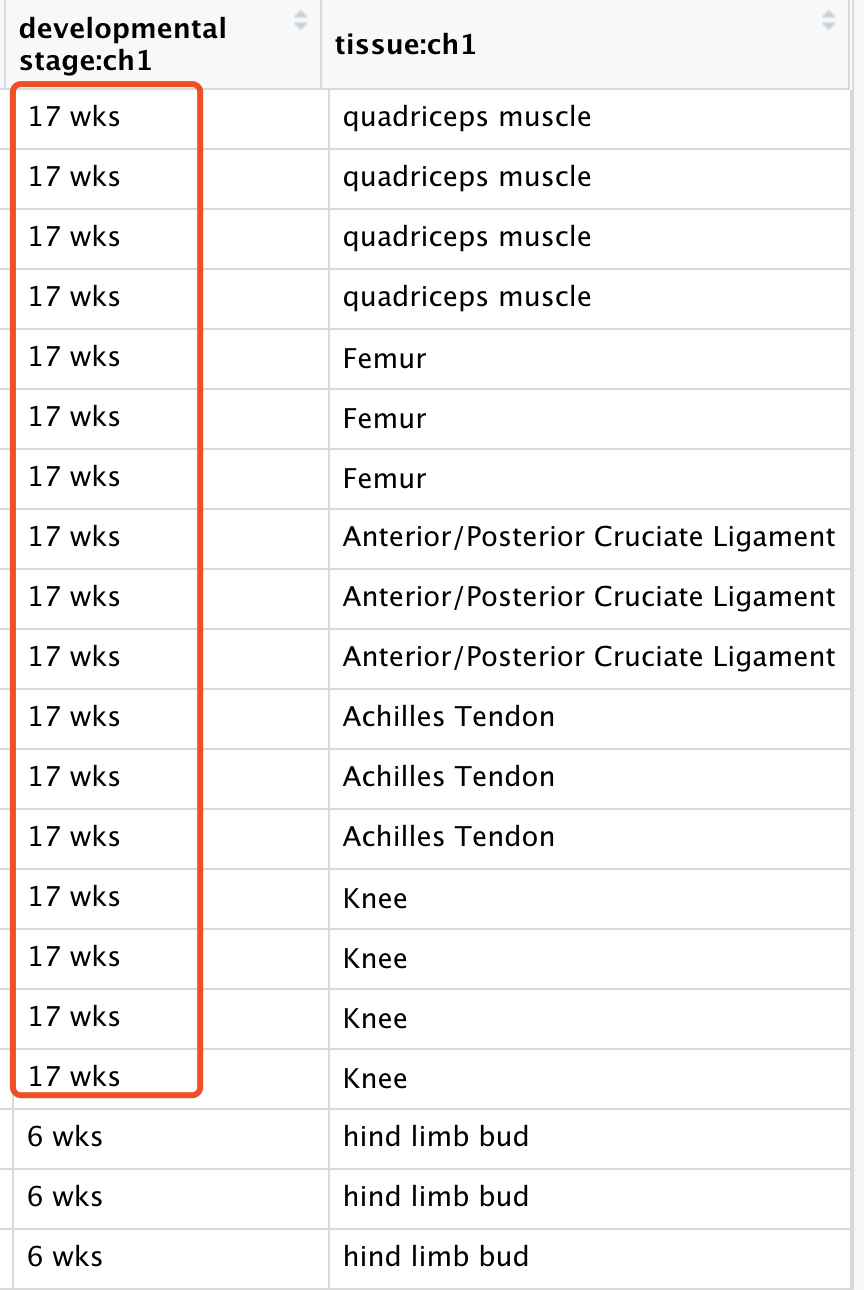
如果你下载到GSE106292 数据集的 Excel表格,就会发现,作者的分组其实很诡异!

有 bone, tendon, muscle, ligament 但是呢,很明显并不是原文的WGCNA里面的:hondrocytes, osteoblasts, myoblasts, tenocytes and ligamentocytes ,如果没有背景知识,就很难办!
文章里面,也是,这5个分组,并不是 hondrocytes, osteoblasts, myoblasts, tenocytes and ligamentocytes
需要细读文章
文章描述WGCNA的段落是:
Here we implemented RNA sequencing to generate cell type- specific transcriptomes for chondrocytes, osteoblasts, myoblasts, tenocytes and ligamentocytes at 17 weeks post-conception (WPC) of human development. We then employed Weighted Gene Co- expression Network Analysis (WGCNA) to define tissue-specific gene modules that represent each cell type.
也就是说,都是 17 weeks post-conception (WPC) ,那么我们的表达矩阵的样本名字里面,的确没有这个肿么办!
当然,就需要祭出我们的大杀器了,GEO数据挖掘流程:library(GEOquery) gset <- getGEO('GSE106292') pd=pData(gset[[1]])就这么简单,就拿到了文章所有的样本的表型信息啦!
感兴趣细节的可以自己去研读挖掘系列推文;
- 解读GEO数据存放规律及下载,一文就够
- 解读SRA数据库规律一文就够
- 从GEO数据库下载得到表达矩阵 一文就够
- GSEA分析一文就够(单机版+R语言版)
- 根据分组信息做差异分析- 这个一文不够的
- 差异分析得到的结果注释一文就够
但是样本信息仍然并不是WGCNA的分组
我们注意到,这个时候已经是5个组了:
的确并不是原文的:hondrocytes, osteoblasts, myoblasts, tenocytes and ligamentocytes ,
肿么办呢?
当然是继续看原文:
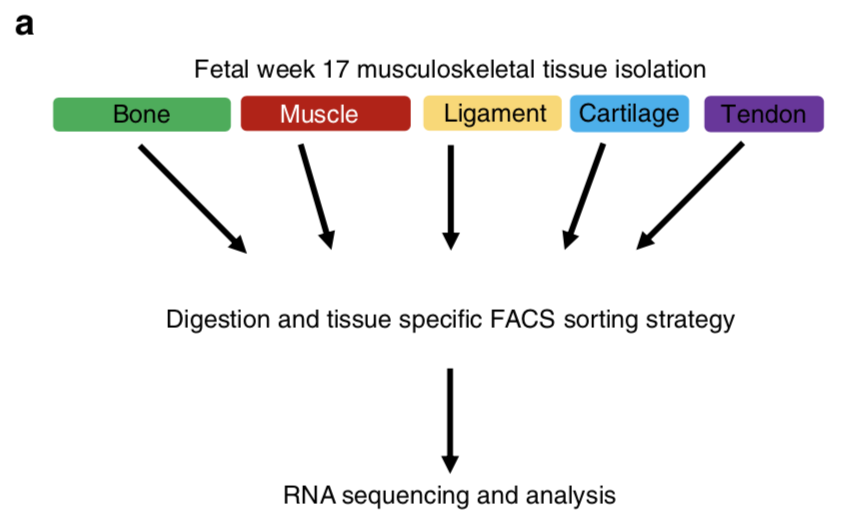
chondrocytes from the knee, myoblasts from the quadriceps, endosteal osteoblasts from the femur, and ligamentocytes and tenocytes from the anterior and posterior cruciate ligament and Achilles tendon, respectively
这样就把样本信息和WGCNA图表信息啦!如果没有生物学背景,必然分析起来很吃力
但是呢,你们有没有注意到,其实我也不知道那些单词背后的生物学背景,但是我仍然是可以找对!
好了,十万粉丝做题活动继续,这两个图难度非常大,基本上相当于半年左右的生信工程师经验了,如果你能做出来,发邮件给我你的全部思考分析过程,你可以获得我认可,毕竟相当于有了我7.6%的功力,已经是非常的了不起了!
但是呢, 因为我点出来了第一个困难,所以,你们的功力相当于大打折扣,不过还是值得我表扬,加油!