相信遇到这样的问题的朋友不在少数,在中国大陆做数据分析,下载软件数据文件遇到困难那是家常便饭。
比如安装GitHub的R包,因为并不是所有的R包都会被正式的发布在CRAN或者bioconductor,所以对于简简单单分享在GitHub的R包一般我们搜索到如下代码:
library(devtools)
# https://github.com/jasdumas/shinyGEO
install_github("jasdumas/shinyGEO")
if(!requireNamespace("ThreeDRNAseq", quietly = TRUE))
devtools::install_github('wyguo/ThreeDRNAseq')
一切看起来那么美好
函数下载困难就浏览器下载
实际上,在很多地方,上面这样的代码,都只能是永无休止的等待。
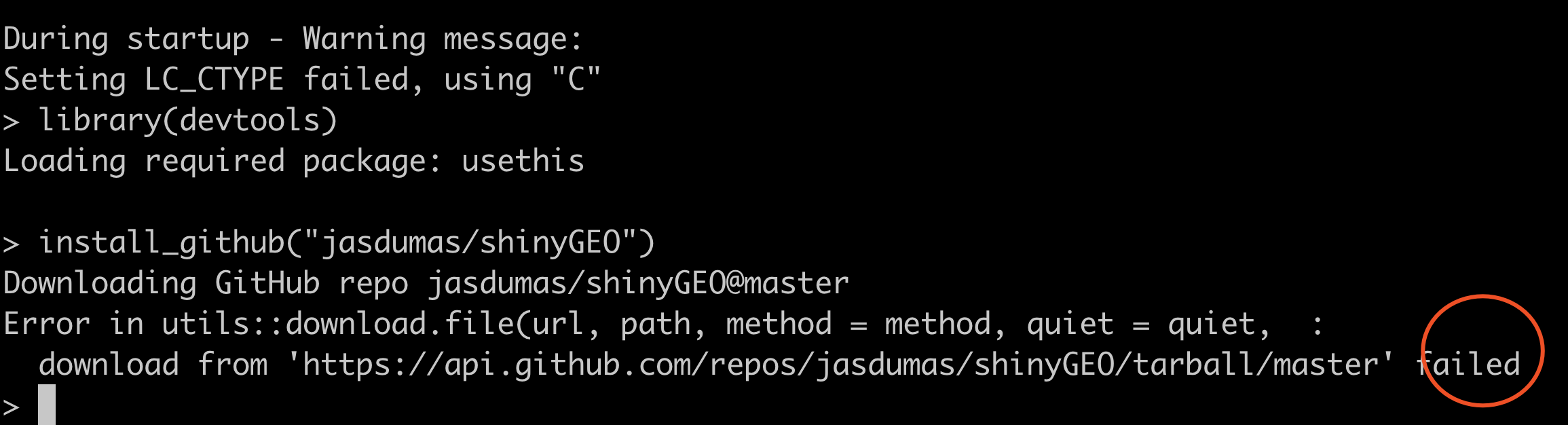
其实还可以先下载GitHub的这个R包的项目文件:
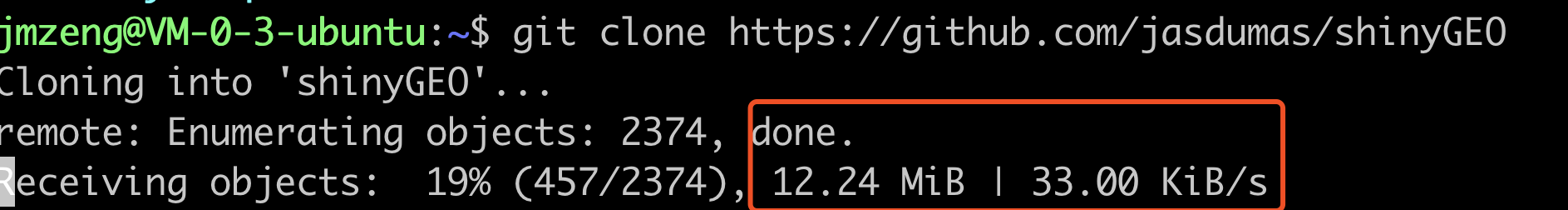
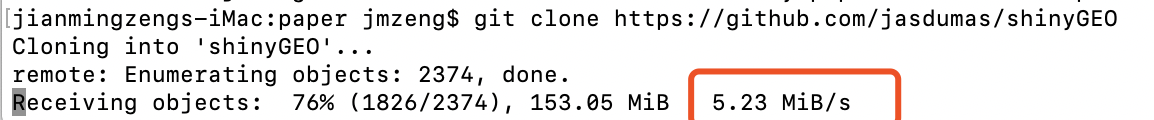
当然了,你可能需要找一个网速稍微好一点的下载渠道:
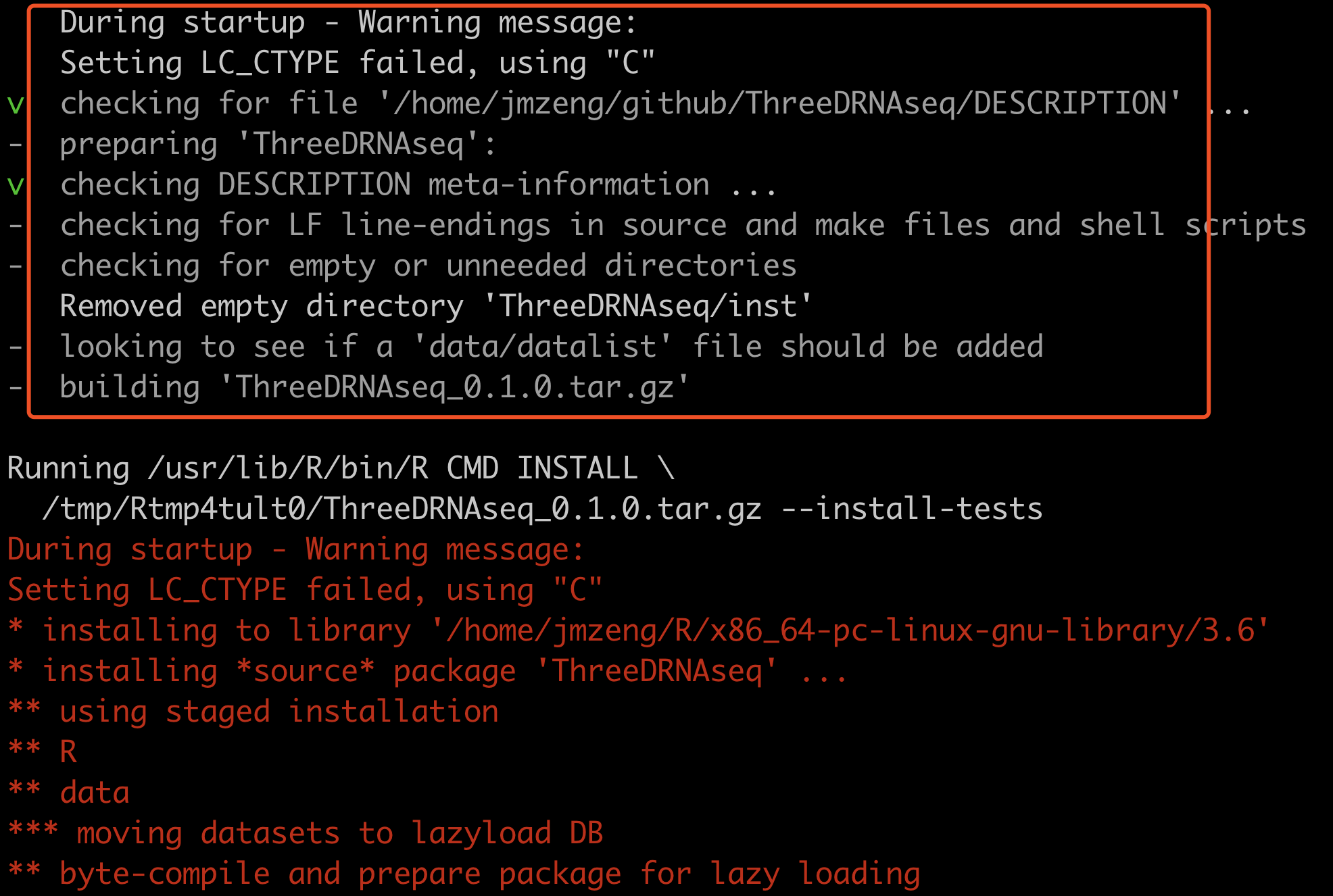
然后再本地安装,这个时候install_github函数可能是有问题的啦,所以需要谷歌搜索,考验大家搜索能力的时候到了,主要是关键词寻找:install github local r package 一般来说,程序员之友论坛就会是答案:https://stackoverflow.com/questions/17366772/install-r-packages-from-github-downloading-master-zip
devtools::install('github/ThreeDRNAseq/')
一般来说,就会成功:
另外一个小技巧
实际上,非常多的时候,我们只是记住了R包的名字,而不知道作者,所以下载的时候给出作者+R包名字有点困难,一个 githubinstall 可以根据R包名字帮你联想,just like install.packages().
library(githubinstall)
githubinstall("AnomalyDetection")
Suggestion:
- twitter/AnomalyDetection Anomaly Detection with R
Do you want to install the package (Y/n)?
githubinstall() suggests GitHub repositories from input package names and asks whether you install it.
当然了,我觉得没啥意思,我通常是连R包都不记得名字,就是复制粘贴嘛!
不过,这个包自己当然是不仅仅这么一点功能啦,感兴趣的可以去看看:https://cran.r-project.org/web/packages/githubinstall/vignettes/githubinstall.html