做生命科学研究,尤其是医学相关课题的,很少能脱离开基因工程小鼠,也就是genetically engeenierd mouse modesl (GEMMs)
最近发现一个很有趣的现象,比如发表于 Science 05 Oct 2018 , 文章是:Reprogramming normal human epithelial tissues to a common, lethal neuroendocrine cancer lineage 的研究人员通过对基因工程小鼠,发现只要改变 5 个基因(这 5 个基因统称为 PARCB )的活性,就能让上皮健康细胞发生癌变,并发展成为小细胞癌。
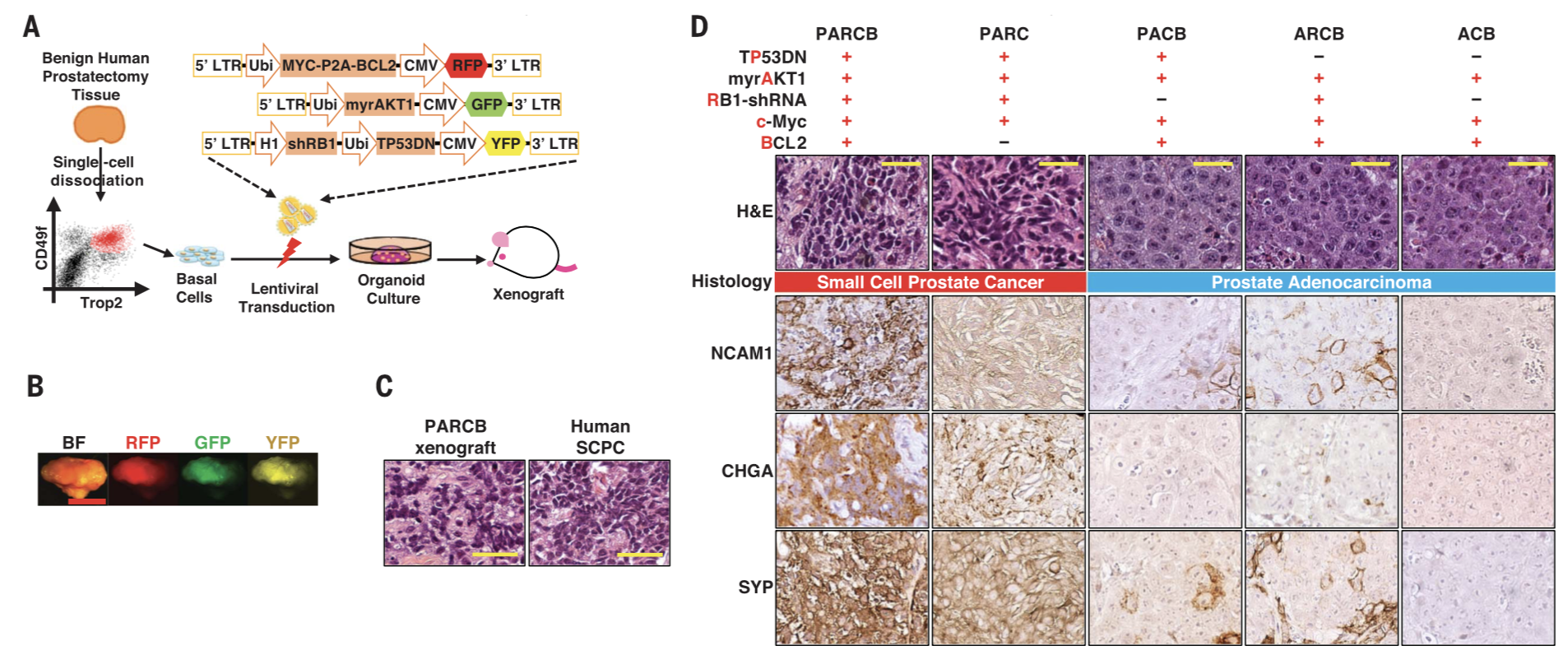
看起来似乎是对小鼠,想怎么敲基因就可以怎么敲,非常的自由,多个基因任何组合随心所欲的敲,是这样吗?
比如我又看到,发表在 Cancer Cell. 2019 Sep 16 的文章:Epithelial NOTCH Signaling Rewires the Tumor Microenvironment of Colorectal Cancer to Drive Poor-Prognosis Subtypes and Metastasis. 也是使用基因工程小鼠。
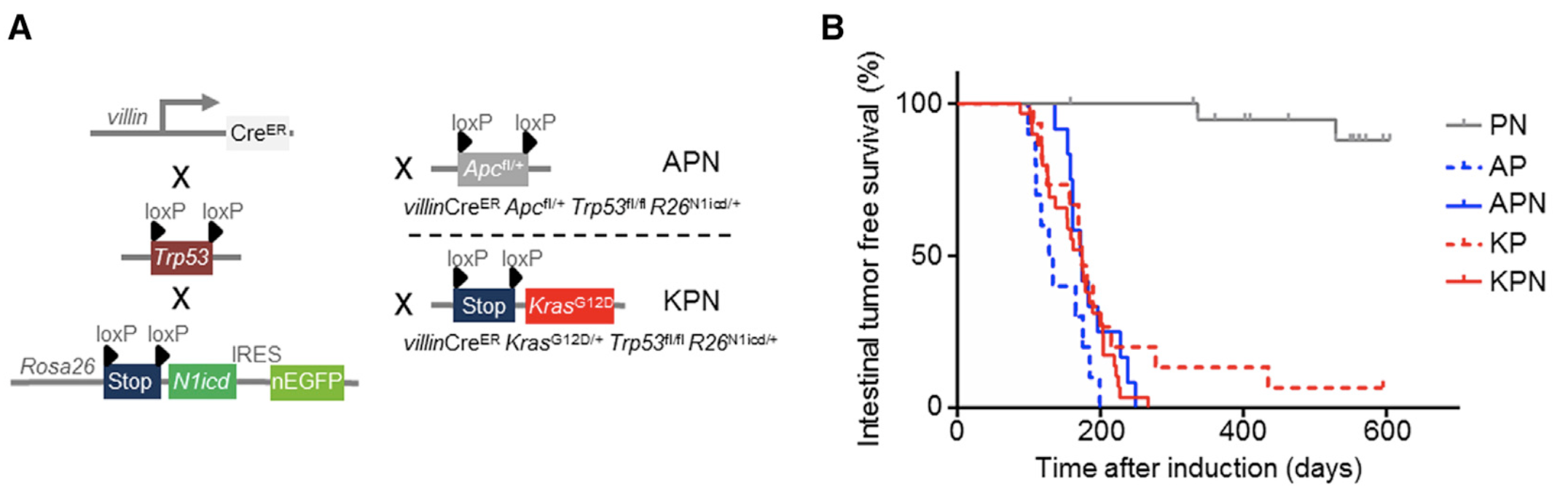
不同的基因干扰组合,就产生了不同的基因型小鼠,分析的时候就可以多元化了。
那么问题来了,是不是可以在基因工程小鼠上面为所欲为干扰任何基因呢?我想从一个生信工程师的角度听一下你的故事,希望你丰富的实验背景能给我带来一些启发,发到我邮箱 jmzeng1314@163.com
当然,作为交换,我会给你价值500元的添加我微信的资格哈!
也会赠送你全网最系统的表达芯片数据处理教程全部视频和代码,反正版权在我手上。
表达芯片数据处理教程,早在2016年我就系统性整理了发布在生信菜鸟团博客:http://www.bio-info-trainee.com/2087.html
配套教学视频在B站:https://www.bilibili.com/video/av26731585/
代码都在:https://github.com/jmzeng1314/GEO 早期目录如下:
- 第一讲:GEO,表达芯片与R
- 第二讲:从GEO下载数据得到表达量矩阵
- 第三讲:对表达量矩阵用GSEA软件做分析
- 第四讲:根据分组信息做差异分析
- 第五讲:对差异基因结果做GO/KEGG超几何分布检验富集分析
- 第六讲:指定基因分组boxplot指定基因list画热图
- 第七讲:根据差异基因list获取string数据库的PPI网络数据
- 第八讲:PPI网络数据用R或者cytoscape画网络图
- 第九讲:网络图的子网络获取
- 第十讲:hug genes如何找
公众号推文在:
- 解读GEO数据存放规律及下载,一文就够
- 解读SRA数据库规律一文就够
- 从GEO数据库下载得到表达矩阵 一文就够
- GSEA分析一文就够(单机版+R语言版)
- 根据分组信息做差异分析- 这个一文不够的
- 差异分析得到的结果注释一文就够