为了分析不同类型、组织起源肿瘤的共性、差异以及新课题。TCGA于2012年10月26日-27日在圣克鲁兹,加州举行的会议中发起了泛癌计划。参考:https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6000284/ 为此我也录制了系列视频教程在:TCGA知识图谱视频教程(B站和YouTube直达)
本研究发表于普通杂志:Sci Rep. 2013 Oct 题目是:Comprehensive identification of mutational cancer driver genes across 12 tumor types. 主要就是使用4个软件分析TCGA数据库的somatic突变结果而已。本文献解读属于100篇泛癌研究文献系列,首发于:http://www.bio-info-trainee.com/4132.html
虽然本文比较简单,但他们使用的数据来源的文章很厉害,是 Nature 502, 333–339 (17 October 2013) doi:10.1038/nature12634 因为是二次分析,所以创新性就不足。
关于驱动基因
癌症发生的根本原因是基因突变。从表现形式上来讲是肿瘤细胞不可控制的增长,主要是肿瘤驱动基因突变导致,这些基因发生突变后,特别是某些基因发生重大突变,肿瘤的发生就不再仅仅是一种风险,而是一种必然。
关于鉴定驱动基因,看我们生信技能树团队成员的一个文献总结:https://life2cloud.com/cn/2018/01/jcb-review-smg/
找driver基因的4个算法
每个算法来判断driver基因,都有自己的优缺点,如下:
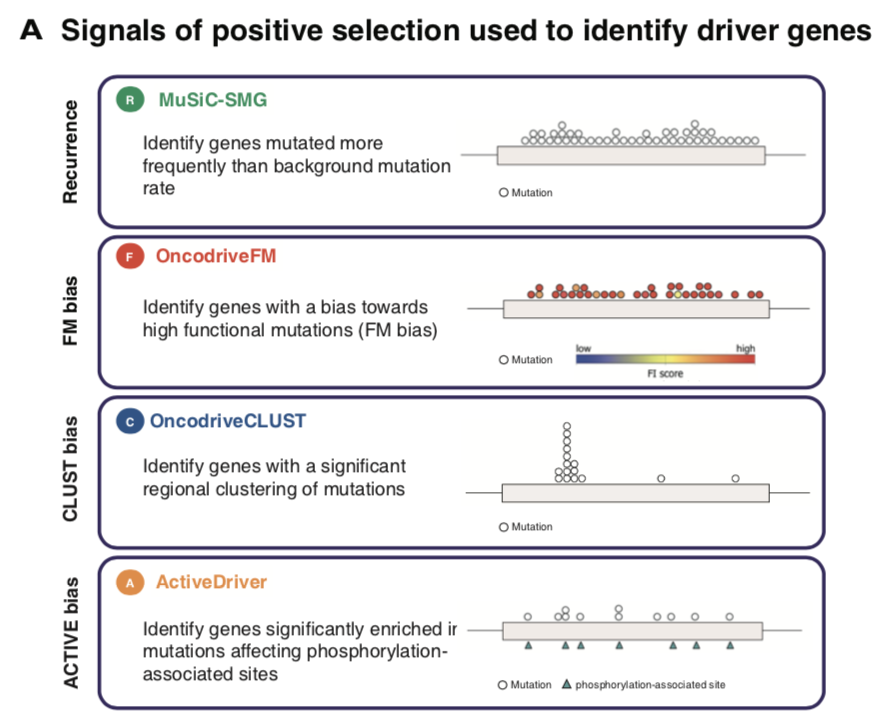
所以作者把这4个算法 (MuSiC, Onco-driveFM, OncodriveCLUST and ActiveDriver) 应用到了TCGA计划的12个癌症的三千多病人数据。
算法的一致性
韦恩图是比较好的展现方式
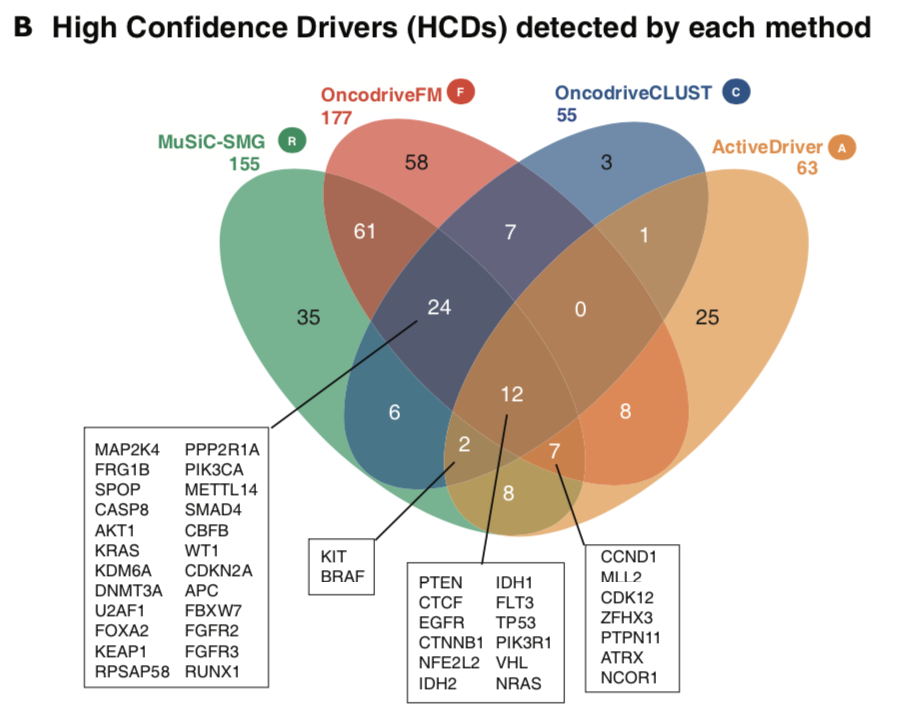
很多重要的的基因都是不同算法被判定为driver基因,比如:
- RB1 possesses both clear recurrence and FM bias, it has undetectable CLUST or ACTIVE biases.
- Mutations in HRAS are both significantly clustered and biased towards high functional impact, but are neither signifi-cantly recurrent nor ACTIVE biased.
- BRAF, on the other hand shows all signals of positive selection, except FM bias.
和CGC比较
COSMIC数据库维护的CGC基因集,Cancer Gene Census - COSMIC
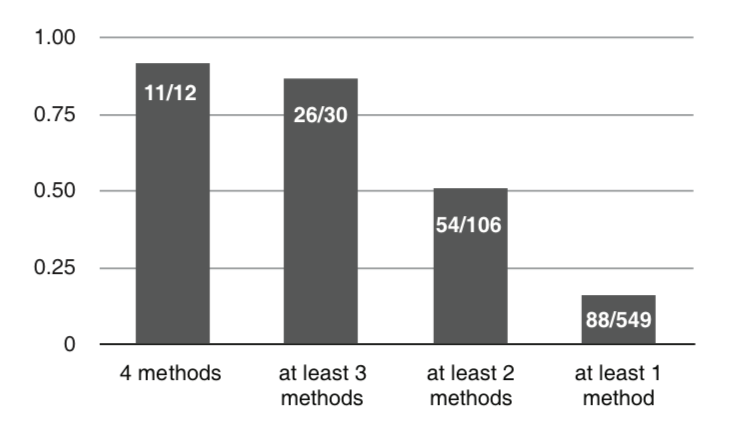
可以看到,当年的549个CGC基因,有88个都是至少被一个算法可以找到。关于HCDs
全文的重点:
We designed a novel approach to elaborate the catalog of high- confidence drivers, HCDs, across the pan-cancer dataset combining the results of multiple methods to identify cancer driver genes.
后记
理论上是可以下载数据重现这个分析,前提是学习这4款软件的用法:https://www.synapse.org/#!Synapse:syn1729383
- Download the MAFs from syn1710680
- Filter out variants that are unlikely to be somatic
2.1. Variants that are common in NHLBI exomes or 1000G (Global MAF > 0.1%)
2.2. Variants in genes without Hugo names (Prefixed with LOC or ENSG)
2.3. Variants in genes known to be problematic on GRCh37 (PDE4DIP, CDC27, MUC4, DUX4, HYDIN, PRIM2)
2.4. Variants annotated to transcripts without a start or stop codon, unless it’s in a pseudogene
2.5. Variants in a blacklist of common artifacts seen during manual review at The Genome Institute, WashU - Classify variants in pseudogenes as Silent (in MAF column 9, Variant_Classification)
这个数据来源于 很牛的文章,Nature 502, 333–339 (17 October 2013) doi:10.1038/nature12634 值得大家仔细学习!
当然了,如果你想超脱于他们的泛癌计划已经发表的研究,那么就非常有必要跟着我读完这100篇泛癌文献!
详见我的100篇泛癌研究文献解读目录:http://www.bio-info-trainee.com/4132.html