文章亮点
文章 Published: 30 July 2018 :Organoid cultures recapitulate esophageal adenocarcinoma heterogeneity providing a model for clonality studies and precision therapeutics
作者成功培养了EAC(食道癌的一种)的organoid ,证明了organoid可以保留肿瘤患者的原位癌
- morphology, genomic, and transcriptomic landscape
- point mutations, copy number alterations, and mutational signatures
提供了非常有意义的a pre-clinical tool食道癌背景知识
食道癌,跟其它癌症一样,区分为腺癌 (adenocarcinoma)/ 鳞癌 (squamous cell carcinoma)或其他形态,也就是ESCC和EAC
- esophageal squamous cell carcinoma (ESCC)
- esophageal adenocarcinoma (EAC)
EAC西方人为主
五年生存率低于15%前期工作
NGS组学技术已经广泛应用于该癌症,包括:
- Alexandrov, L. B. et al. Signatures of mutational processes in human cancer. Nature 500, 415–421 (2013).
- Weaver, J. M. J. et al. Ordering of mutations in preinvasive disease stages of esophageal carcinogenesis. Nat. Genet. 46, 837–843 (2014).
- Dulak, A. M. et al. Exome and whole-genome sequencing of esophageal adenocarcinoma identifies recurrent driver events and mutational complexity. Nat. Genet. 45, 478–U437 (2013).
- Nones, K. et al. Genomic catastrophes frequently arise in esophageal adenocarcinoma and drive tumorigenesis. Nat. Commun. 5, 5224 (2014).
- Cancer Genome Atlas Research, N. et al. Integrated genomic characterization of oesophageal carcinoma. Nature 541, 169–175 (2017).
- Secrier, M. & Li, X. et al. Mutational signatures in esophageal adenocarcinoma define etiologically distinct subgroups with therapeutic relevance. Nat. Genet. 42, 1131–1141 (2016).
其中2016的. Nat. Genet. 文章是作者做的,使用全基因组测序手段量化了接近100个EAC患者的基因组特性。
而且说明了EAC的细胞系表现不好,急需其它模型,比如最近大放异彩的类器官。
所以作者开始这个研究计划:类器官的培养
成功率尚可,达到 31% (10 of 32 samples), 跟其它恶性癌症相当。
那些培养失败的原因包括: - lack of growth from culture initiation (n = 11 cultures),
- infection (n = 5)
- fibroblast overgrowth (n = 4)
- arrested growth (n = 2)
摸索这个培养条件并非易事。
培养类器官成功的病人信息如下:

WGS和RNA-seq测序
数据都在European Genome-phenome Archive (EGA) under accession EGAD00001004007. 需要申请才能下载。
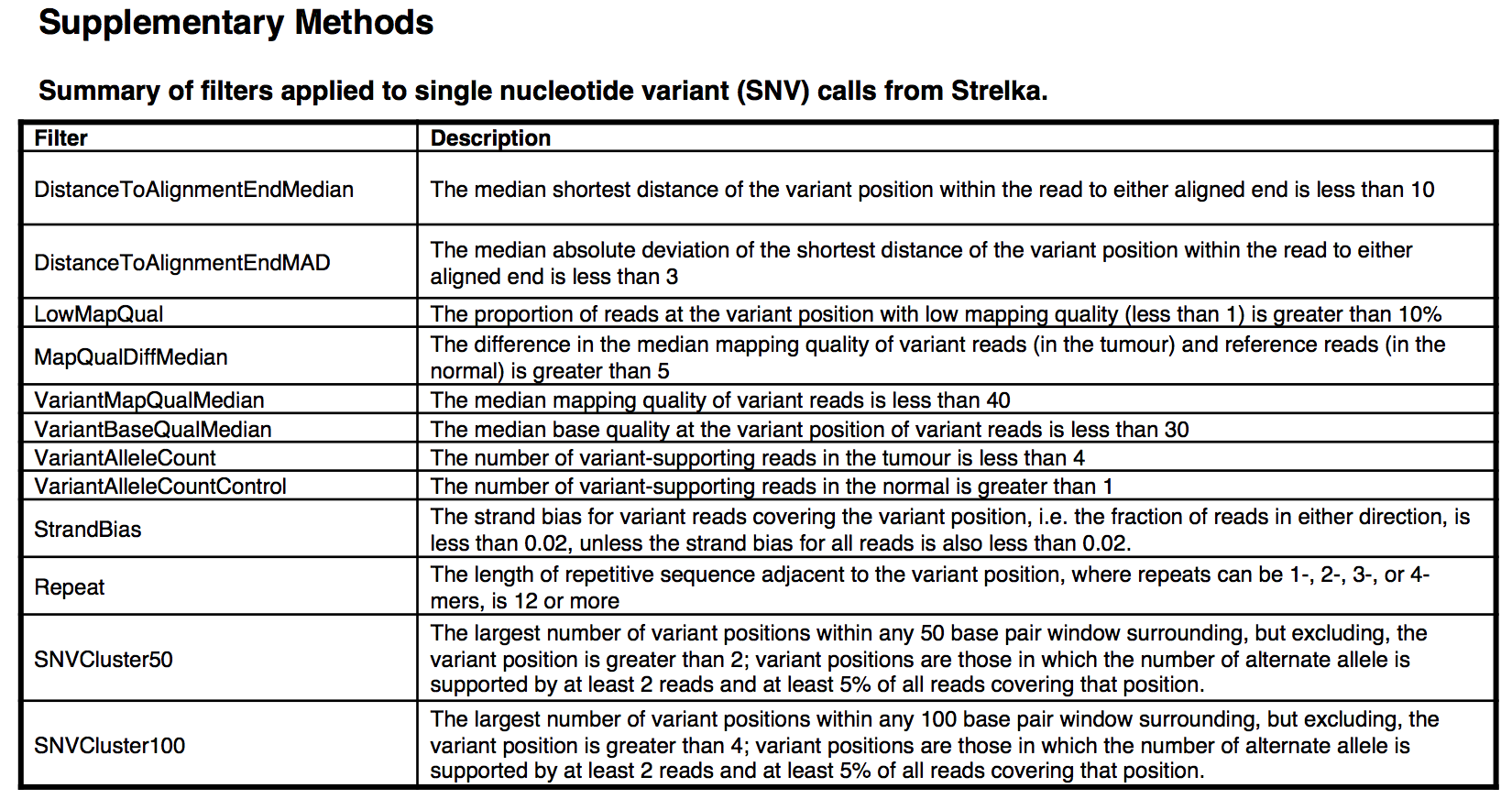
数据分析比较常规,也就是一些软件的流程连接,值得注意的是作者并没有选择GATK的mutect2来找somatic的SNVs,而是采用了strelka,参数如下:
基于RNA-seq测序转录组测序主要看表达量,作者同时也探索了RNA-seq的变异位点。类器官能重现其原位癌的突变
所有的类器官文章都必须说明这一点,当然要想尽一切办法来提高同一个病人的原位癌及其对应的类器官测序得到的somatic突变的一致性。
因为作者在2016年了做了n = 129的癌症队列,其somatic突变结果用MutSigCV找到了可能的driver somatic events,这样能比较的基因就非常少,这样能提供突变的一致性。
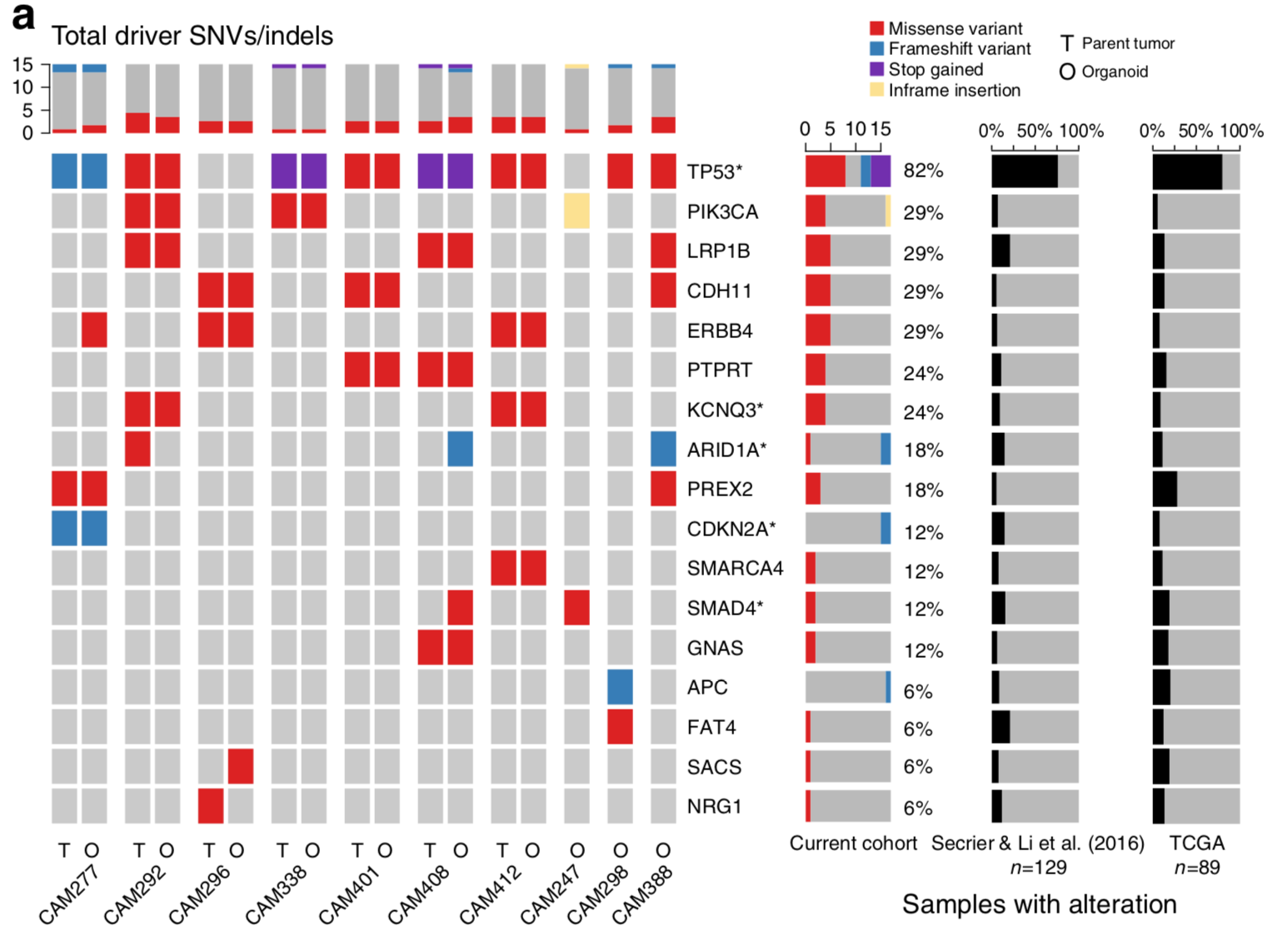
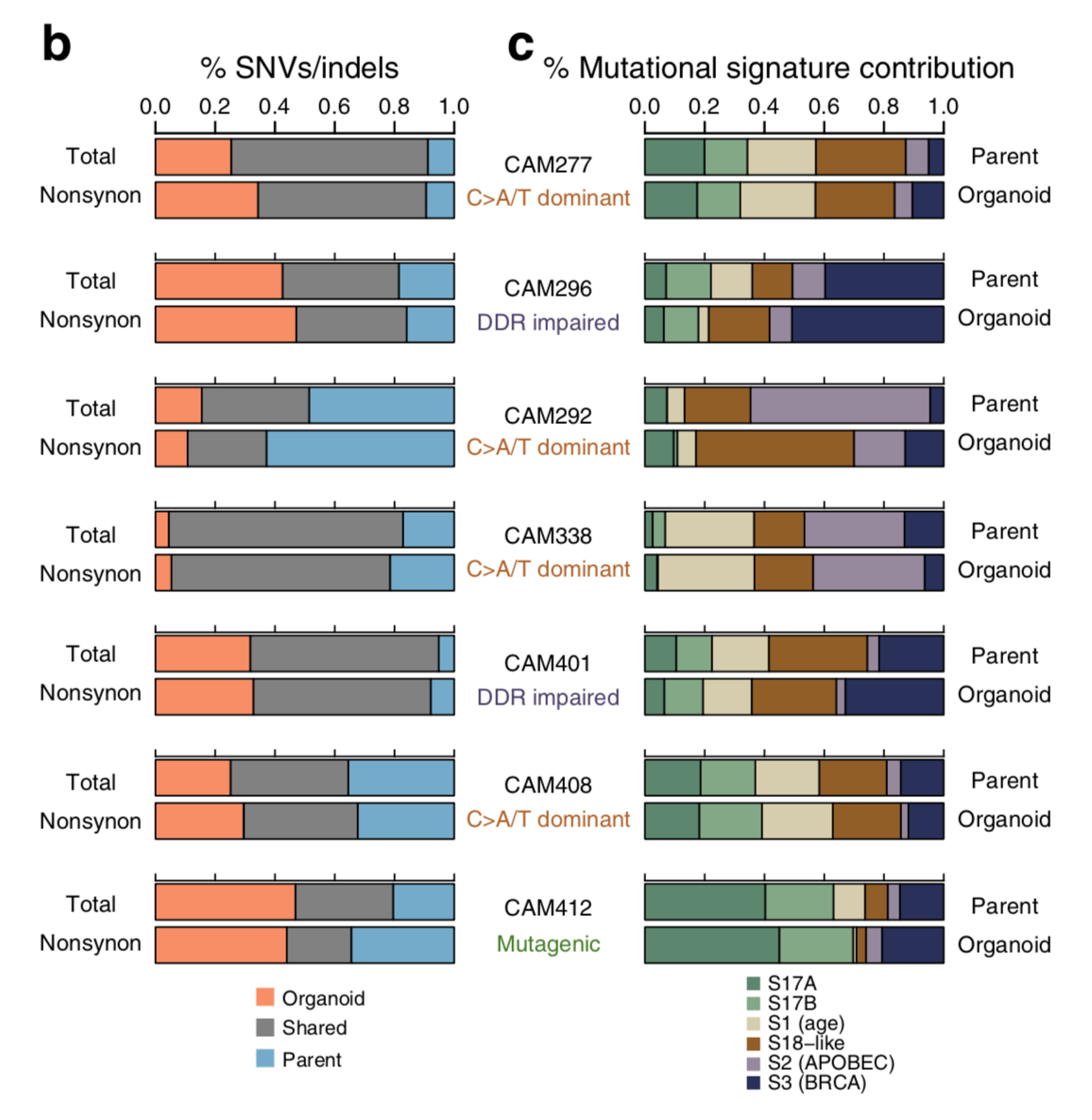
不过这里作者并不是取巧,因为他们仍然是比较了全部的突变,从SNV,CNV,SV的角度分别比较,都得到了非常好的一致性。
首先看全部的SNVs的一致性,如下:
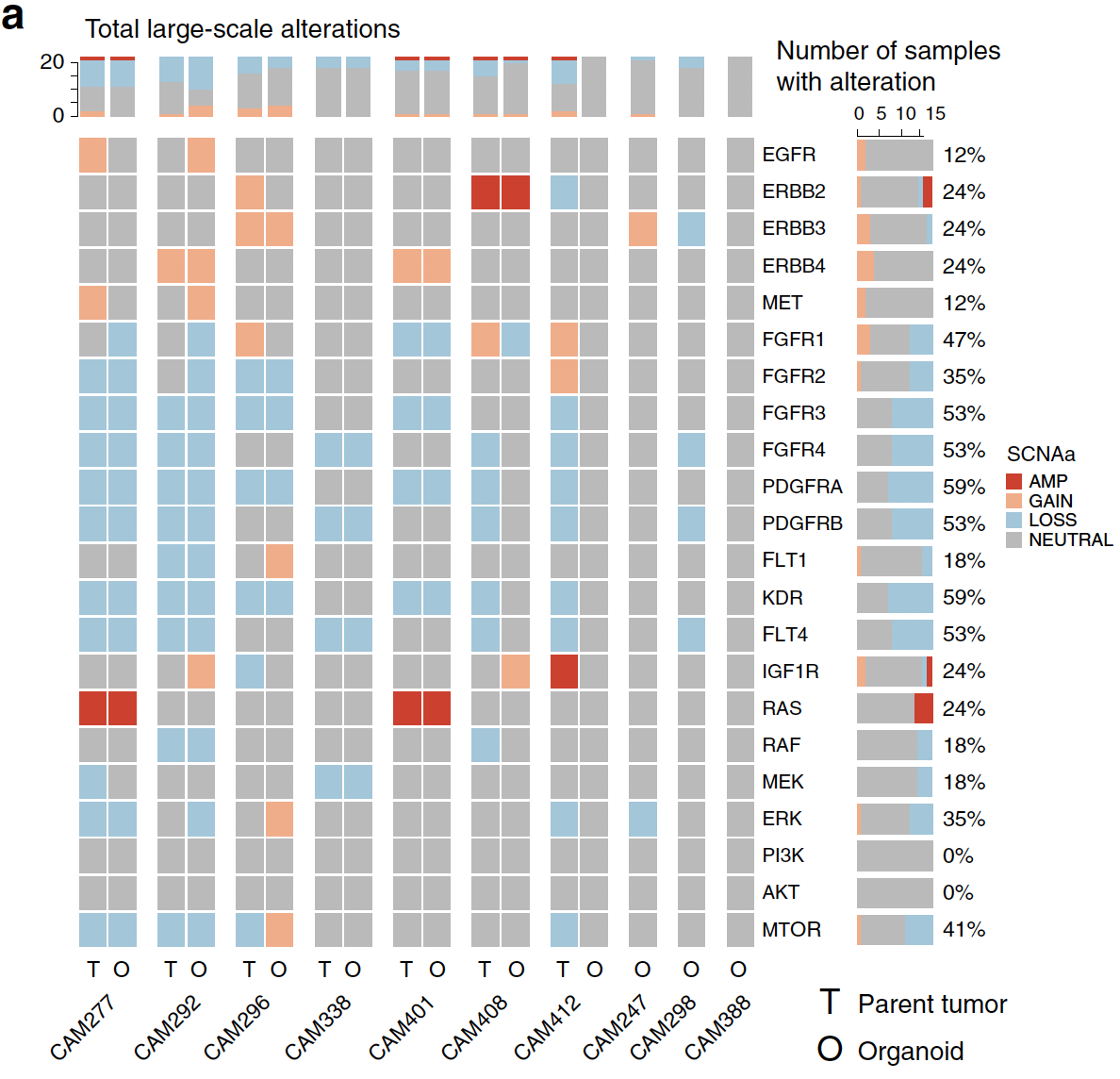
然后看全部的CNV的一致性,如下:
最后看全部的SV的一致性,如下:
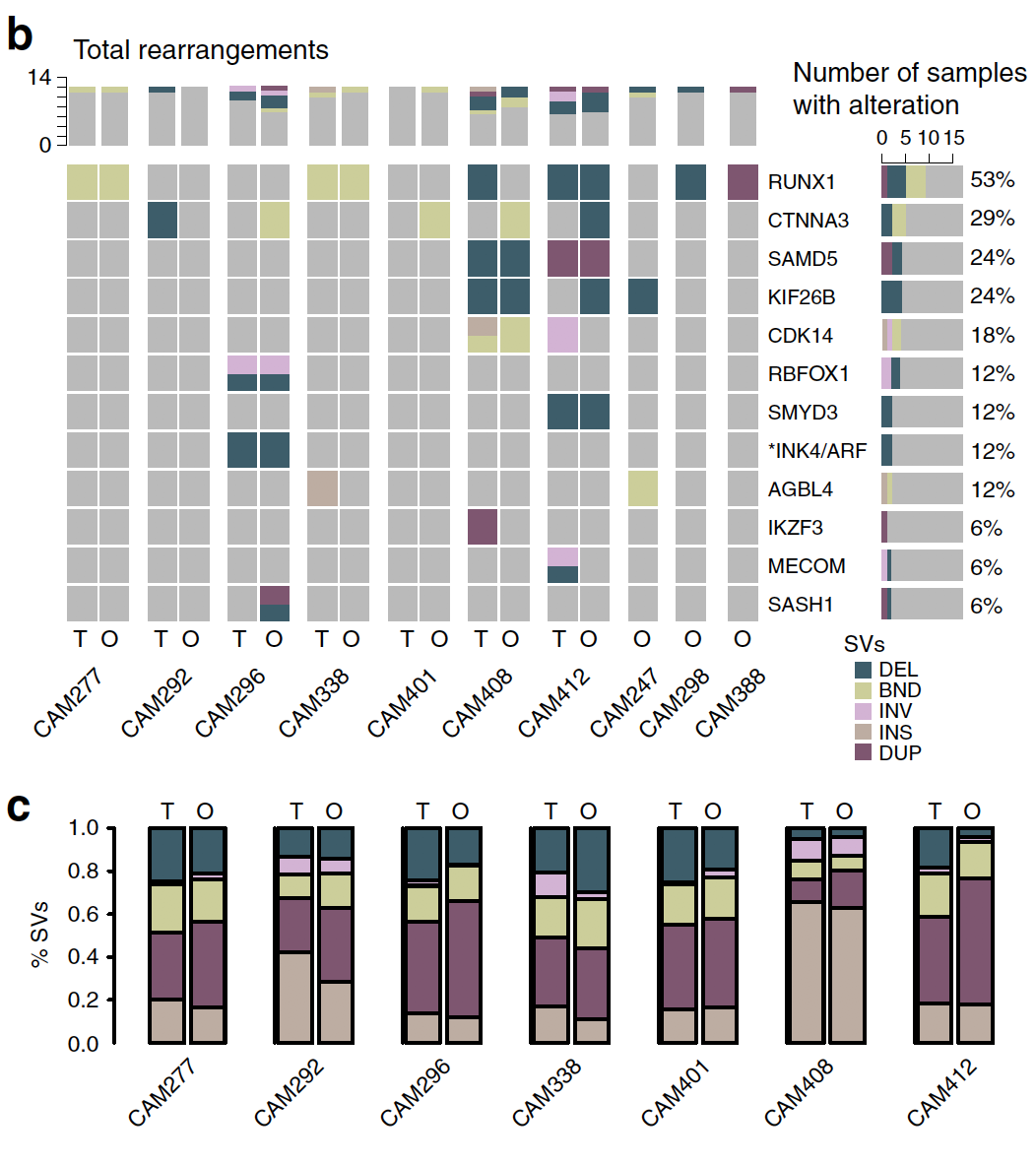
总而言之,organoids比较完美的保留其来源病人的原位癌的分子特性。
还验证了基于RNA-seq表达量及变异的一致性,也非常好!!!肿瘤亚克隆
使用Battenberg 算法对原位癌和类器官都计算copy number aberrations, tumor ploidy, and purity ,而且只探索four tumors (CAM277, CAM296, CAM338, and CAM401).
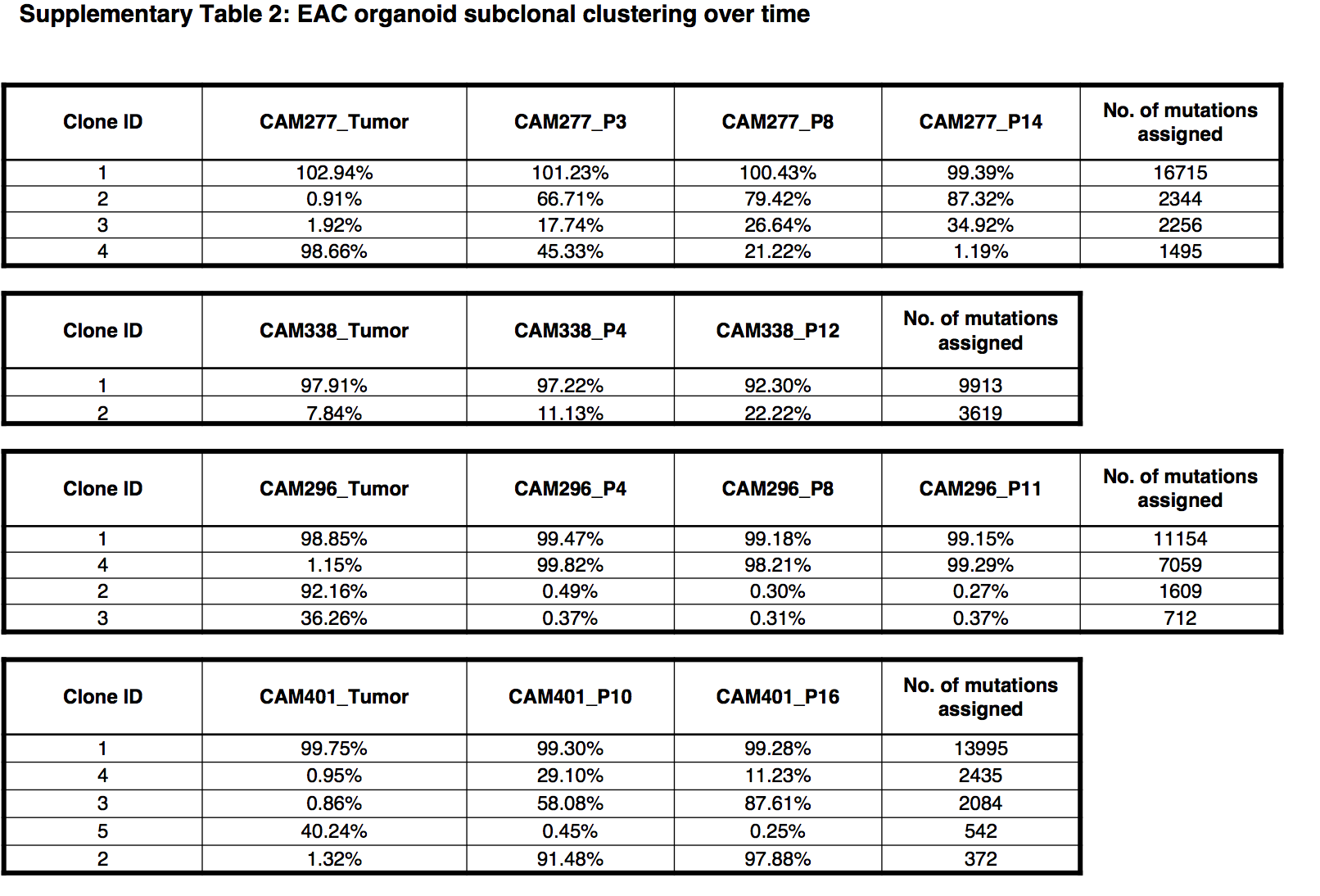
多时间点测序类器官看克隆进化
主图放2个病人,附件也放两个病人。
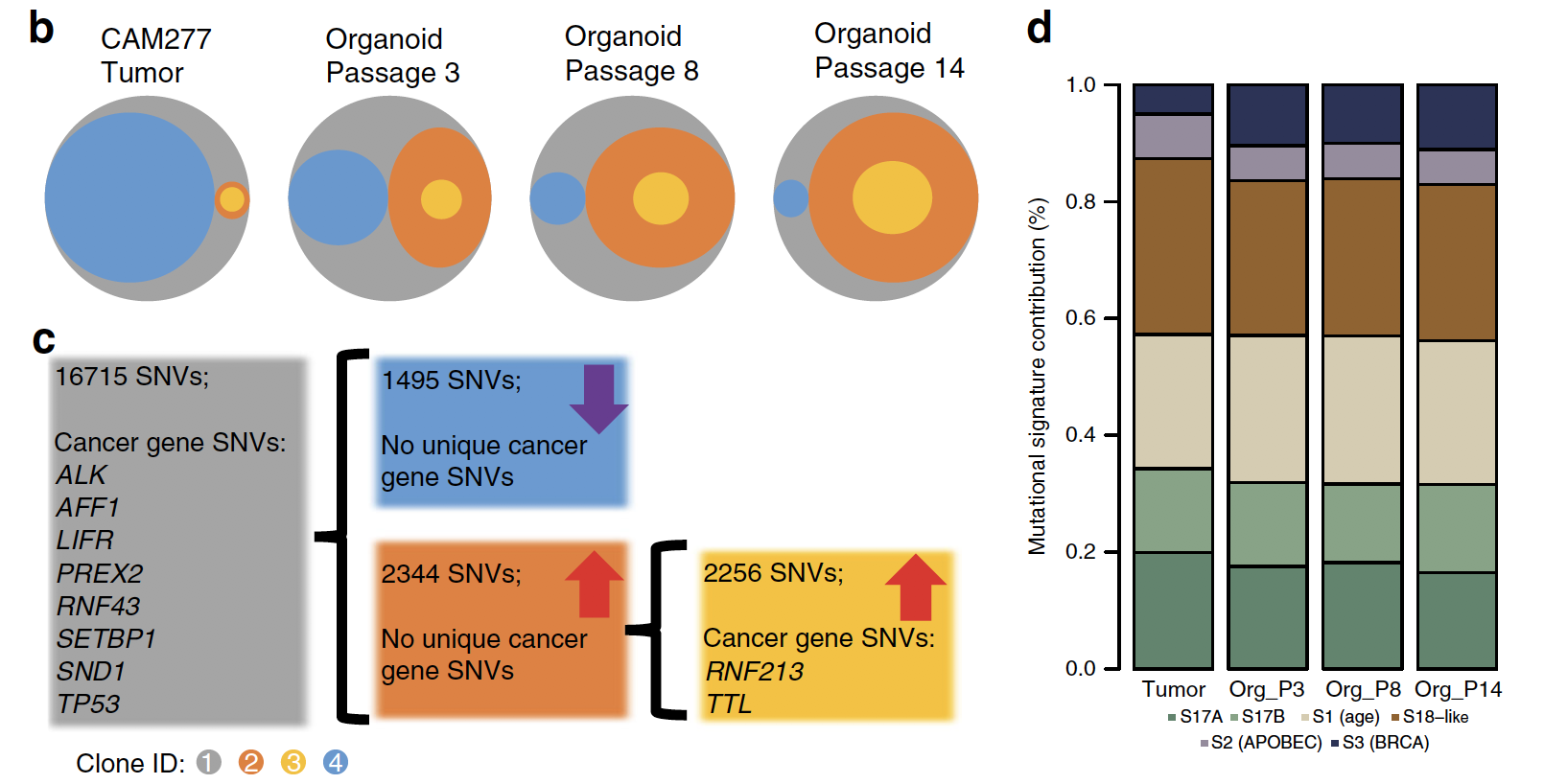
这个病人的类器官培养传代次数增加,其遗传特性也变化剧烈。

这个病人的类器官培养传代次数增加,其遗传特性很稳定。药物处理异质性
作者这里做了 Drug sensitivity profiling of 24 compounds against nine EAC organoids,然后把药物反应的IC50结果跟其基因组和转录组特性简单关联如下;
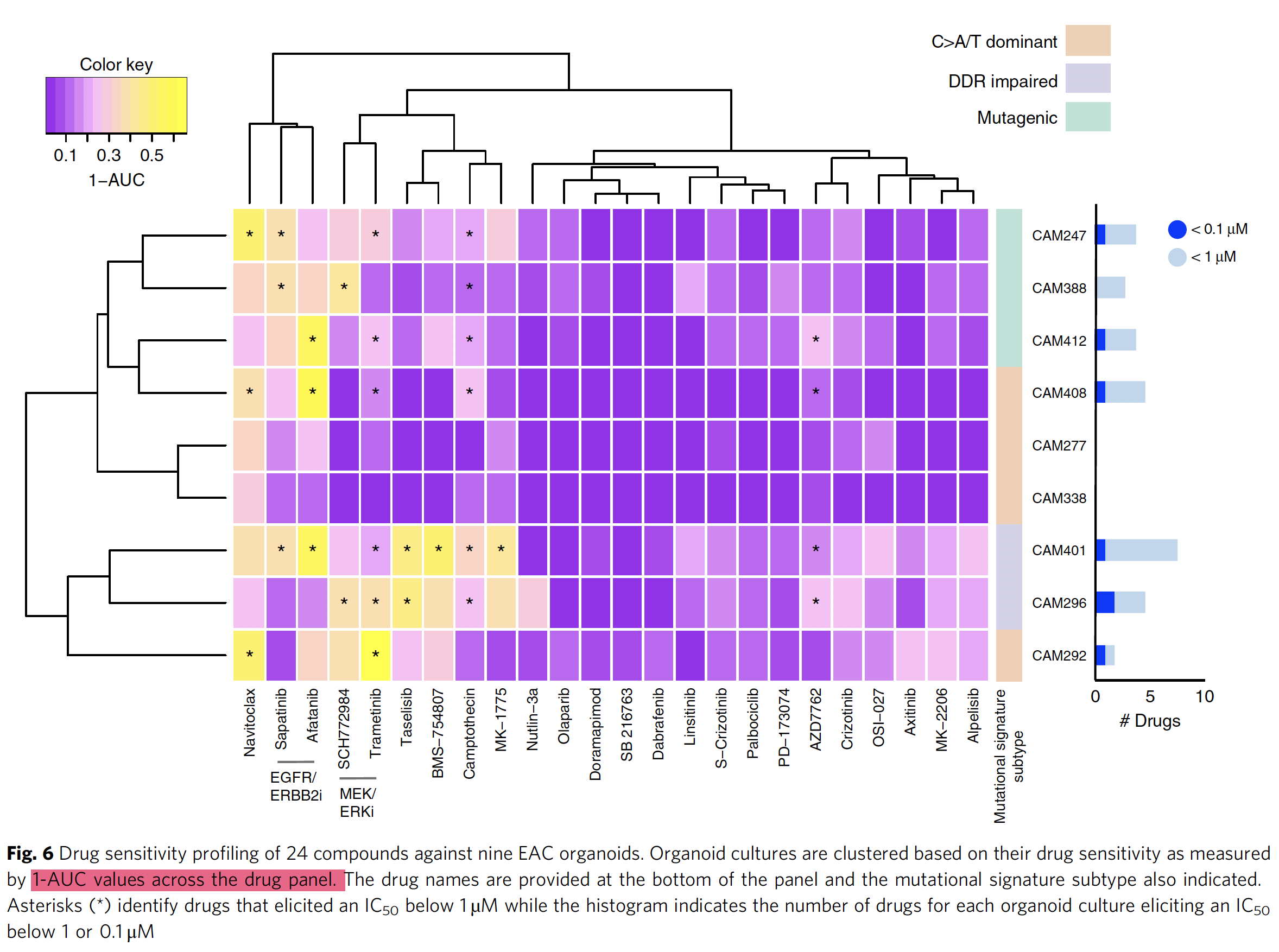
也比较好理解,作者试图说明某些organoids之所以对某些药物敏感,是因为它们有某种突变或者某些基因表达谱特性。