前面我们介绍了单细胞转录组表达矩阵可以推断CNV的文献出处及历史,也简单过了broad开发的inferCNV软件,在其提供的测试数据上面成功运行了,也测试了airway这个转录组数据,但是效果不好,现在看看CCLE数据库的测试结果吧,比较文章里面对之进行过同样的处理。
CCEL数据库介绍
需要简单注册后才能下载:https://portals.broadinstitute.org/ccle/users/sign_in 可以先看看CCLE里面收集的细胞系表型信息,大家可以自由统计。
options(stringsAsFactors = F)
a=read.table('data/CCLE_sample_info_file_2012-10-18.txt',sep = '\t',header = T)
as.data.frame(sort(table(a$Source),decreasing = T))
## Var1 Freq
## 1 ATCC 437
## 2 DSMZ 221
## 3 HSRRB 121
## 4 KCLB 66
## 5 RIKEN 66
## 6 ECACC 62
## 7 Academic Lab / Achilles 42
## 8 Academic Lab 12
## 9 ICLC 7
## 10 NCI/DCTD 7
## 11 5
准备输入数据
目前总共是1046个细胞系,注册并且下载数据导入R 的步骤我就不赘述了,如下;
load('CCLE_array_input.Rdata')
res[duplicated(res[,1]),1]
res[res[,1]=='TTL',1:6]
which(res[,1]=='TTL')
res=res[-14841,]
exprSet=res[,6:ncol(res)]
rownames(exprSet)=res[,1]
write.table(exprSet,'ccle_array_exprSet.txt',quote = F,sep = '\t')
pos = res[,1:4]
table(pos[,2])
write.table(pos,'ccle_array_pos.txt',row.names = F,col.names = F,sep = '\t',quote = F)
dim(exprSet)
dim(pos)
## 这个数据库res里面就包含了 所有细胞系的 芯片表达量,以及探针ID对应的基因及坐标。
# 可能是我前期数据预处理步骤失误,导致有一个基因留下来了两个探针,所以我需要删除掉其中一个。
还是跟前面一样把数据写出来,运行inferCNV这个软件。
> res[1:4,1:8]
gene chr start end Name LN18_CENTRAL_NERVOUS_SYSTEM
1 A1BG 19 58856544 58864865 1_at 6.965083
2 A1BG-AS1 19 58859117 58866549 503538_at 5.112451
3 A1CF 10 52559169 52645435 29974_at 3.576425
4 A2M 12 9220260 9268825 2_at 4.200074
> dim(exprSet)
[1] 16220 1037
> dim(pos)
[1] 16220 4
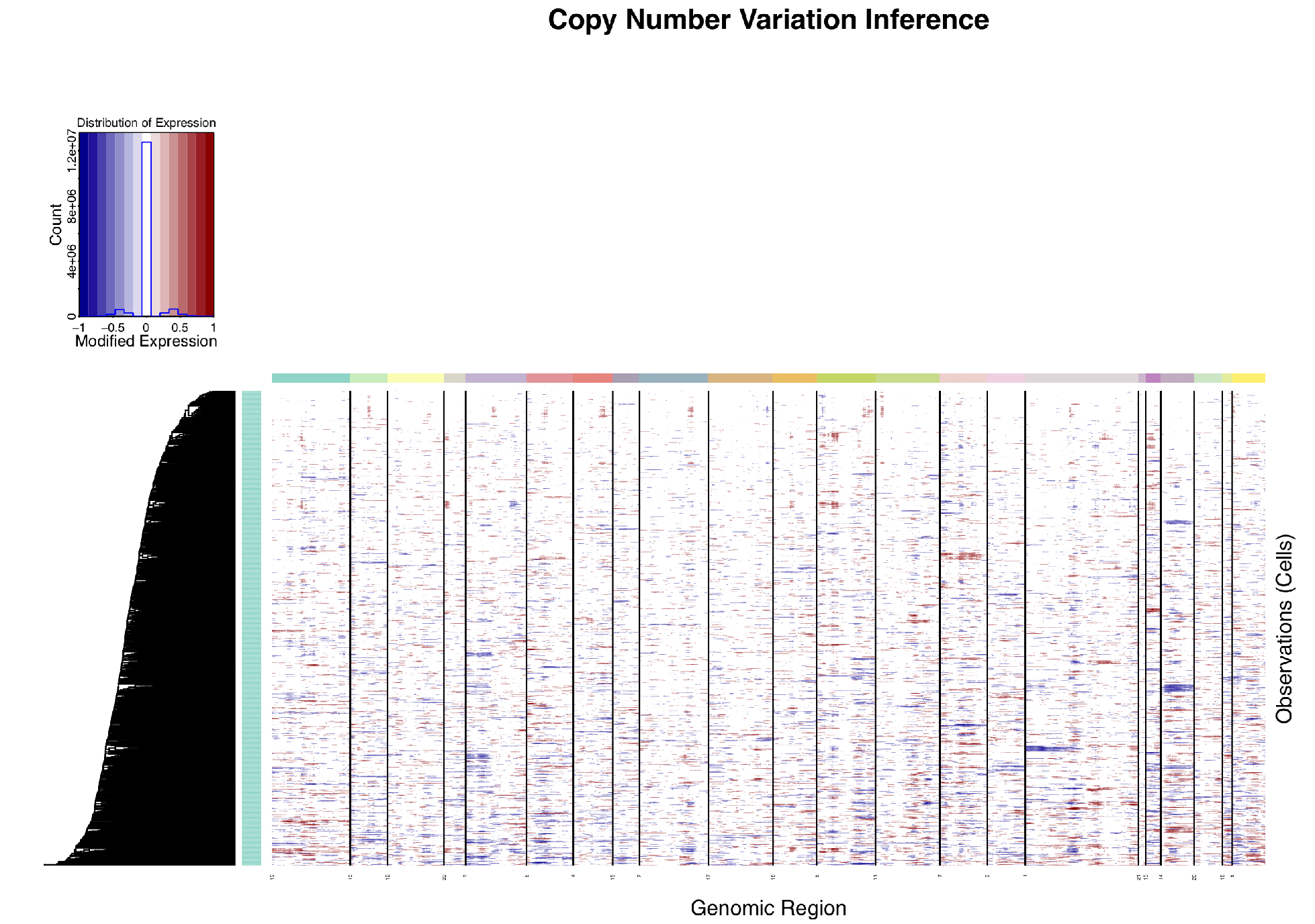
运行inferCNV软件
前面我们详细介绍了软件,这里直接给代码:
我的运行代码如下;
../inferCNV/scripts/inferCNV.R \
--cutoff 4.5 \
--noise_filter 0.3 \
--output_dir test \
--vis_bound_threshold " -1,1" \
ccle_array_exprSet.txt ccle_array_pos.txt
软件倒是可以成功运行,出来的图如下:
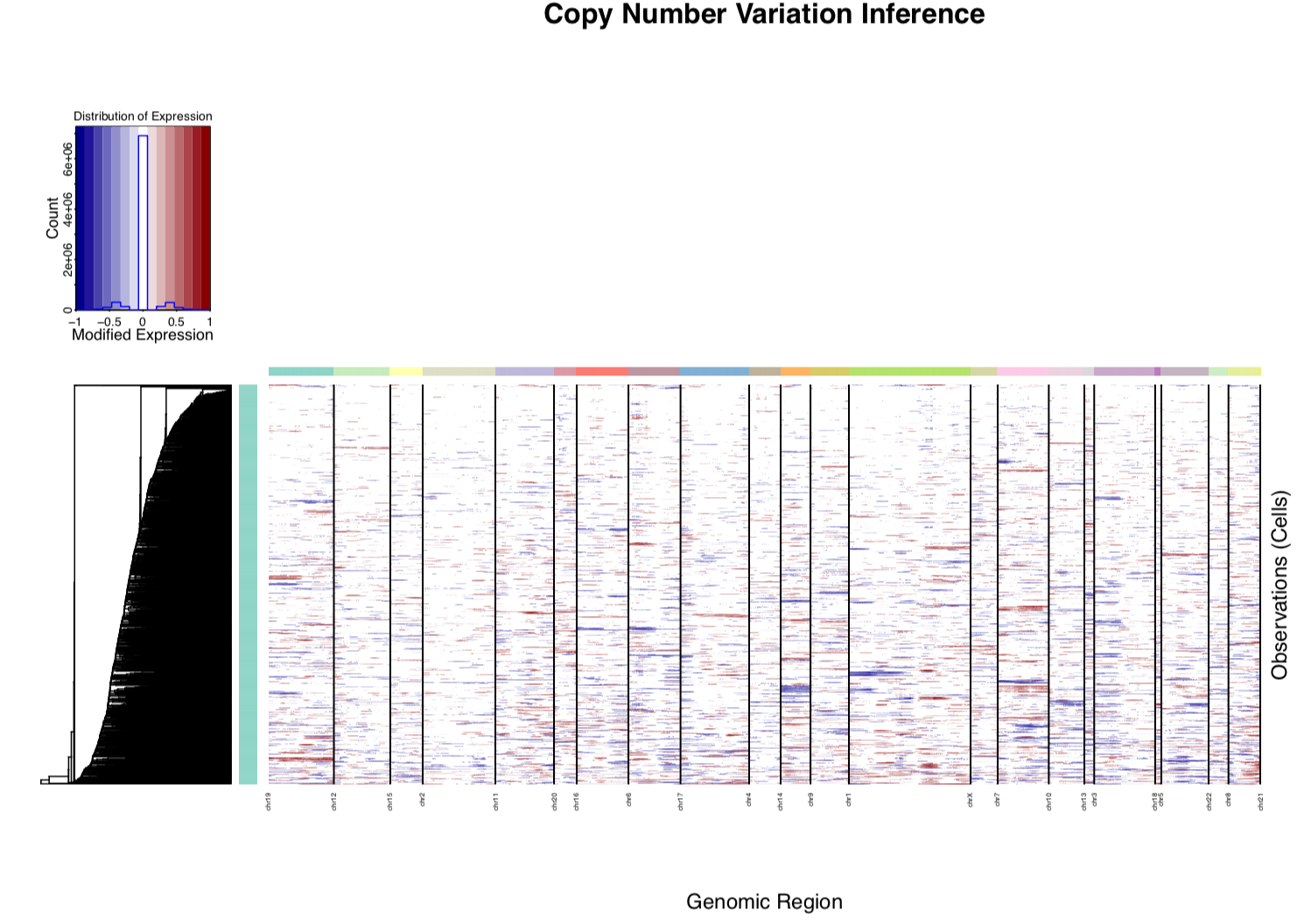
对CCLE的转录组数据同样处理
rm(list = ls())
load('CCLE_rnaseq_input.Rdata')
res[1:4,1:8]
res[duplicated(res[,1]),1]
res[res[,1]=='COG8',1:8]
res=res[!duplicated(res[,1]),]
exprSet=res[,7:ncol(res)]
rownames(exprSet)=res[,1]
library(edgeR)
exprSet=log(cpm(exprSet)+1)
write.table(exprSet,'ccle_rnaseq_exprSet.txt',quote = F,sep = '\t')
pos = res[,1:4]
table(pos[,2])
write.table(pos,'ccle_rnaseq_pos.txt',row.names = F,col.names = F,sep = '\t',quote = F)
dim(exprSet)
dim(pos)出图如下:
这里面的CCLE数据可以自行下载处理,也可以发邮件找我申请我处理好的,邮件发给 jmzeng1314@163.com
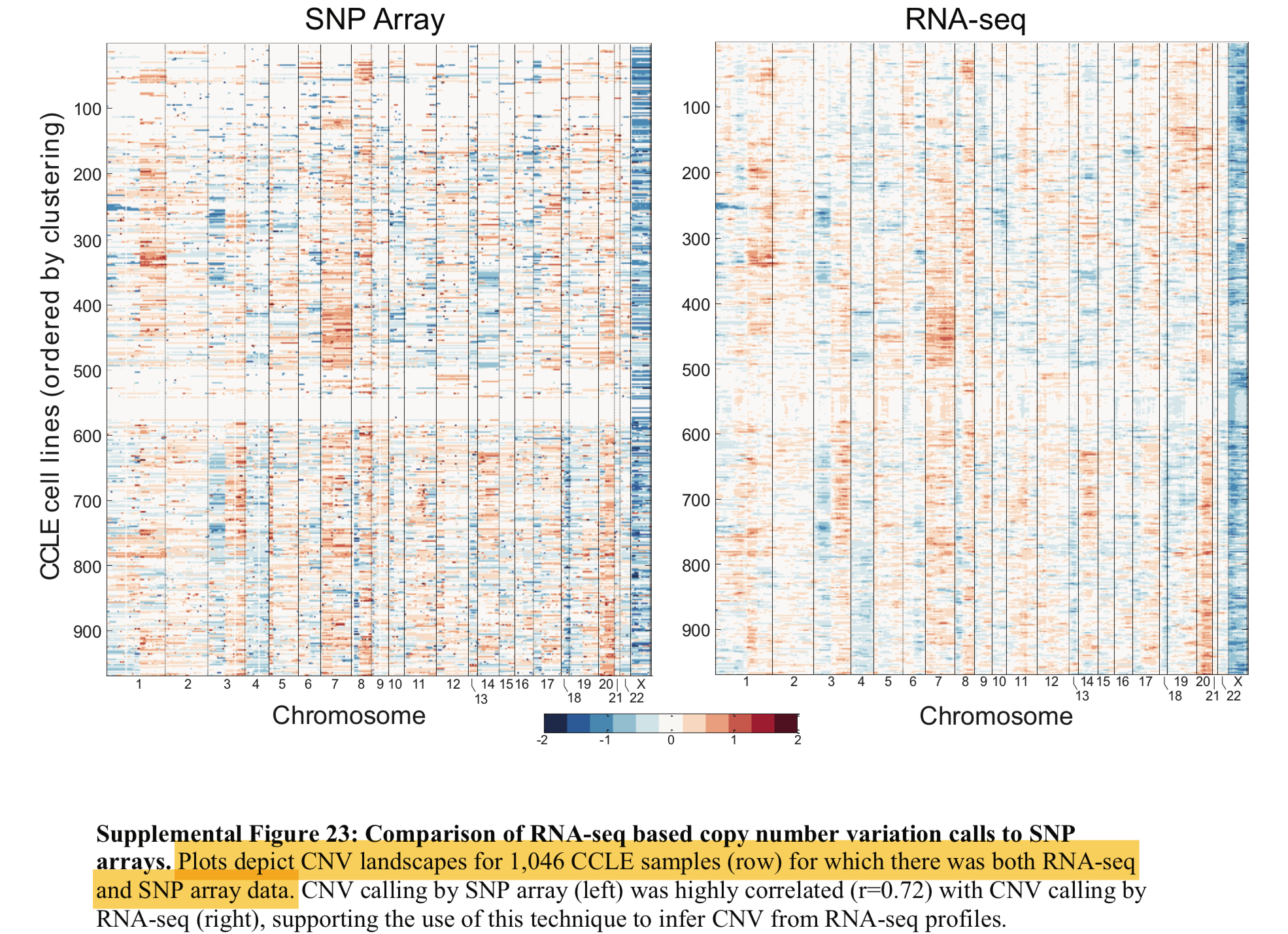
似乎是跟其2014年发在science上文章(PMID: 24925914)的补充材料最后一个图不相似:
[
一些其它关于CCLE数据库的资源
Cell line (1035个细胞系简介)Gene Sets,1035 sets of genes with high or low expression in each cell line relative to other cell lines from the CCLE Cell Line Gene Expression Profiles dataset.
http://amp.pharm.mssm.edu/Harmonizome/dataset/CCLE+Cell+Line+Gene+Expression+Profiles
一些关于CCLE数据库的文章:
http://cancerres.aacrjournals.org/content/73/8_Supplement/2409.short
http://cancerres.aacrjournals.org/content/74/22/6390.short
https://clincancerres.aacrjournals.org/content/19/19_Supplement/IA2.abstract
http://onlinelibrary.wiley.com/doi/10.1002/cncy.21471/pdf 介绍了几个类似的数据库资源
http://journals.plos.org/plosone/article?id=10.1371/journal.pone.0088557 讲解了high/low的知识
http://ieeexplore.ieee.org/xpls/abs_all.jsp?arnumber=7060697 药物相关
Anticancer drug sensitivity analysis: An integrated approach applied to Erlotinib sensitivity prediction in the CCLE database
http://biorxiv.org/content/biorxiv/early/2015/10/02/028159.full.pdf 比较了CCLE和TCGA的数据
we have developed a database “CancerDR”, which provides information of 148 anti-cancer drugs, and their pharmacological profiling across 952 cancer cell lines.