cel数据,处理成segment及genotype数据
一、程序安装
这本来是一个matlab程序,但是有linux版本,需要安装matlab编译环境
下载解压之后首先安装matlab环境:
./MCRInstaller.bin -console
因为我的服务器没有界面,所以我用了-console执行安装程序,很简单就安装好了
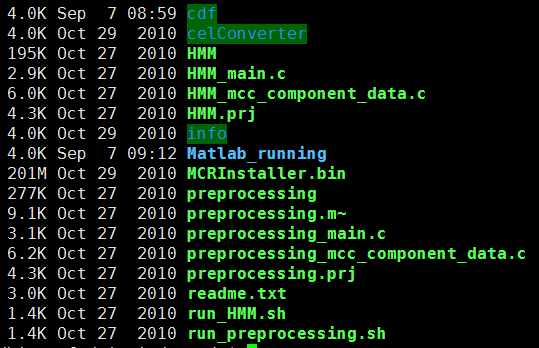
celConverter目录下面是一个java程序,可以把芯片出来的cel文件转为flat file
其中Matlab_running 是我执行安装的matlab环境
其余两个脚本run_HMM.sh run_preprocessing.sh是picnic程序的第二步和第三步
二、输入数据准备
我随便找了两个snp6.0芯片的raw data
-rw-rw-r-- 1 jmzeng jmzeng 66M Dec 30 06:30 GSM1949207.CEL
-rw-rw-r-- 1 jmzeng jmzeng 66M Sep 9 11:08 GSM887898.CEL
三、程序使用
程序主要分为三个步骤来实现
Step 1: Convert the binary *.cel file to a flat file
Step 2: Normalise and estimate the ploidy of the genome and the level of normal contamination
Step 3: Segment the data and produce the genotype
第一步:
暂时还不需要matlab工作环境
示例的code是:java -Xmx2G -jar CelFileConverter.jar -m Snp6FeatureMappings.csv -c 'cdf_file_including_path' -s 'directory name
of cel files' -t rootDir/outdir/raw
我的code是:
jmzeng@ubuntu:/home/jmzeng/bio-soft/picnic/c_code/celConverter$ java -jar CelFileConverter.jar -m Snp6FeatureMappings.csv -c ../cdf/GenomeWideSNP_6.Full.cdf -s ../celFiles/ -t ../result/
Loading CDF file: ../cdf/GenomeWideSNP_6.Full.cdf
Loading feature mapping file: Snp6FeatureMappings.csv
Processing CEL file: /home/jmzeng/bio-soft/picnic/c_code/celConverter/../celFiles/GSM1949207.CEL
Created feature intensity file: /home/jmzeng/bio-soft/picnic/c_code/celConverter/../result/GSM1949207.feature_intensity
Processing CEL file: /home/jmzeng/bio-soft/picnic/c_code/celConverter/../celFiles/GSM887898.CEL
Created feature intensity file: /home/jmzeng/bio-soft/picnic/c_code/celConverter/../result/GSM887898.feature_intensity
然后在输出目录就有了一个feature_intensity后缀的文本文件,约110M
-rw-rw-r-- 1 jmzeng jmzeng 105M Dec 30 06:35 GSM1949207.feature_intensity
-rw-rw-r-- 1 jmzeng jmzeng 109M Dec 30 06:35 GSM887898.feature_intensity
第二步:
这里需要用matlab啦,但是这里经常出现库的问题!!!
示例的code是
sh run_preprocessing.sh mcr_dir cell_name info_dir feature_int_dir normalised_outdir outdir sample_type in_pi
我的code是:
jmzeng@ubuntu:/home/jmzeng/bio-soft/picnic/c_code$ sh run_preprocessing.sh Matlab_running/v710/ GSM1949207 info/ result/ result/output result/
------------------------------------------
Setting up environment variables
---
LD_LIBRARY_PATH is .:Matlab_running/v710//runtime/glnxa64:Matlab_running/v710//bin/glnxa64:Matlab_running/v710//sys/os/glnxa64:Matlab_running/v710//sys/java/jre/glnxa64/jre/lib/amd64/native_threads:Matlab_running/v710//sys/java/jre/glnxa64/jre/lib/amd64/server:Matlab_running/v710//sys/java/jre/glnxa64/jre/lib/amd64/client:Matlab_running/v710//sys/java/jre/glnxa64/jre/lib/amd64
My Own Exception: Fatal error loading library /home/jmzeng/bio-soft/picnic/c_code/Matlab_running/v710/bin/glnxa64/libmwmclmcr.so
Error: libXp.so.6: cannot open shared object file: No such file or directory
第三步:
也需要用matlab,库的问题必须解决!!!
我另外一台服务器的结局办法是用v714的matlab
示例的code是
sh run_HMM.sh /nfs/team78pc2/kwl_temp/segments/PICNIC/C/release/Matlab_Compiler_Runtime/v710 A01_CGP_PD3945a.feature_intensity '/nfs/team78pc3/KWL/segments/PICNIC/matlab/C/release/info/' '/nfs/team78pc2/kwl_temp/segments/PICNIC/data/normalized/' '/nfs/team78pc2/kwl_temp/segments/PICNIC/data/' '10' '0.33598' '1.9915' '0.40997'
我的code是:无所谓了,这个服务器不知道怎么回事,总是出现库文件的问题,而这个问题需要root权限,我懒得弄了,我在其它的服务器上面都木有问题的!!!
所以我就换了MATLAB,毕竟,这个软件本来就是matlab版本的,第一步照旧,第二步在matlab里面运行!
我这里只用GSM1949207做测试吧!!!
Genomic DNA was extracted from saliva, peripheral blood, or fibroblast cell lines using the QIAamp DNA Blood Mini Kit or QIAamp DNA Mini Kit. DNA quality and quantity was assessed using a Nanodrop Spectrophotometer and agarose gel electrophoresis.
打开matlab,进入picnic目录
重复第二步,输入:
preprocessing('GSM1949207.feature_intensity','info\','result\raw\','result\output\','result\')
需要7个参数,我这个是cell lines数据,所以后面两个参数省略不写!info文件夹自己下载放在picnic目录,其中result\raw 存放你第一步的结果文件!
这一步运行,会比较久!
同时可以看到程序在我的result目录里面新增加了两个目录用来存放结果,不过这一步的结果还是中间文件,就不解释了!
然后再重复第三步,输入:
HMM('GSM1949207.feature_intensity','info\','result\output\','result\',10,0,2.0221,0.40997)
这个软件的参数很没规律,最好不要像说明书那些用output做文件夹名字,不然,很容易出错。
这一步好像也很耗时间!